Anwendung von Protein-Fragment-Komplementations-Assays in der Zellbiologie
- Einführung
- Prinzip
- Einschränkungen
- Standardkontrollen für eine PCA-Studie
- Anwendung von PCA im Proteindesign: Bibliothek vs. Bibliotheksscreening für optimal interagierende Proteine
- Anwendung von PCA auf cDNA-Bibliotheksscreening in Säugerzellen
- Verwendung von PCA als molekularem Lineal: Rezeptorstudien
- Mapping Biochemical Networks
- Schlussfolgerung
- Danksagungen
Einführung
Biochemische Wege sind wirklich Systeme der dynamischen Montage und Demontage von Proteinkomplexen, und so beschäftigt sich ein Großteil der modernen biologischen Forschung damit, wie, wann und wo Proteine mit anderen Proteinen interagieren, die an biochemischen Prozessen beteiligt sind. Die Nachfrage nach einfachen Ansätzen zur Untersuchung von Protein-Protein-Wechselwirkungen, insbesondere im großen Maßstab, ist in letzter Zeit mit dem Fortschritt in Genomprojekten gewachsen, da die Assoziation unbekannter mit bekannten Genprodukten einen entscheidenden Weg zur Etablierung der Funktion eines Gens darstellt. Mit Blick auf diese Herausforderung entwickelte unser Labor Protein-Fragment-Komplementierungstests (PCAs). Bei dieser Strategie werden zwei Proteine von Interesse (Proteine A und B) mit komplementären Fragmenten eines Reporterproteins (einem Enzym, fluoreszierendem Protein usw.) fusioniert.). Wenn die Proteine A und B interagieren, werden die Reporterfragmente zusammengebracht, falten sich in die native Struktur des Reporters und stellen seine Aktivität wieder her (Abbildung 1). PCA-Reporterproteine wurden als solche ausgewählt, die eine Vielzahl von nachweisbaren Aktivitäten erzeugen, einschließlich fluoreszierender, lumineszierender und kolorimetrischer Signale sowie einfacher Überlebensauswahltests (1-14). Wir haben gezeigt, dass die PCA-Strategie die folgenden Fähigkeiten aufweist: (i) Sie ermöglicht den Nachweis von Protein-Protein-Wechselwirkungen in vivo und in vitro in jedem Zelltyp; (ii) es ermöglicht den Nachweis von Protein-Protein-Wechselwirkungen in geeigneten subzellulären Kompartimenten oder Organellen; (iii) es ermöglicht den Nachweis von Wechselwirkungen, die spezifisch als Reaktion auf Entwicklungs-, Ernährungs-, Umwelt- oder hormoninduzierte Signale induziert werden; (iv) es ermöglicht die Überwachung kinetischer und Gleichgewichtsaspekte der Proteinassemblierung in Zellen; und (v) es ermöglicht das Screening auf neuartige Protein-Protein-Wechselwirkungen in jedem Zelltyp (2,3,6,9) (15-19).
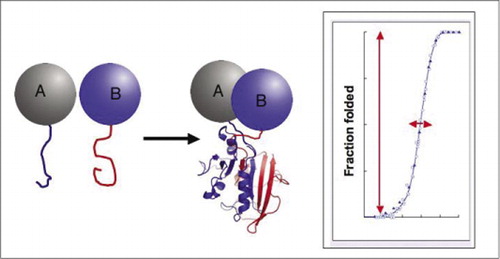
Wenn die beiden Proteine interagieren, werden die Reporterfragmente zusammengebracht, falten sich in die native Struktur des Reporterproteins und seine Aktivität wird rekonstituiert (links). Diese Proteinfragmentkomplementierungstests (PCAs) haben physikalische Eigenschaften, die sie besonders nützlich als Reporter dynamischer Proteinkomplexe machen. Auf der rechten Seite ist eine Proteinfaltungskurve, wobei die x-Achse ein variabler Parameter ist (z., Konzentration eines Fragments relativ zu einem anderen). Die hohe Kooperativität dieses Prozesses (extrem starker Anstieg des Anteils gefalteter Spezies über einen sehr engen Bereich) bedeutet, dass die Assays einen enormen dynamischen Bereich aufweisen, was den Nachweis eines Komplexes zu einem virtuellen All-or-None-Phänomen macht. Dies steht im Gegensatz zu Methoden wie dem Fluoreszenz-Resonanz-Energietransfer (FRET), der einen sehr geringen Dynamikbereich aufweist und eine sorgfältige Optimierung einer Reihe von Parametern erfordert. Im Gegensatz dazu ist die Messung der Proteinkomplexbildung durch PCA nicht schwieriger als die Messung der Aktivität des intakten Reporterenzyms.
Prinzip
Wir demonstrierten das Prinzip der PCA ausgehend vom Enzym Dihydrofolatreduktase (DHFR) als Reporter (1). Es war offensichtlich, dass, wenn die Faltung des Enzyms aus seinen Fragmenten (wie durch Rekonstitution der Aktivität nachgewiesen) absolut von der Bindung der wechselwirkenden Proteine abhängt, das beschriebene System tatsächlich ein Detektor der Wechselwirkungen ist. Wir und andere haben seitdem gezeigt, dass dieses Prinzip auf eine Reihe von Enzymen verallgemeinert werden kann, darunter Gaussia- und Renilla-Luciferasen, TEM-β-Lactamase sowie grün fluoreszierendes Protein (GFP) und seine Varianten (1-14). Ein entscheidendes Merkmal von PCA-Fragmenten ist, dass sie sich nicht spontan falten, ohne durch die Wechselwirkung der Proteine, mit denen sie fusioniert sind, in unmittelbare Nähe gebracht zu werden (1,20). Wenn eine spontane Faltung auftrat, würde PCA einfach nicht funktionieren. Spontanes Falten würde zu einem falsch positiven Signal führen, eine Situation, die die Interpretation von Bibliotheksbildschirmen in vivo hoffnungslos verwirren würde (voraussichtlich eine wichtige Anwendung). Im Gegensatz zu PCA gibt es Assaysysteme auf Basis von β-Galactosidase und gespaltenen Inteinen, die PCA ähneln, sich aber konzeptionell und praktisch unterscheiden (21,22). In beiden Fällen werden bekannte natürlich vorkommende und spontan assoziierende Untereinheiten der Enzyme zu interagierenden Proteinen fusioniert. Das zentrale Problem dabei ist, dass Untereinheiten, auch wenn sie schwach assoziiert sind, immer in der Lage sind, dies zu einem gewissen Grad zu tun, was bedeutet, dass es einen konstanten Hintergrund der spontanen Assemblierung gibt.
Einschränkungen
Die PCA-Strategie ist in dem Sinne allgemein, dass sie nicht auf einen einzelnen Enzymreporter beschränkt ist, und sie wurde in verschiedenen Formen entwickelt, von denen jede am besten geeignet ist, eine bestimmte Frage zu beantworten. Zum Beispiel sind einfache Überleben-Auswahl PCAs, wie die, die auf DHFR basieren, für Bibliotheksselektion am nützlichsten, während Lumineszenz- oder Fluoreszenzauslesung PCAs für Studien der räumlichen und zeitlichen Dynamik von Proteinkomplexen am besten sind. Da die Fusionsproteine in Zellen exprimiert werden können, die relevante Modelle für die Untersuchung eines spezifischen biochemischen Weges sind, befinden sie sich wahrscheinlich in ihrem nativen biologischen Zustand, einschließlich der korrekten posttranslationalen Modifikationen (offensichtlich dürfen die PCA-Fragmente selbst das Targeting oder die Modifikation der Proteine nicht stören, und dies muss getestet werden).
Zu den einfachsten und daher beliebtesten PCAs gehören solche, die auf fluoreszierenden Proteinen basieren (wie GFP und Varianten), da das Signal vom intrinsischen Fluorophor (7-9) bereitgestellt wird (14,15,17,23). Fluoreszierende Proteine müssen jedoch auf hohem Niveau exprimiert werden, um sicherzustellen, dass das Signal über der zellulären Hintergrundfluoreszenz liegt, und fluoreszierende Protein-PCAs haben sich als irreversibel erwiesen, was nützlich sein kann (Einfangen und Visualisieren seltener Komplexe), aber auch zu einer Fehlinterpretation des Umsatzes oder der Lokalisierung von wechselwirkenden Proteinen führen kann (8,23,24). Andererseits wurde nachgewiesen, dass PCAs, die auf DHFR und β-Lactamase als Reporter basieren, nach Unterbrechung der Wechselwirkungen indirekt reversibel sind, während ein PCA, das auf Gaussia-Luciferase basiert, direkt reversibel ist (2,3,6). Die Reversibilität von PCA ermöglicht somit den Nachweis von kinetischen und Gleichgewichtsaspekten der Proteinkomplexmontage und -demontage in lebenden Zellen (Abbildung 2).
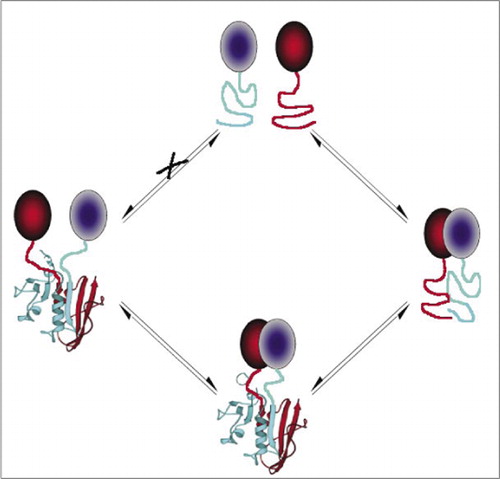
Dies verhindert eine spontane Assoziation der Fragmente (Weg X), die zu einem falschen Signal führen kann. Ebenso werden Fragmente ausgewählt, für die eine spontane Entfaltung von Fragmenten auftreten soll, wenn der Proteinkomplex gestört ist (linke Seite).
Standardkontrollen für eine PCA-Studie
Die PCA-Strategie erfordert, dass sich die Fragmente des Reporterproteins zusammensetzen und falten, nachdem die interessierenden Proteine einen Komplex gebildet haben. Der Zusammenbau und die korrekte Faltung des Reporters ist abhängig von der Wiederherstellung sowohl der strukturellen Geometrie, die den Reporterproteinen innewohnt, als auch des Komplexes, der durch die wechselwirkenden Proteine gebildet wird. Dies ist einer der Hauptunterschiede der PCA-Assays im Vergleich zu Fluoreszenz-Resonanz-Energie-Transfer (FRET) oder Biolumineszenz-Resonanz-Energie-Transfer (BRET) oder Hefe-Zwei-Hybrid-Assays, und diese Funktion ermöglichte es uns, eine strukturbasierte Studie des Erythropoietin-Rezeptors durchzuführen (19). Wir fügen typischerweise einen flexiblen Polypeptidlinker mit 10 Aminosäuren ein, der aus (Gly.Gly.Gly.Gly.Ser)2 zwischen dem interessierenden Protein und dem PCA-Reporterfragment (für beide Fusionen). Dieser Linker wurde gewählt, weil er so flexibel wie möglich ist, und wir haben empirisch beobachtet, dass Linker dieser Länge ausreichend lang sind, damit Fragmente sich finden und falten können, unabhängig von der Größe der wechselwirkenden Proteine, mit denen die Fragmente fusioniert sind (16).
Um sicherzustellen, dass keine unspezifischen Reaktionen auftreten, sollte eine Reihe von Kontrollen durchgeführt werden. Diese Kontrollen könnten Folgendes umfassen, obwohl die erste die wichtigste ist: (i) Nicht wechselwirkende Proteine. Eine PCA-Reaktion sollte nicht beobachtet werden, wenn nicht interagierende Proteine als PCA-Partner verwendet werden; auch sollte die Überexpression eines nicht wechselwirkenden Proteins allein nicht um die bekannte Wechselwirkung konkurrieren. (ii) Partnerprotein-Grenzflächenmutationen. Eine Punkt- oder Deletionsmutation eines Partners, von der bekannt ist, dass sie eine Interaktion stört, sollte ebenfalls eine PCA-Reaktion verhindern. iii) Wettbewerb. Eine PCA-Reaktion sollte durch die gleichzeitige Überexpression eines oder des anderen der interagierenden Proteine, das nicht mit einem komplementären PCA-Fragment fusioniert ist, vermindert werden. (iv) Fragmenttausch. Eine beobachtete Wechselwirkung zwischen zwei Proteinen sollte auch dann auftreten, wenn die Proteine mit den jeweiligen Reporterfragmenten getauscht werden.
Anwendung von PCA im Proteindesign: Bibliothek vs. Bibliotheksscreening für optimal interagierende Proteine
Zu den ersten Anwendungen eines PCA gehörte ein Proteindesignproblem. Der DHFR-PCA-Assay wurde in Escherichia coli verwendet, um zwei Bibliotheken komplementär gestalteter Leucin-Reißverschluss-bildender Sequenzen mit 1010 potentiell interagierenden Paaren zu screenen, von denen wir praktisch 106 abdecken konnten. Wir zeigten, dass das PCA sowohl hinsichtlich der optimalen Bindungsspezifität als auch der Löslichkeit und Expression interagierender Reißverschlüsse ausgewählt wurde (18,25). Das wichtigste Merkmal dieses Ansatzes ist, dass es möglich war, zwei Bibliotheken gleichzeitig gegeneinander zu screenen, ein Prozess, der mit vergleichbaren Hefe-Zwei-Hybrid-Screens nicht leicht zu erreichen ist. Die Einfachheit dieses Ansatzes und die Spezifität der erhaltenen Informationen über die Designstrategie deuten auf einen breiten Nutzen des DHFR-PCA im Proteindesign und in gerichteten Evolutionsexperimenten hin. Es zeigt auch, dass PCA die Phagen-Display-Strategien ergänzt, da die gesamten Auswahl-, Optimierungs- und Stringenztests in vivo durchgeführt werden, wodurch dieser Ansatz leicht ausgeführt werden kann.
Anwendung von PCA auf cDNA-Bibliotheksscreening in Säugerzellen
Ein erster Schritt zur Definition der Funktion eines neuartigen Genprodukts besteht darin, seine Wechselwirkungen mit anderen Genprodukten zu bestimmen. Ein rein proteininteraktionsbasierter Screening-Ansatz (z. B. Hefe-Zwei-Hybrid) ist jedoch begrenzt, da er nur angibt, dass zwei Proteine interagieren, während keine anderen Informationen bereitgestellt werden, die ein Protein mit seiner Funktion verknüpfen könnten. Daher haben wir gezeigt, dass PCA in einer cDNA-Bibliotheks-Screening-Strategie verwendet werden kann, die einen einfachen zellbasierten Protein-Interaktionsbildschirm mit spezifischen funktionellen Assays kombiniert, die eine erste Validierung der biologischen Relevanz der Interaktion ermöglichen (9). Der erste Schritt besteht aus dem Screening auf physikalische Wechselwirkungen zwischen dem Köder und einer Bibliothek cDNA-kodierter Beuteproteine, indem die Rekonstitution des PCA-Reporters in intakten lebenden Zellen überwacht wird. Ein wichtiges Merkmal dieses ersten Schrittes ist, dass Wechselwirkungen direkt und zwischen Proteinen voller Länge in Zellen nachgewiesen werden können, in denen das Köder-Protein normalerweise funktioniert, wodurch sichergestellt wird, dass notwendige subzelluläre Targeting, posttranslationale Modifikationen und Wechselwirkungen mit anderen Proteinen auftreten können. Für die experimentelle Validität muss offensichtlich nachgewiesen werden, dass die PCA-Fragmente das Targeting oder die Modifikation der Proteine nicht stören. Im zweiten Schritt kann die Proteininteraktion wie folgt funktionell validiert werden: erstens muss die von PCA nachgewiesene Proteininteraktion durch Wirkstoffe wie Hormone oder spezifische Inhibitoren gestört werden, von denen bekannt ist, dass sie den spezifischen biochemischen Weg modulieren, an dem die Proteine beteiligt sind. Wir haben dies für die DHFR-PCA demonstriert und diese Eigenschaft genutzt, um Signalwege in lebenden Säugetierzellen abzubilden (16). Zweitens könnte die subzelluläre Lokalisation der Proteininteraktion, die wiederum von PCA nachgewiesen wird, durch Mittel verändert werden, die den Signalweg modulieren. Somit kombiniert die PCA-basierte Screening-Strategie einen einfachen Screening-Schritt mit direkten funktionellen Assays. Wir und andere haben diese Strategie auf die Identifizierung neuer Substrate oder Regulatoren der Serin / Threonin-Proteinkinase PKB / Akt (9,15,26,27) angewendet.
Verwendung von PCA als molekularem Lineal: Rezeptorstudien
Eine Besonderheit von PCA-Strategien besteht darin, dass, wenn wir die dreidimensionale Struktur des Reporterenzyms kennen, genau vorhergesagt werden kann, wie nahe die Fragmente beieinander liegen müssen, um sicherzustellen, dass sich das Enzym korrekt faltet und eine messbare Aktivität aufweist. Diese Tatsache wurde genutzt, um ein strukturelles allosterisches Modell zur Aktivierung des dimeren Erythropoietinrezeptors (EpoR) unter Verwendung des DHFR-PCA zu testen, und der Ansatz konnte auf die Untersuchung allosterischer Übergänge in dimeren oder multimeren Proteingrenzflächen ausgeweitet werden (19). Im EpoR-Fall wurde gezeigt, dass die Transmembrandomänen des Rezeptordimers durch 73 Å getrennt sind, wie in der Kristallstruktur von unligiertem EpoR beobachtet. Es wurde argumentiert, dass, wenn dieser inaktive Zustand auf der Membran einer lebenden Zelle existierte, DHFR-Fragmente, die mit den C-Termini der Transmembrandomänen fusioniert waren, sich nur falten würden, wenn ein Ligand eine Konformationsänderung induzierte, die es den Fragmenten ermöglichte, nahe genug zusammenzukommen, um sicherzustellen, dass die genaue dreidimensionale Struktur von DHFR gebildet werden konnte (19,28). Dies würde erfordern, dass die N Endpunkte der Fragmente 8 Å voneinander entfernt sind. Die Insertion flexibler Linkerpeptide zwischen der Transmembrandomäne und DHFR-Fragmenten ermöglichte es uns, den Abstand zwischen den Insertionspunkten des extrazellulären Domänendimers zu untersuchen und zu bestätigen, dass Linker, die lang genug sind, um 73 Å zu überspannen, benötigt wurden, damit sich DHFR aus seinen Fragmenten faltet.
Mapping Biochemical Networks
Zelluläre biochemische Maschinen für Stoffwechsel, Signalkaskaden und Zellzyklus sind Beispiele für die dynamische Montage und Demontage makromolekularer Komplexe. Diese werden definiert, indem interagierende Proteine nach ihren ähnlichen Reaktionen auf eine Reihe von Störungen (Hormone, Metaboliten, Enzyminhibitoren usw.) gruppiert werden.). Protein-Protein-Wechselwirkungen können verwendet werden, um ein Protein unbekannter Funktion mit Proteinen zu verknüpfen, von denen bekannt ist, dass sie an einem bekannten biochemischen Prozess beteiligt sind. Wir haben gezeigt, dass pharmakologisches Profiling (Überwachung der Auswirkungen von signalwegspezifischen Arzneimitteln und Proteinhormonen auf Protein-Protein-Wechselwirkungen) und die Bestimmung des zellulären Ortes von Protein-Protein-Wechselwirkungen mit PCAs erreicht werden können (9)(15-17)(26). Die Analyse dieser Ergebnisse ermöglicht eine Darstellung, wie sich biochemische Netzwerke in Zeit und Raum und als Reaktion auf bestimmte Reize entwickeln. Als Proof of Principle berichteten wir über die Anwendung dieser Strategie auf die Kartierung eines Signaltransduktionsweges, der durch Rezeptor-Tyrosinkinasen (RTKs) vermittelt wird (16). Die pharmakologischen Profile und die zelluläre Lage der Wechselwirkungen, die wir beobachteten, ermöglichten es uns, jedes Genprodukt an seinem relevanten Punkt in den Signalwegen zu platzieren (Abbildung 3). Aus den Ergebnissen unserer Analyse ergab sich eine Karte der Organisation des RTK-Netzwerks, die mit bestehenden Modellen übereinstimmte, aber auch mehrere neuartige Interaktionen enthielt. Die Fähigkeit, ein Netzwerk von Proteininteraktionen in lebenden Zellen zu überwachen, das alle Komponenten des untersuchten zugrunde liegenden Signalwegs enthält, ergab versteckte Verbindungen, die zuvor trotz intensiver Untersuchung dieses Netzwerks nicht beobachtet wurden. Die vorgestellten Ergebnisse zeigen, dass die PCA-Strategie die Merkmale aufweist, die für eine allgemeine Genfunktionsvalidierungs- und Pathway-Mapping-Strategie erforderlich sind. Eine kürzlich durchgeführte Anwendung eines größeren Satzes von PCAs ermöglichte die Entwicklung eines allgemeinen Ansatzes, um die Wirkungen von Arzneimitteln auf bestimmte Signalwege zu verknüpfen und unvorhergesehene Aktivitäten von Arzneimitteln zu erkennen (17).
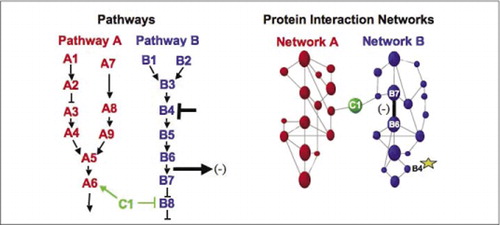
(Links) Die Wirkung eines hemmenden Störmittels, das auf Protein B4 wirkt (T-Bar), wird stromabwärts durch eine Änderung der Wechselwirkung der Proteine B6 und B7 miteinander nachgewiesen (Pfeil). In diesem Fall ist der Effekt der Störung eine Abnahme der Anzahl der wechselwirkenden Proteine (-), wie sie von einem Reporter dieser Wechselwirkung detektiert werden (Ausgangssignal der Wechselwirkung, wie es beispielsweise von PCA Sentinel detektiert wird). Der Effekt könnte jedoch auch positiv sein, abhängig von den Folgen der Hemmung des Upstream-Proteins. (Rechts) Innerhalb des Protein-Interaktionsnetzwerks für Weg B breitet sich eine Störung des Proteins B4 (Stern) irgendwie durch das Netzwerk aus, um irgendwie die Verbindung (breiter Balken) zwischen den Proteinen B6 und B7 zu beeinflussen. Dies bedeutet nicht, dass Protein B4 physikalisch mit B6 oder B7 interagiert; Die Ausbreitung eines Affekts durch das Proteininteraktionsnetzwerk kann auf direkte physikalische Verbindungen oder auf enzymatische Prozesse zurückzuführen sein, die im Netzwerk nicht offensichtlich sind.
Schlussfolgerung
Die Entwicklung und Anwendung von PCA sind noch im Gange. Zum Beispiel, zusätzlich zu den begrenzten, obwohl informativ, Sätze von Anwendungen hier beschrieben, Die Strategie wird auf groß angelegte Screening von ganzen Genomen angewendet. Komplexere Probleme des Proteindesigns und der Proteinfaltung werden untersucht, einschließlich Studien der Faktoren, die die Auswahl von Sequenzen für optimale Wechselwirkungen zwischen Proteinen, Proteinen und Nukleinsäuren sowie Proteinen und kleinen organischen Molekülen steuern. PCA ist ein sehr allgemeiner und flexibler experimenteller Ansatz, und daher sollten wir in naher Zukunft eine wachsende Anzahl neuer Anwendungen dieses grundlegenden Werkzeugs in der Molekular- und Zellbiologie erwarten.
Danksagungen
Stephen Michnick ist Inhaber des Canada Research Chair in Integrative Genomics. Forschungsergebnisse aus unserem Labor wurden von den Canadian Institutes of Health Research finanziert.
- 1. Pelletier, J.N., F.X. Campbell-Valois und S.W. Michnick. 1998. Oligomerisierung domänengerichtete Reassemblierung der aktiven Dihydrofolatreduktase aus rational gestalteten Fragmenten. Prok. Natl. Acad. Sci. USA 95: 12141-12146.Crossref, Medline, CAS, Google Scholar
- 2. Remy, I. und S.W. Michnick. 1999. Klonale Selektion und in vivo Quantifizierung von Proteininteraktionen mit Proteinfragmentkomplementierungstests. Prok. Natl. Acad. Sci. USA 96: 5394-5399.Crossref, Medline, CAS, Google Scholar
- 3. Galarneau, A., M. Primeau, L.E. Trudeau und S.W. Michnick. 2002. Beta-Lactamase-Proteinfragmentkomplementierungstests als In vivo- und In vitro-Sensoren für Protein-Protein-Interaktionen. Nat. In: Biotechnol. 20:619–622.Crossref, Medline, CAS, Google Scholar
- 4. Wehrman, T., B. Kleaveland, J.H. Her, R.F. Balint und H.M. Blau. 2002. Protein-Protein-Interaktionen in Säugetierzellen durch Komplementierung von Beta-Lactamase-Enzymfragmenten überwacht. Prok. Natl. Acad. Sci. USA 99: 3469-3474.Crossref, Medline, CAS, Google Scholar
- 5. Spotts, J.M., R.E. Dolmetsch und M.E. Greenberg. 2002. Zeitraffer-Bildgebung einer dynamischen phosphorylierungsabhängigen Protein-Protein-Interaktion in Säugetierzellen. Prok. Natl. Acad. Sci. USA 99: 15142-15147.Crossref, Medline, CAS, Google Scholar
- 6. Remy, I. und S.W. Michnick. 2006. Ein hochempfindlicher Protein-Protein-Interaktionstest basierend auf Gaussia Luciferase. Nat. Methoden 3: 977-979.Crossref, Medline, CAS, Google Scholar
- 7. Ghosh, I., A.D. Hamilton und L. Regan. 2000. Antiparallel Leucin Zipper-directed Protein reassembly: Anwendung auf das grün fluoreszierende Protein. J. Am. Chem. Soc. 122:5658–5659.Crossref, CAS, Google Scholar
- 8. Hu, C.D., Y. Chinenov und T.K. Kerppola. 2002. Visualisierung der Wechselwirkungen zwischen Proteinen der bZIP- und Rel-Familie in lebenden Zellen mittels bimolekularer Fluoreszenzkomplementierung. Mol. Zelle 9: 789-798.Crossref, Medline, CAS, Google Scholar
- 9. Remy, I. und S.W. Michnick. 2004. Eine funktionelle Screening-Strategie für cDNA-Bibliotheken, die auf fluoreszierenden Proteinkomplementations-Assays basiert, um neuartige Komponenten von Signalwegen zu identifizieren. Methoden 32:381-388.Crossref, Medline, CAS, Google Scholar
- 10. Remy, I., F.X. Campbell-Valois, G. Ghaddar, S. Aquin und S.W. Michnick. 2005. Nachweis von Proteininteraktionen und Bibliotheksscreening mit Protein-Fragment-Komplementations-Assays, S. 637-672. In Protein-Protein-Interaktionen: Ein Handbuch zum molekularen Klonen, 2nd ed. CSH Laboratory Press, Cold Spring Harbor, NY.Google Scholar
- 11. Paulmurugan, R. und S.S. Gambhir. 2003. Überwachung von Protein-Protein-Interaktionen mit gespaltener synthetischer Renilla-Luciferase-Protein-Fragment-assistierter Komplementierung. Anal. Chem. 75:1584–1589.Crossref, Medline, CAS, Google Scholar
- 12. Paulmurugan, R., Y. Umezawa und S.S. Gambhir. 2002. Nichtinvasive Bildgebung von Protein-Protein-Interaktionen bei lebenden Probanden unter Verwendung von Reporterproteinkomplementierungs- und Rekonstitutionsstrategien. Prok. Natl. Acad. Sci. USA 99: 15608-15613.Crossref, Medline, CAS, Google Scholar
- 13. Luker, K.E., M.C. Smith, G.D. Luker, S.T. Gammon, H. Piwnica-Worms und D. Piwnica-Worms. 2004. Kinetik der regulierten Protein-Protein-Wechselwirkungen mit Firefly Luciferase Komplementation Imaging in Zellen und lebenden Tieren aufgedeckt. Prok. Natl. Acad. Sci. USA 101: 12288-12293.Crossref, Medline, CAS, Google Scholar
- 14. Jach, G., M. Pesch, K. Richter, S. Frings, und J.F. Uhrig. 2006. Ein verbessertes mRFP1 fügt der bimolekularen Fluoreszenzkomplementierung Rot hinzu. Nat. Methoden 3: 597-600.Crossref, Medline, CAS, Google Scholar
- 15. Remy, I., A. Montmarquette und S.W. Michnick. 2004. PKB/ Akt moduliert die TGF-Beta-Signalisierung durch direkte Interaktion mit Smad3. Nat. In: Cell Biol. 6:358–365.Crossref, Medline, CAS, Google Scholar
- 16. Remy, I. und S.W. Michnick. 2001. Visualisierung biochemischer Netzwerke in lebenden Zellen. Prok. Natl. Acad. Sci. USA 98: 7678-7683.Crossref, Medline, CAS, Google Scholar
- 17. Die Ergebnisse der Studie werden von der University of New York, J. Lamerdin, S. Owens, B.H. Keon, G.K. Bilter, Z. Shang, Z. Huang, H. Yu, et al.. 2006. Identifizierung von Off-Target-Effekten und versteckten Phänotypen von Medikamenten in menschlichen Zellen. Nat. Chem. Biol. 2:329–337.Crossref, Medline, CAS, Google Scholar
- 18. Pelletier, J.N., K.M. Arndt, A. Pluckthun und S. W. Michnick. 1999. Eine in vivo Library-versus-Library-Auswahl optimierter Protein-Protein-Interaktionen. Nat. In: Biotechnol. 17:683–690.Crossref, Medline, CAS, Google Scholar
- 19. Remy, I., I.A. Wilson und S.W. Michnick. 1999. Aktivierung des Erythropoietinrezeptors durch eine ligandeninduzierte Konformationsänderung. Wissenschaft 283: 990-993.Crossref, Medline, CAS, Google Scholar
- 20. Gegg, C.V., K.E. Bowers und C.R. Matthews. 1997. Untersuchung minimaler unabhängiger Falteinheiten in Dihydrofolatreduktase durch molekulare Dissektion. Protein Sci. 6:1885–1892.Crossref, Medline, CAS, Google Scholar
- 21. Rossi, F., C.A. Charlton und H.M. Blau. 1997. Überwachung der Protein-Protein-Interaktionen in intakten eukaryotischen Zellen durch Beta-Galactosidase-Komplementierung. Prok. Natl. Acad. Sci. USA 94: 8405-8410.Crossref, Medline, CAS, Google Scholar
- 22. Ozawa, T., S. Nogami, M. Sato, Y. Ohya und Y. Umezawa. 2000. Fluoreszenzindikator zum Nachweis von Protein-Protein-Wechselwirkungen in vivo basierend auf Proteinspleißen. Anal. Chem. 72:5151–5157.Crossref, Medline, CAS, Google Scholar
- 23. Magliery, T.J., C.G. Wilson, W. Pan, D. Mishler, I. Ghosh, A. D. Hamilton und L. Regan. 2005. Nachweis von Protein-Protein-Wechselwirkungen mit einer grün fluoreszierenden Proteinfragment-Reassemblierungsfalle: Umfang und Mechanismus. J. Am. Chem. Soc. 127:146–157.Crossref, Medline, CAS, Google Scholar
- 24. Nyfeler, B., S.W. Michnick und H.P. Hauri. 2005. Erfassung von Proteininteraktionen im sekretorischen Weg lebender Zellen. Prok. Natl. Acad. Sci. USA 102: 6350-6355.Crossref, Medline, CAS, Google Scholar
- 25. Arndt, K.M., J.N. Pelletier, K.M. Müller, T. Alber, S. W. Michnick und A. Pluckthun. 2000. Ein heterodimeres Coiled-Coil-Peptidpaar, das in vivo aus einem entworfenen Library-versus-Library-Ensemble ausgewählt wurde. In: J. Mol. Biol. 295:627–639.Crossref, Medline, CAS, Google Scholar
- 26. Remy, I. und S.W. Michnick. 2004. Regulation der Apoptose durch das Ft1-Protein, einen neuen Modulator der Proteinkinase B / Akt. Mol. Zelle. Biol. 24:1493–1504.Crossref, Medline, CAS, Google Scholar
- 27. Ding, Z., J. Liang, Y. Lu, Q. Yu, Z. Songyang, S.Y. Lin und G.B. Mills. 2006. Ein Retrovirus-basierter Proteinkomplementations-Assay-Bildschirm zeigt funktionelle AKT1-Bindungspartner. Prok. Natl. Acad. Sci. USA 103:15014–15019.Crossref, Medline, CAS, Google Scholar
- 28. Cody, V., J.R. Luft, E. Ciszak, T.I. Kalman, and J.H. Freisheim. 1992. Crystal structure determination at 2.3 A of recombinant human dihydrofolate reductase ternary complex with NADPH and methotrexate-gamma-tetrazole. Anticancer Drug Des. 7:483–491.Medline, CAS, Google Scholar