L’Institut de recherche sur la création
 L’un des arguments les plus populaires utilisés pour les humains supposés évoluer à partir de singes est connu sous le nom de fusion des chromosomes. L’impulsion de ce concept est le problème évolutif selon lequel les singes ont une paire supplémentaire de chromosomes — les humains en ont 46 tandis que les singes en ont 48. Si les humains ont évolué à partir d’une créature ressemblant à un singe il y a seulement trois à six millions d’années, un simple coup dans le grand schéma de l’histoire de l’évolution, pourquoi les humains et les singes ont-ils cette divergence?
L’un des arguments les plus populaires utilisés pour les humains supposés évoluer à partir de singes est connu sous le nom de fusion des chromosomes. L’impulsion de ce concept est le problème évolutif selon lequel les singes ont une paire supplémentaire de chromosomes — les humains en ont 46 tandis que les singes en ont 48. Si les humains ont évolué à partir d’une créature ressemblant à un singe il y a seulement trois à six millions d’années, un simple coup dans le grand schéma de l’histoire de l’évolution, pourquoi les humains et les singes ont-ils cette divergence?
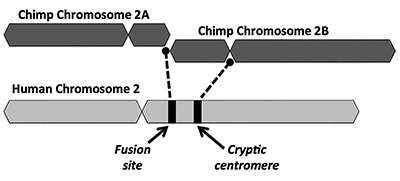
La solution évolutive propose qu’une fusion de bout en bout de deux petits chromosomes ressemblant à des singes (nommés 2A et 2B) a produit le chromosome 2 humain (Figure 1). Le concept de fusion est apparu pour la première fois en 1982 lorsque les scientifiques ont examiné les similitudes des chromosomes humains et des singes au microscope. Alors que la technique était un peu grossière, c’était suffisant pour faire avancer l’idée.1
Le Site Dit De Fusion
La première signature ADN réelle d’un événement de fusion possible a été découverte en 1991 sur le chromosome 2.2 humain. Les chercheurs ont trouvé un petit groupe confus de séquences terminales ressemblant à des télomères qui ressemblaient vaguement à une fusion possible. Les télomères sont une séquence à six bases des lettres d’ADN TTAGGG répétées encore et encore aux extrémités des chromosomes.
Cependant, la signature de fusion était en quelque sorte une énigme basée sur les fusions réelles qui se produisent occasionnellement dans la nature. Toutes les fusions documentées chez les animaux vivants impliquent un type spécifique de séquence appelé ADN satellite (ADNAT) situé dans les chromosomes et trouvé dans les ruptures et les fusions.3-5 La signature de fusion sur le chromosome 2 humain manquait à cet ADNc révélateur.6
Un autre problème est la petite taille du site de fusion, qui ne mesure que 798 lettres d’ADN. Les séquences de télomères aux extrémités des chromosomes ont une longueur de 5 000 à 15 000 bases. Si deux chromosomes avaient fusionné, vous devriez voir une signature télomère fusionnée de 10 000 à 30 000 bases de long — pas 798.
Non seulement la petite taille est un problème pour l’histoire de fusion, mais la signature ne représente pas vraiment une fusion claire des télomères. La figure 2 montre les lettres d’ADN du site de fusion à 798 bases avec les séquences de télomères intactes à six bases (lettre d’ADN) soulignées en caractères gras. Lorsque la séquence de fusion est comparée à celle d’une signature de fusion vierge de même taille, elle n’est globalement identique qu’à 70%.

Des chercheurs laïques ont souligné cet écart et ont qualifié le site de fusion de “dégénéré” de manière significative.” 7 Compte tenu du modèle théorique standard de l’évolution humaine, il devrait être identique à environ 98 à 99%, et non à 70%. Les chercheurs décrivant cette découverte ont commenté: “Les réseaux de répétitions en tête-à-tête sur le site de fusion ont dégénéré de manière significative (14%) à partir des réseaux presque parfaits de (TTAGGG) n trouvés aux télomères” et ont posé la question pertinente “Si la fusion s’est produite dans les réseaux de répétitions télomères inférieurs à ~ 6 Mya, pourquoi les réseaux sur le site de fusion dégénèrent-ils si?”7 Il convient de noter que la dégénérescence de 14% citée par les auteurs se réfère à la corruption de seulement les séquences à six bases elles-mêmes, et non de l’ensemble des 798 bases.
Le Site de fusion À L’intérieur d’un Gène ?
La découverte anti-évolutive la plus remarquable sur le site de fusion s’est avérée être son emplacement et ce qu’il fait réellement. Cette découverte est survenue alors que je lisais le document de recherche qui rapportait une analyse détaillée de 614 000 bases de séquences d’ADN entourant le site de fusion présumé. J’ai remarqué dans l’une des figures que le site de fusion était situé à l’intérieur d’un gène, et de manière assez remarquable, cette bizarrerie n’était même pas reconnue dans le texte de l’article.8
Une telle découverte est très remarquable. Peut-être que cette information aurait été le clou dans le cercueil évolutif, pour ainsi dire, c’est pourquoi les chercheurs ont refusé d’en discuter. Cette anomalie majeure m’a incité à examiner de plus près le site de fusion. Cet article a été publié en 2002 et j’en ai pris connaissance en 2013. Une énorme quantité de données sur la structure et la fonction du génome humain avaient été publiées entre-temps, et il y avait probablement beaucoup plus à découvrir.
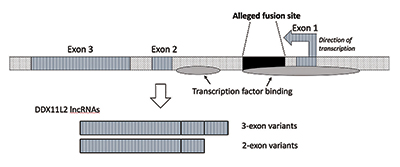
Lorsque j’ai effectué d’autres recherches, j’ai vérifié que le site de fusion était positionné à l’intérieur d’un gène de l’ARN hélicase maintenant appelé DDX11L2. La plupart des gènes chez les plantes et les animaux ont leurs segments codants en morceaux appelés exons afin qu’ils puissent être alternativement épissés. Sur la base de l’ajout ou de l’exclusion d’exons, les gènes peuvent produire une variété de produits. Les régions intermédiaires entre les exons sont appelées introns, qui contiennent souvent une variété de signaux et de commutateurs qui contrôlent la fonction génique. Le site de fusion présumé est positionné à l’intérieur du premier intron du gène DDX11L2 (Figure 3).9
La molécule d’ADN est double brin, avec un brin plus et un brin moins. Il a été conçu de cette façon pour maximiser la densité d’informations tout en augmentant l’efficacité et la fonction. En conséquence, il y a des gènes qui courent dans des directions différentes sur les brins opposés. Il s’avère que le gène DDX11L2 est codé sur le brin moins. Parce que les gènes chez l’homme sont comme des couteaux suisses et peuvent produire une variété d’ARN, dans le cas du gène DDX11L2, il produit des variants courts composés de deux exons et des variants longs avec trois (Figure 3).9
Le Site de Fusion Est un Promoteur du Gène
Que pourrait faire ce gène DDX11L2? Mes recherches ont montré qu’il est exprimé dans au moins 255 types de cellules ou de tissus différents.9 Il est également co-exprimé (activé en même temps) avec une variété d’autres gènes et est connecté aux processus associés à la signalisation cellulaire dans la matrice extracellulaire et à la production de cellules sanguines. L’emplacement de la séquence dite de fusion à l’intérieur d’un gène fonctionnel associé à la génétique d’une variété de processus cellulaires réfute fortement l’idée que c’est le sous-produit accidentel d’une fusion télomérique tête-à-tête. Les gènes ne sont pas formés par des fusions chromosomiques catastrophiques!
Encore plus étonnant est que le site de fusion est lui-même fonctionnel et remplit un objectif technique important. Le site agit en fait comme un commutateur pour contrôler l’activité des gènes. À cet égard, une multitude de données biochimiques ont montré que 12 protéines différentes appelées facteurs de transcription régulent ce segment du gène. L’une d’elles n’est autre que l’ARN polymérase II, l’enzyme principale qui copie les molécules d’ARN de l’ADN dans un processus appelé transcription. Cette découverte est étayée par le fait que le processus réel de transcription s’initie à l’intérieur de la région du site dit de fusion.
Techniquement, nous appellerions l’activité dans le site de fusion présumé une région promotrice. Les promoteurs sont les principaux commutateurs au début des gènes qui les activent et sont également l’endroit où l’ARN polymérase commence à créer un ARN. De nombreux gènes ont des promoteurs alternatifs comme le gène DDX11L2.
Il existe en fait deux zones de liaison au facteur de transcription dans le gène DDX11L2. Le premier est dans le promoteur directement devant le premier exon, et le second est dans le premier intron correspondent à la séquence de site de fusion. Non seulement le gène DDX11L2 est lui-même contrôlé de manière complexe, la séquence de fusion présumée jouant un rôle clé, mais même les transcrits d’ARN produits sont très complexes. Les ARN eux-mêmes contiennent une grande variété de sites de liaison et de contrôle pour une classe de petites molécules régulatrices appelées microARN.9
Les séquences de Télomères internes fonctionnelles Sont partout Dans le Génome
La présence de séquences de télomères situées à l’intérieur se retrouve partout dans le génome humain. Ces répétitions de télomères apparemment déplacées ont été surnommées télomères interstitiels. La présence de ces séquences présente un autre défi pour l’idée de site de fusion. C’est un fait que très peu de répétitions de télomères dans le site de fusion se produisent en tandem. Comme indiqué à la figure 2, la séquence du site de fusion à 798 bases ne contient que quelques cas où deux répétitions sont en fait en tandem et aucun n’a trois répétitions ou plus. Cependant, il existe de nombreux autres sites de télomères interstitiels dans tout le génome humain où les répétitions se produisent en tandem parfait trois à dix fois ou plus.10-11
Même en plus de leur rôle aux extrémités des chromosomes, il semble que les répétitions télomères interstitielles puissent remplir une fonction importante dans le génome liée à l’expression des gènes. Dans un projet de recherche récent, j’ai identifié des répétitions de télomères dans tout le génome humain, puis j’ai recoupé leurs emplacements génomiques avec une diversité d’ensembles de données contenant des informations biochimiques fonctionnelles pour l’activité des gènes.12 Littéralement, des milliers de répétitions télomères internes à travers le génome ont été directement associées aux caractéristiques de l’expression génique. Le même type de liaison aux facteurs de transcription et d’activité génique se produisant au site de fusion présumé se produisait également à l’échelle du génome à de nombreuses autres répétitions télomériques interstitielles. De toute évidence, ces caractéristiques de l’ADN ne sont pas des accidents de l’évolution, mais un code fonctionnel conçu de manière délibérée et intelligente.
Faux Centromère cryptique À l’intérieur d’un gène
Un autre problème clé avec le modèle de fusion est le manque de preuves viables d’une signature d’une région de centromère supplémentaire. Les centromères sont des sections de chromosomes, souvent situées à des endroits centraux, qui jouent un rôle clé lors de la division cellulaire. Comme le montre la figure 1, le chromosome chimérique nouvellement formé aurait eu deux sites centromères immédiatement après la prétendue fusion tête à tête des deux chromosomes. Dans un tel cas, l’un des centromères serait fonctionnel tandis que l’autre serait désactivé. La présence de deux centromères actifs est une mauvaise nouvelle pour les chromosomes et entraînerait un dysfonctionnement et une destruction cellulaire.
Fait intéressant, la preuve d’un centromère cryptique (désactivé) sur le chromosome 2 humain est encore plus faible que celle d’un site de fusion riche en télomères. Les évolutionnistes expliquent l’absence d’un centromère secondaire non fonctionnel clairement distinguable en faisant valoir qu’un deuxième centromère aurait été rapidement sélectionné contre. Après cela, le centromère handicapé se serait détérioré avec le temps car il n’y avait plus de contraintes fonctionnelles en faisant quelque chose d’utile dans le génome.
Cependant, la preuve d’un second centromère rémanent à n’importe quel stade de la dégénérescence de séquence est problématique pour le paradigme évolutif. Les séquences de centromères fonctionnels sont composées d’un type répétitif d’ADN appelé séquences alphoïdes, chaque répétition alphoïde ayant une longueur d’environ 171 bases. Certains types de répétitions alphoïdes se retrouvent partout dans le génome, tandis que d’autres sont spécifiques aux centromères. La structure des séquences trouvées sur le site cryptique du centromère sur le chromosome 2 humain ne correspond pas à celles associées aux centromères humains fonctionnels.13 Pire encore pour le modèle évolutif, c’est qu’ils n’ont pas d’homologues très similaires dans le génome du chimpanzé — ils sont spécifiques à l’homme.13
Le prétendu centromère fossile est également exceptionnellement petit par rapport à un vrai. La taille d’un centromère humain normal varie en longueur entre 250 000 et 5 000 000 bases.14 Le prétendu centromère cryptique ne mesure que 41 608 bases, mais il est également important de noter qu’il en existe trois régions différentes qui ne sont même pas des répétitions alphoïdes.15 Deux d’entre elles sont appelées rétroéléments, l’une étant une répétition LPA3/LIGNE longue de 5 957 bases et l’autre un élément SVA-E avec 2 571 bases. Lorsque nous soustrayons les insertions de ces séquences non alphoïdes, cela donne une longueur de seulement 33 080 bases, ce qui est une fraction de la longueur d’un centromère réel.
Le problème évolutif le plus grave avec l’idée d’un centromère fossile, cependant, est que, comme le site de fusion présumé, il est positionné à l’intérieur d’un gène. Le prétendu centromère cryptique est situé à l’intérieur du gène ANKRD30BL, et sa séquence couvre à la fois les régions intron et exon du gène.12,15
En fait, la partie de la séquence présumée du centromère fossile qui atterrit à l’intérieur d’un exon code en fait pour les acides aminés dans la protéine du gène résultant. Le gène produit une protéine qui est censée être impliquée dans l’interaction du réseau structurel de protéines à l’intérieur de la cellule appelé cytosquelette en relation avec des protéines réceptrices intégrées dans la membrane cellulaire.16 Le fait que le soi-disant centromère fossile ou cryptique soit une région fonctionnelle à l’intérieur d’un gène codant une protéine importante réfute complètement l’idée qu’il s’agisse d’un centromère disparu.
Conclusion: Aucune fusion
En raison des signatures confuses et des petites tailles des sites de fusion et de centromères fossiles présumés, il est très discutable que leur séquence ait été dérivée évolutivement d’une fusion chromosomique ancienne. Non seulement cela, ils représentent une séquence fonctionnelle à l’intérieur des gènes. Le site de fusion présumé est un commutateur génétique important appelé promoteur à l’intérieur du gène de l’ARN non codant long DDX11L2, et le soi-disant centromère fossile contient à la fois une séquence codante et une séquence non codante à l’intérieur d’un grand gène codant les protéines à répétition de l’ankyrine.
C’est un double coup de massue indéniable contre toute l’idée mythique de fusion, détruisant totalement sa validité. La conclusion scientifique accablante est que la fusion n’a jamais eu lieu.
- Yunis, J. J. et O. Prakash. 1982. L’origine de l’homme: un héritage pictural chromosomique. Sciences. 215 (4539): 1525-1530.
- Ijdo, J. W. et coll. 1991. Origine du chromosome 2 humain : Une fusion télomère-télomère ancestrale. Actes de l’Académie nationale des Sciences. 88 (20): 9051-9055.
- Chaves, R. et al. 2003. Analyse cytogénétique moléculaire et organisation satellite centromérique d’une nouvelle translocation 8;11 chez le mouton: un intermédiaire possible dans l’évolution des chromosomes biarmés. Génome des mammifères. 14 (10): 706-710.
- Tsipouri, V. et al. 2008. Des analyses de séquences comparatives révèlent des sites de fusions chromosomiques ancestrales dans le génome indien de muntjac. Biologie du génome. 9 (10): R155.
- Adega, F., H. Guedes-Pinto et R. Chaves. 2009. ADN satellite dans l’évolution du Caryotype des Animaux Domestiques – Considérations cliniques. Recherche cytogénétique et génomique. 126 (1-2): 12-20.
- Tomkins, J. P. et J. Bergman. 2011. Télomères: implications pour le vieillissement et preuves pour la conception intelligente. Journal de la Création. 25 (1): 86-97.
- Fan, Y. et al. 2002. Genomic Structure and Evolution of the Ancestral Chromosome Fusion Site in 2q13-2q14.1 and Paralogous Regions on Other Human Chromosomes. Recherche sur le génome. 12 (11): 1651-1662.
- Fan, Y. et al. 2002. Gene Content and Function of the Ancestral Chromosome Fusion Site in Human Chromosome 2q13-2q14.1 and Paralogous Regions. Recherche sur le génome. 12 (11): 1663-1672.
- Tomkins, J. P. 2013. Le “Site de Fusion” Présumé du Chromosome 2 Humain Code un Domaine Actif de Liaison à l’ADN À L’Intérieur d’une Fusion Complexe et Hautement Exprimée De Négation de Gènes. Journal de recherche sur les réponses. 6: 367-375.
- Azzaline, C. M., S. G. Nergadze et E. Giulotto. 2001. Répétitions télomériques intrachromosomiques humaines: organisation des séquences et mécanismes d’origine. Chromosome. 110: 75-82.
- Ruiz-Herrera, A. et al. 2008. Répétitions télomères loin des fins: mécanismes d’origine et rôle dans l’évolution. Recherche cytogénétique et génomique. 122 (3-4): 219-228.
- Tomkins, J. P. 2018. Les données génomiques combinatoires réfutent la fusion évolutive du chromosome 2 humain et construisent un modèle de conception fonctionnelle pour les répétitions télomériques interstitielles. Dans les actes de la Huitième Conférence internationale sur le créationnisme. J. H. Whitmore, éd. Pittsburgh, PA: Bourse de recherche en sciences de la création, 222-228.
- Tomkins, J. et J. Bergman. 2011. Le modèle de fusion du chromosome 2 de l’évolution humaine — partie 2: réanalyse des données génomiques. Journal de la Création. 25 (2): 111-117.
- Aldrup-Macdonald, M. E. et B. A. Sullivan. 2014. Le Passé, le Présent et l’avenir de la Génomique des Centromères Humains. Genes (Bâle). 5 (1): 33-50.
- Tomkins, J. P. 2017. Démystifier les Démystificateurs: Une Réponse aux Critiques et à l’Obscurcissement Concernant la Réfutation de la Fusion du Chromosome 2 Humain. Journal de recherche sur les réponses. 10: 45-54.
- Voronin, D. A. et E. V. Kiseleva. 2008. Rôle Fonctionnel des Protéines Contenant des Répétitions d’Ankyrine. Biologie cellulaire et tissulaire. 49 (12): 989-999.
* Le Dr Tomkins est directeur des sciences de la vie à l’Institute for Creation Research et a obtenu son doctorat en génétique de l’Université de Clemson.