Qu’est-ce que la PCR des colonies ?
La PCR des colonies est une méthode de PCR rapide et à haut débit permettant de déterminer la présence ou l’absence de l’ADN inséré dans le plasmide directement à partir des colonies bactériennes.
Le clonage moléculaire est l’une des méthodes de transformation de l’ADN les plus populaires depuis longtemps. Cependant, pour déterminer la présence ou l’absence de l’insert d’ADN, nous devons effectuer des expériences de transformation.
La PCR des colonies est une nouvelle méthode dans laquelle, en concevant les amorces spécifiques à l’ADN inséré, nous pouvons identifier si notre ADN d’intérêt est inséré dans le plasmide ou non.
Cependant, ce n’est pas aussi simple que nous en discutons.
Dans cet article, nous nous concentrerons sur la PCR de colonie en particulier, le principe de la PCR de Colonie, ses avantages et ses limites.
Pour cela, nous devons comprendre plusieurs termes et sujets. Nous allons commencer notre sujet à partir des bases. Le contenu de l’article est,
Thèmes Clés:
” Un plasmide est l’ADN circulaire bactérien qui se réplique indépendamment du chromosome bactérien et utilisé dans la manipulation et le transfert de gènes.”
Le clonage génétique est un outil de génétique moléculaire traditionnel utilisé depuis longtemps dans les laboratoires. En bref, dans le clonage de gènes, le gène de notre intérêt est inséré dans le plasmide par des moyens artificiels. Cet ADN est répliqué indépendamment du chromosome bactérien.
Les plasmides sont en fait utilisés pour générer de nombreuses copies de segments courts d’ADN. Parce que les bactéries se répliquent plus rapidement que tout autre organisme, nous pouvons générer de nombreuses copies du gène de notre intérêt en l’insérant dans le plasmide bactérien.
Le plasmide F, le plasmide Col, le plasmide dégradant et le plasmide de résistance sont plusieurs types courants de plasmides présents dans les bactéries.
De plus, le plasmide peut fonctionner comme un support moléculaire qui transfère de courts segments d’ADN d’une cellule à une autre cellule.
Nous avons couvert un article étonnant et approfondi sur l’ADN plasmidique. Lisez-le ici: ADN Plasmidique – Structure, Fonction, Isolement Et Applications.
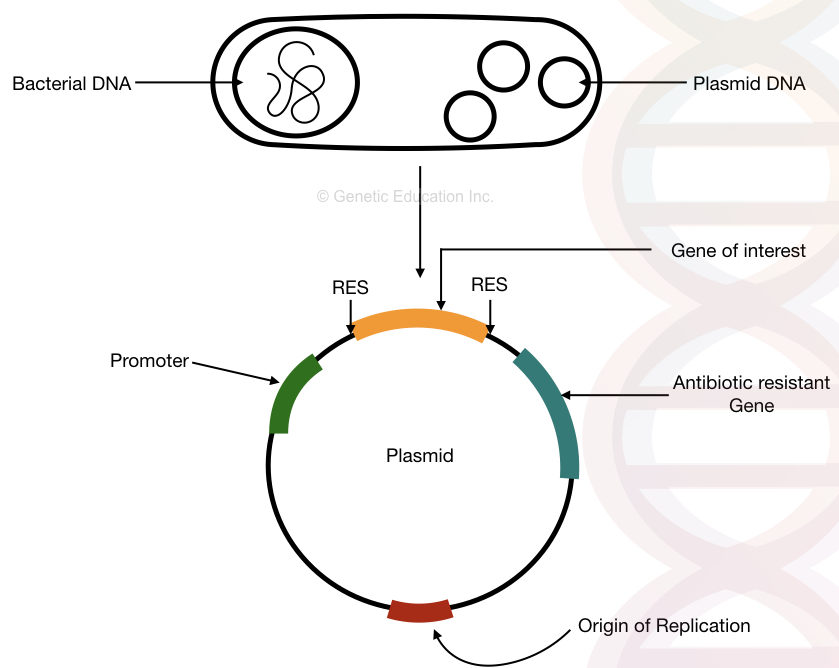
La structure de l’ADN plasmidique bactérien avec, l’origine de la réplication, le gène de résistance aux antibiotiques, le promoteur et le gène d’intérêt.
Outre les bactéries, plusieurs autres procaryotes contiennent également de l’ADN plasmidique. La fonction principale du plasmide chez les bactéries est leur survie dans des conditions difficiles.
Comme le plasmide transfère le gène de notre intérêt, il est très important de déterminer si notre gène d’intérêt est inséré ou non dans le plasmide.
Pour cela, nous pouvons utiliser plusieurs méthodes telles que la PCR et la culture microbienne.
Plaquer les colonies prend plus de temps et la sensibilité de la méthode n’est pas non plus bonne. Le risque de contamination est toujours élevé dans les méthodes de culture bactérienne.
Les résultats ne sont donc pas exacts.
Notre PCR aide ici aussi. En utilisant la méthode PCR des colonies, un insert d’ADN peut être déterminé ou identifié.
- Comment mettre en place un laboratoire d’extraction d’ADN: Un guide complet (produits chimiques, instruments et autres utilitaires).
- Délétion du chromosome 6p: Une raison pour ne pas souffrir, ne pas avoir faim et ne pas dormir
Qu’est-ce que la PCR des colonies?
La PCR des colonies est la modification de la PCR conventionnelle dans laquelle les colonies bactériennes sont directement utilisées comme matrice de PCR.
L’ADN plasmidique qui contient l’ADN de notre intérêt est amplifié dans les conditions dépendantes de la température cyclique.
La représentation graphique de la PCR de la colonie est illustrée dans la figure ci-dessous,
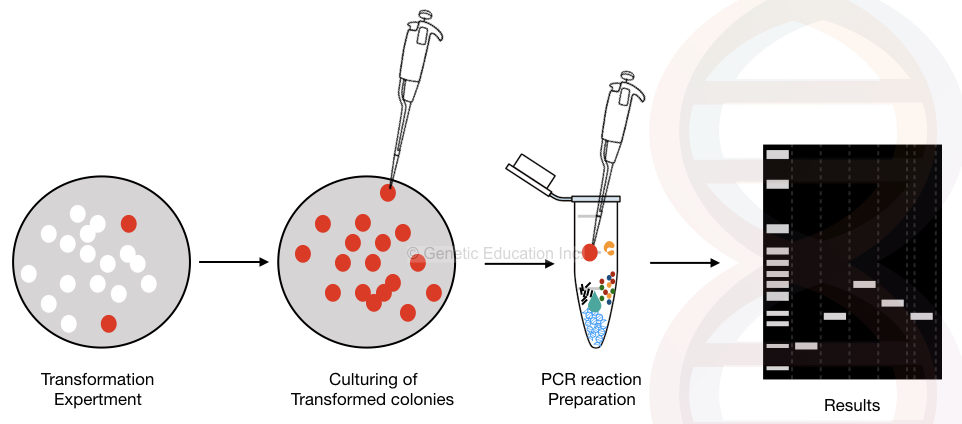
Aperçu général de la méthode de PCR des colonies.
Principe de la PCR des colonies:
La colonie bactérienne contenant le plasmide peut être directement amplifiée à l’aide de deux jeux d’amorces. Les amorces spécifiques à l’insert qui amplifient la séquence d’insertion et les amorces flanquantes spécifiques au vecteur, qui amplifient l’ADN plasmidique autre que l’ADN inséré (régions flanquantes des deux côtés de l’insert).
En utilisant les amorces de flanquement de l’insert (qui amplifient le reste de l’ADN), la taille de notre insert d’ADN peut être déterminée.
Une colonie bactérienne est prélevée et ajoutée directement dans le mélange-maître contenant tous les réactifs de PCR. De plus, en ajoutant une étape initiale de chauffage à la PCR, l’ADN plasmidique sort de la cellule bactérienne et est amplifié dans la réaction.
C’est le principe de base de la PCR des colonies, Cependant, elle peut être modifiée en fonction des exigences.
Le protocole de PCR de colonie :
La PCR de colonie est l’une des excellentes modifications de la PCR conventionnelle. Au lieu de l’ADN modèle, les colonies bactériennes sont directement ajoutées à la réaction. En outre, l’ADN polymérase Taq, les amorces, le tampon de réaction PCR et le DD / W sont également ajoutés dans la réaction PCR.
Ici, dans la PCR de la colonie, la sélection des amorces est très importante. De plus, la sélection des amorces dépend de l’objectif de notre expérience.
Quel type d’information voulons-nous de notre expérience de PCR de colonie?
-
-
- Informations sur la présence ou l’absence de l’insert uniquement.
- Informations sur la taille de l’insert.
- Informations sur l’orientation de l’insert.
-
En fonction de cela, différentes amorces de PCR sont conçues pour la PCR de la colonie.
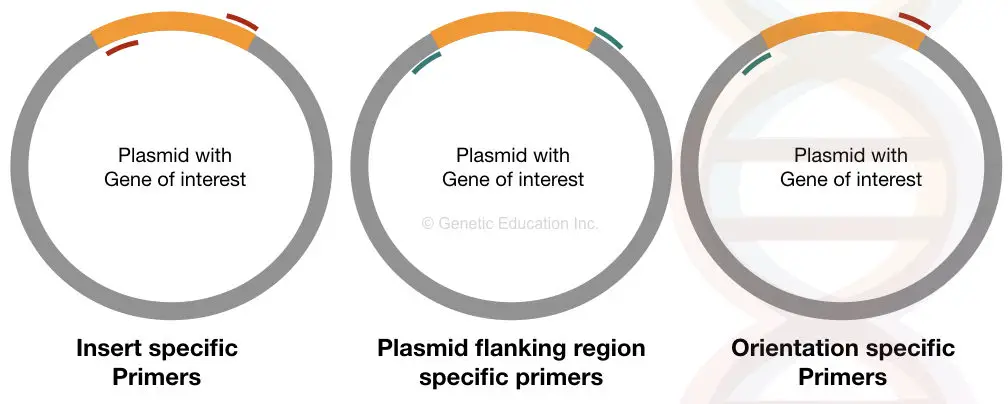
Les amorces spécifiques à l’insert se lient à l’emplacement spécifique des deux côtés de l’ADN inséré de notre intérêt. S’il est transféré correctement dans le plasmide, ces amorces peuvent s’y lier sinon il ne peut pas se lier.
Cet ensemble d’amorces fournit des informations concernant la présence ou l’absence de l’insert.
Les amorces spécifiques à l’orientation sont des amorces uniques dans lesquelles une amorce se lie à l’intérieur de l’insert et une autre amorce se lie à la séquence d’ADN plasmidique (séquence autre que l’ADN de l’insert).
Ce type d’ensemble d’amorces fournit des informations sur l’orientation de l’ADN inséré de notre intérêt. Si notre ADN d’insertion n’est pas correctement ligaturé dans le vecteur, l’amorce spécifique de ce côté de la séquence ne peut pas se lier et nous n’obtiendrons pas l’amplification.
Les amorces spécifiques au plasmide sont aussi importantes que les amorces spécifiques à l’orientation. Cet ensemble d’amorces est conçu à partir de la région flanquante de l’insert qui se lie à l’extérieur de l’ADN de notre intérêt.
Cet ensemble d’apprêt aide à déterminer la taille de l’insert. Il dilate des régions autres que l’ADN d’insertion.
La réaction de PCR pour effectuer la PCR de colonie est la suivante,
| Composant | Concentration | Quantité |
| Mélange maître (Spécial
pour la PCR des colonies) |
1X | 12 µl |
| Tampon de réaction PCR
Avec 2 Mm MgCl2* |
1X | 5 ΜL |
| Amorce avant | 22pM | 1µL |
| Amorce inverse | 10pM | 1µL |
| Surnageant | 3µL | |
| Eau | 3µL | |
| Total | ——— | 25µL |
La procédure de PCR de colonie:
Eh bien, la PCR de colonie n’a pas besoin d’ADN extrait.
Nous n’extrayons pas d’ADN ici. Au lieu de cela, plusieurs autres méthodes sont utilisées pour augmenter la sensibilité de la réaction.
Ok, pourquoi nous n’extrayons pas d’ADN pour l’ADN plasmidique ?
Parce que la raison est simple, la membrane cellulaire de la cellule bactérienne est très lisse.
Nous avions déjà discuté de la membrane cellulaire de la cellule bactérienne. Lisez-le ici: Différents types de méthodes d’extraction de l’ADN
Une bactérie contient une membrane cellulaire molle qui peut facilement être lysée en la chauffant ou en la centrifugant à grande vitesse.
De plus, nous n’avons pas besoin du propre ADN bactérien. Le plasmide circulaire circulant est présent dans le cytoplasme des bactéries, de sorte que des étapes de purification supplémentaires ne sont pas également nécessaires. En rompant la membrane cellulaire, notre ADN modèle est prêt pour l’amplification.
Ok, passons rapidement en revue la méthode pour obtenir un bon ADN plasmidique.
À l’aide du sélecteur stérile, prélevez plusieurs colonies bactériennes et transférez-les dans le tube d’Eppendorf.
Ajoutez-y maintenant du tampon TE et mélangez-le bien. Vous pouvez également utiliser D / W.
Chauffer l’échantillon au bain-marie bouillant pendant 20 minutes.
Le sommet doucement.
Centrifuger l’échantillon à grande vitesse pendant 2 minutes.
Transférez le surnageant dans un autre tube et utilisez-le comme ADN modèle.
Un échantillon de 20µL est ajouté dans la réaction.
Informations supplémentaires:
Pourquoi surnageant et non pas en granulés?
L’ADN est une biomolécule de la vie. L’ADN plasmidique est encore plus petit que l’ADN nucléaire bactérien. Il ne contient que plusieurs gènes allant jusqu’à 1000 pb à 20 000 pb.
Par conséquent, en le centrifugant uniquement, l’ADN plasmidique plus léger sort de la cellule et se dépose dans le surnageant tandis que le culot contient des protéines et de l’ADN nucléaire, nous ne l’utilisons donc pas.
Passons maintenant au point.
Notre plasmide est prêt pour l’amplification.
Dans une autre méthode,
Utilise directement la colonie bactérienne.
Cette méthode est une combinaison de la PCR Hotstart et de la PCR des colonies.
Les colonies bactériennes sont prélevées et ajoutées au tube réactionnel PCR.
Les tubes sont placés dans la machine PCR. Une étape de chauffage supplémentaire est ajoutée.
En le chauffant 5 à 7 minutes, l’ADN plasmidique sort de la cellule.
Maintenant, les amorces spécifiques à l’insert amplifient l’ADN que nous avons inséré. Et les amorces flanquantes amplifient le reste de l’ADN.
L’amplification se fait pendant 20 à 25 cycles. Les conditions de cyclisme pour la PCR de la colonie sont énumérées ci-dessous,
| Étapes de PCR | Dénaturation initiale | Dénaturation | Recuit | Extension | Extension finale |
| Température | 95 C | 95 C | 55-65 C | 72 C | 72 C |
| Temps | 3 min | 10 sec | 45 sec | 50 sec | 5 min |
| ——- | ——- | 25 cycles | —– | ——- |
Lisez l’article intéressant sur la PCR conventionnelle: Un Guide complet de la Réaction en chaîne de la polymérase
Conseils d’amélioration:
N’utilisez que quelques colonies, car de nombreuses colonies augmentent les chances de liaisons non spécifiques.
Utiliser un contrôle positif et un contrôle négatif.
Comme témoin positif utilisé l’amorce flanquante même si l’insert n’est pas présent, la réaction de PCR donne une bande d’ADN de l’ADN plasmidique qui indique que la réaction que nous avons préparée est correcte.
En tant que témoin négatif, utilisez le plasmide non transformé (plasmide sans ADN insert), cet ADN plasmidique n’est amplifié que si l’insert est présent.
Comme un insert utilise des séquences d’ADN courtes, des séquences d’ADN plus longues augmentent le risque de liaisons non spécifiques et d’échec de la réaction de PCR.
De plus, utilisez des programmes PCR plus courts.
L’application principale de la PCR des colonies est l’identification de la ligature correcte et l’insertion de l’ADN d’insertion dans les bactéries ainsi que dans le plasmide de levure.
Après l’achèvement de la réaction de PCR de colonie, les produits de PCR sont exécutés sur le gel d’agarose à 2%. Les résultats de l’expérience sont présentés dans la figure ci-dessous,
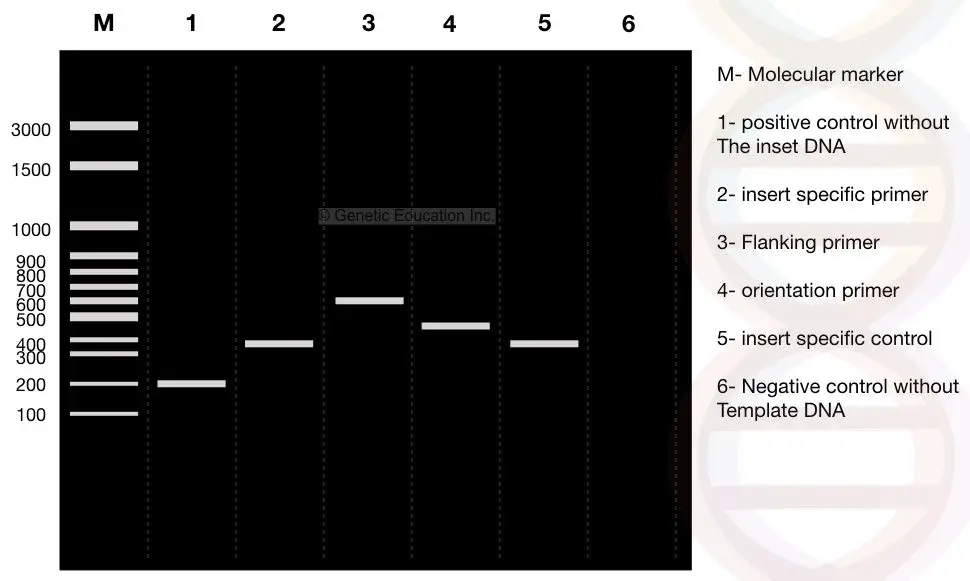
Maintenant, observez attentivement les résultats, le M est le marqueur d’ADN moléculaire 3000bp. Supposons que l’ADN de notre intérêt, “insert” soit un fragment de 400bp qui est inséré dans le plasmide.
Voir la voie 2 : le fragment de 400 pb de notre insert.
Nous avons conçu des amorces latérales à 100 bp des deux côtés de l’insert. Si l’amorce flanquante amplifie l’ADN avec l’insert, le produit est de 600 pb, voir la voie 3 (400 pb d’ADN insert + région flanquante de 200 pb).
Maintenant, voir la voie 1, c’est un contrôle positif sans l’insert ou un plasmide normal sans l’ADN transformé. Par conséquent, les amorces flanquantes n’amplifient que 200 pb d’ADN.
Voir la voie 1, fragment d’ADN 200bp sans insert (témoin positif).
Maintenant, observez la voie 4. La voie 4 est le résultat des amorces spécifiques à l’orientation. L’apprêt spécifique à l’orientation est une combinaison d’apprêt spécifique à l’insert et d’apprêt spécifique à la région flanquante.
Une amorce provenant de l’ADN d’insertion et une amorce provenant de l’amorce spécifique à la région flanquante sont sélectionnées pour une amplification d’amorce spécifique à l’orientation.
Par conséquent, un fragment de 100 pb de l’amorce de la région flanquante et 400 pb de l’ADN d’insertion sont amplifiés et un fragment d’ADN de 500 pb est observé dans la voie 4.
La voie 5 est le contrôle spécifique de l’insert qui donne un fragment d’ADN de 400 pb.
La voie 6 est le contrôle négatif sans le gabarit. En utilisant un contrôle négatif, toute contamination peut être identifiée. Le tube de réaction contient tous les ingrédients sauf le gabarit. Donc, idéalement, aucune bande d’ADN n’est présente dans cette voie.
Si une bande d’ADN est observée, l’échantillon est contaminé.
Avantages de la PCR des colonies:
- La technique est rapide et rentable.
- De plus, la précision et la spécificité de la technique sont plus élevées.
- La mise en place est simple tout comme la PCR conventionnelle, l’extraction de l’ADN et la purification des plasmides comme des étapes laborieuses ne sont pas nécessaires.
- Pas besoin de digestion par restriction pour l’identification de l’ADN de l’insert.
- Toute l’expérience peut être terminée en 90 minutes.
Inconvénients de la PCR des colonies:
- La méthode est rentable, rapide et fiable, Cependant, aucune mutation dans l’insert ne peut être détectée.
- De plus, les informations de séquence ne peuvent pas être obtenues par PCR de colonie. nous devons faire un séquençage pour la confirmation de la transformation de l’ADN.
- Le risque de résultats faussement positifs est élevé.
En savoir plus;
- Qu’est-ce qu’une PCR multiplex ?
Après la fin de l’expérience, l’échantillon est envoyé pour le séquençage où la séquence d’ADN de notre intérêt peut être déterminée.
nous pouvons même effectuer une PCR multiplex en combinant à la fois des amorces spécifiques à l’insertion et des amorces spécifiques au plasmide.
Conclusion:
Bien que la PCR des colonies soit le meilleur choix pour l’identification du transfert de gènes, la seule technique de PCR des colonies n’est pas suffisante pour interpréter les résultats. Il est possible que certaines des mutations présentes dans l’insert ne puissent pas être détectées par la PCR.
Pour confirmer les résultats, un séquençage de l’ADN est nécessaire. Après avoir déterminé l’ordre de séquence, nous pouvons dire si notre gène d’intérêt est inséré correctement ou non.