創造研究研究所
 おそらく類人猿から進化する人間のために使用されるより一般的な議論の一つは、染色体 この概念の推進力は、類人猿が染色体の余分なペアを持っているという進化の問題です—人間は46を持っていますが、類人猿は48を持っています。 人間が300万年から600万年前に類人猿のような生き物から進化したならば、進化の物語の壮大な計画の単なるブリップ、なぜ人間と類人猿はこの不一致を持っていますか?
おそらく類人猿から進化する人間のために使用されるより一般的な議論の一つは、染色体 この概念の推進力は、類人猿が染色体の余分なペアを持っているという進化の問題です—人間は46を持っていますが、類人猿は48を持っています。 人間が300万年から600万年前に類人猿のような生き物から進化したならば、進化の物語の壮大な計画の単なるブリップ、なぜ人間と類人猿はこの不一致を持っていますか?
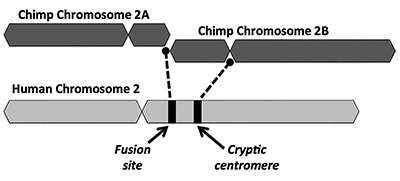
進化的解決策は、2つの小さな猿のような染色体(2Aと2Bという名前)のエンドツーエンド融合がヒト染色体2を産生することを提案している(図1)。 融合の概念は、科学者が顕微鏡下でヒトとサルの染色体の類似点を調べた1982年に最初に生まれました。 技術はやや粗雑でしたが、アイデアを軌道に乗せるのに十分でした。1
いわゆる融合サイト
可能な融合イベントの最初の実際のDNA署名は、1991年にヒト染色体番号2.2の研究者が漠然と可能な融合に似ていたテロメアのような末端配列の小さ テロメアは、染色体の末端で何度も何度も繰り返されるDNA文字TTAGGGの六塩基配列である。
しかし、融合の署名は、自然界で時折発生する実際の融合に基づいた謎のものでした。 生きている動物のすべての文書化された融合は、染色体に位置し、破損および融合に見られる衛星DNA(satDNA)と呼ばれる特定のタイプの配列を含む。3-5ヒト染色体2の融合シグネチャは、この証拠となるsatDNAを欠いていた。6
もう一つの問題は、融合部位のサイズが小さいことであり、これはDNA文字の長さがわずか798である。 染色体の末端のテロメア配列は5,000-15,000塩基の長さである。 二つの染色体が融合していた場合、あなたは10,000から30,000塩基の長さの融合テロメア署名が表示されるはずです—798ではありません。
小さなサイズが融合物語の問題であるだけでなく、署名はテロメアの明確な融合を実際に表すものではありません。 図2は、798塩基融合部位のdna文字を六塩基(DNA文字)無傷のテロメア配列と太字で強調したものを示している。 融合配列を、同じサイズの原始的な融合シグネチャのものと比較すると、全体的に70%しか同一ではない。

世俗的な研究者はこの不一致を指摘し、融合部位を有意に「退化している」とラベル付けした。”7人間の進化の標準的な理論モデルを考えると、それは約98-99%同一であり、70%ではないはずです。 この発見を説明している研究者は、”核融合部位の反復配列は、テロメアで見つかったnear perfect array of(TTAGGG)nから有意に(14%)退化している”とコメントし、関連する質問をした”核融合がテロメアの反復配列内で6Mya未満で発生した場合、核融合部位の配列はなぜ退化しているのですか?”7著者が引用した14%の変性は、798塩基全体ではなく、六つの塩基配列自体の破損を指していることに留意すべきである。
遺伝子内の融合部位?
融合部位についての最も顕著な反進化的発見は、その位置と実際に何をしているかであることが判明した。 この発見は、疑惑の融合部位を取り巻く614,000塩基のDNA配列の詳細な分析を報告した研究論文を読んでいる間に起こりました。 私は、融合部位が遺伝子の中に位置していたことを図の一つで気づいた、と非常に顕著にこの奇妙さも、論文のテキストで認められていませんでした。8
このような発見は非常に注目に値する。 おそらく、この情報は進化の棺の釘であったでしょう、いわば、それが研究者がそれを議論することを拒否した理由です。 この大きな異常は、融合サイトにもっと詳しく調べるように私に促しました。 この論文は2002年に出版され、私は2013年にそれに気づきました。 その間にヒトゲノムの構造と機能に関する膨大な量のデータが公開されており、明らかにする必要がある話にははるかに多くの可能性がありました。
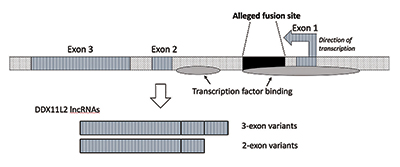
私はさらなる研究を行ったとき、私は融合部位が現在DDX11L2と呼ばれるRNAヘリカーゼ遺伝子の内側に位置していたことを確認しました。 植物や動物のほとんどの遺伝子は、エクソンと呼ばれる断片にコードセグメントを持っているので、代わりにスプライスすることができます。 エクソンの添加または排除に基づいて、遺伝子は様々な生成物を産生することができる。 エクソン間の介在領域はイントロンと呼ばれ、多くの場合、遺伝子機能を制御する様々なシグナルおよびスイッチを含む。 疑惑の融合部位は、DDX11L2遺伝子の最初のイントロンの内側に位置しています(図3)。9
DNA分子は二本鎖であり、プラス鎖とマイナス鎖を有する。 また効率および機能を高めている間情報密度を最大にすることをこのように設計した。 その結果、反対の鎖に異なる方向に走っている遺伝子が存在する。 結局のところ、DDX11L2遺伝子はマイナス鎖にコードされています。 ヒトの遺伝子はスイスアーミーナイフのようなものであり、様々なRnaを産生することができるため、DDX11L2遺伝子の場合、二つのエクソンからなる短い変異体と三つの長い変異体を産生する(図3)。9
融合部位は遺伝子プロモーター
このDDX11L2遺伝子は何をしているのでしょうか? 私の研究は、それが少なくとも255の異なる細胞または組織タイプで発現していることを示しました。9それはまた、他の様々な遺伝子と共発現(同時にオン)され、細胞外マトリックスにおける細胞シグナル伝達および血球産生に関連するプロセスに関連 様々な細胞プロセスの遺伝学に関連する機能的遺伝子内のいわゆる融合配列の位置は、それが頭から頭へのテロメア融合の偶発的な副産物であると 遺伝子は壊滅的な染色体融合によって形成されていません!
さらに驚くべきことは、融合サイト自体が機能的であり、重要な工学的目的を果たしていることです。 この部位は、実際には遺伝子活性を制御するためのスイッチとして作用する。 この点で、豊富な生化学的データは、転写因子と呼ばれる12の異なるタンパク質が遺伝子のこのセグメントを調節することを示した。 これらの一つは、転写と呼ばれるプロセスでDNAからRNA分子をコピーする主な酵素であるRNAポリメラーゼII以外のものではありません。 この発見をさらに支持するのは、転写の実際のプロセスがいわゆる融合部位の領域内で開始するという事実である。
技術的には、我々は主張された融合部位での活性をプロモーター領域と呼ぶだろう。 プロモーターは、それらをオンにする遺伝子の開始時の主要なスイッチであり、RNAポリメラーゼがRNAを作成し始める場所でもあります。 多くの遺伝子にDDX11L2遺伝子のような代わりとなるプロモーターがあります。
DDX11L2遺伝子には実際には2つの転写因子結合領域があります。 最初のものは最初のエクソンの直前のプロモーターにあり、第二のものは融合部位配列に対応する最初のイントロンにある。 DDX11L2遺伝子自体が複雑に制御されているだけでなく、疑わしい融合配列が重要な役割を果たしているだけでなく、生成されたRNA転写物さえも非常に Rna自体は、マイクロrnaと呼ばれる小さな調節分子のクラスのための結合および制御部位の多種多様を含んでいます。9
機能的な内部テロメア配列はゲノム全体にあります
内部に位置するテロメア配列の存在は、ヒトゲノム全体に見られます。 これらの一見場違いテロメアの繰り返しは間質テロメアと呼ばれています。 これらの配列の存在は、融合部位のアイデアのための別の課題を提示する。 これは、融合部位におけるテロメアリピートの非常に少数がタンデムで発生するという事実です。 図2に記載されているように、798塩基融合部位の配列には、二つの繰り返しが実際に直列にあり、三つ以上の繰り返しを持たないいくつかの例が含まれています。 しかし、ヒトゲノム全体には他にも多くの間質テロメア部位があり、その繰り返しは三から十倍以上の完全なタンデムで起こる。10-11
染色体の末端での役割に加えて、間質テロメア反復が遺伝子発現に関連するゲノムにおいて重要な機能を果たす可能性があるようである。 最近の研究プロジェクトでは、私はヒトゲノム全体のテロメアリピートを同定し、遺伝子活性のための機能的生化学的情報を含む多様なデータセットとそのゲノム位置を交差させた。ゲノム全体で12文字通り何千もの内部テロメアリピートは、遺伝子発現の特徴と直接関連していた。 主張された融合部位で発生する転写因子結合および遺伝子活性の同じタイプは、他の多数の間質テロメア反復でゲノムワイドに発生していた。 明らかに、これらのDNAの特徴は進化の事故ではなく、意図的かつインテリジェントに設計された機能コードです。
遺伝子内の偽の不可解な動原体
融合モデルのもう一つの重要な問題は、余分な動原体領域の署名のための実行可能な証拠の欠如です。 動原体は、細胞分裂の間に重要な役割を果たす染色体の切片であり、しばしば中央の位置にある。 図1に示されているように、新たに形成されたキメラ染色体は、2つの染色体の頭から頭への融合の直後に2つの動原体部位を有していたであろう。 このような場合、動原体の一方は機能的であり、他方は無効である。 二つの活性動原体の存在は、染色体のための悪いニュースであり、機能不全や細胞破壊につながるでしょう。
興味深いことに、ヒト2番染色体上の不可解な(無効な)動原体の証拠は、テロメアが豊富な融合部位の証拠よりもさらに弱い。 進化論者は、第二動原体が急速に選択されていたであろうと主張することによって、明確に区別可能な非機能的な二次動原体の欠如を説明します。 その後、無効な動原体は、ゲノムに有用な何かをすることによってもうそれに置かれた機能的拘束がなかったので、時間の経過とともに悪化したで
しかし、配列変性の任意の段階での第二の残存動原体の証拠は、進化のパラダイムにとって問題である。 機能性セントロメア配列は、アルフォイド配列と呼ばれる反復型のDNAで構成され、各アルフォイドリピートは約171塩基長である。 他の人が動原体に特異的である一方で、アルフォイドリピートのいくつかのタイプは、すべてのゲノム上で発見されています。 ヒト第2染色体上の不可解な動原体部位で見つかった配列の構造は、機能的なヒト動原体に関連するものと一致しない。13進化モデルにとってさらに悪いことに、彼らはchimpゲノムに非常に類似した対応物を持っていないということです—彼らは人間に特異的です。13
疑惑の化石動原体も、実際のものと比較して非常に小さい。 正常な人間の動原体のサイズは250,000と5,000,000塩基の間の長さの範囲です。14疑わしい不可解な動原体はわずか41,608塩基の長さですが、アルファイドリピートでさえない三つの異なる領域があることに注意することも重要です。15これらのうちの2つはレトロエレメントと呼ばれ、1つは5,957塩基長のLPA3/LINE repeatであり、もう1つは2,571塩基を持つSVA-E要素である。 これらの非アルフォイド配列の挿入を減算すると、実際の動原体の長さの割合であるわずか33,080塩基の長さが得られます。
化石動原体の考えの最も深刻な進化上の問題は、しかし、主張されている融合部位のように、それが遺伝子の中に位置しているということです。 疑わしい不可解な動原体はANKRD30BL遺伝子の中に位置し、その配列は遺伝子のイントロン領域とエクソン領域の両方にまたがる。12,15
実際、エクソンの中に着地する化石動原体配列の一部は、実際に得られた遺伝子のタンパク質のアミノ酸をコードしています。 この遺伝子は、細胞膜に埋め込まれた受容体タンパク質と関連して、細胞骨格と呼ばれる細胞内のタンパク質の構造ネットワークの相互作用に関16いわゆる化石または不可解な動原体が重要なタンパク質コード遺伝子の中の機能領域であるという事実は、それが消滅した動原体であるという考
まとめ: 融合なし
融合と化石の動原体部位の特徴が混乱し、サイズが小さいため、それらの配列が進化的に古代の染色体融合に由来していることは非常に疑問である。 それだけでなく、それらは遺伝子内の機能的配列を表しています。 この融合部位は、DDX11L2長い非コードRNA遺伝子内のプロモーターと呼ばれる重要な遺伝的スイッチであり、いわゆる化石セントロメアは大きなアンキリンリピートタンパク質コード遺伝子内のコード配列と非コード配列の両方を含む。
これは神話上の融合のアイデア全体に対する否定できない二重の強打であり、その妥当性を完全に破壊する。 圧倒的な科学的結論は、融合は決して起こらなかったということです。
- Yunis,J.J.and O.Prakash. 1982. 人間の起源:染色体の絵の遺産。 科学だ 215 (4539): 1525-1530.
- Ijdo,J.W.et al. 1991. ヒト第2染色体の起源:先祖のテロメア-テロメア融合。 国立科学アカデミーの学位を取得している。 88 (20): 9051-9055.
- Chaves,R.et al. 2003. 分子細胞遺伝学的分析とヒツジにおける新規8;11転座の動原体衛星組織:バイアーム染色体進化の可能な中間体。 哺乳類のゲノム。 14 (10): 706-710.
- Tsipouri,V.et al. 2008. 比較配列解析は、インドのmuntjacゲノムにおける先祖の染色体融合のサイトを明らかにした。 ゲノムバイオロジー… 9(10):R155.
- Adega,F.,H.Guedes-Pinto,R.Chaves. 2009. 家畜の核型進化における衛星DNA-臨床的考察。 細胞遺伝学およびゲノム研究。 126 (1-2): 12-20.
- Tomkins,J.P.and J.Bergman. 2011. テロメア:老化のための含意および理性的な設計のための証拠。 作成のジャーナル。 25 (1): 86-97.
- Fan,Y.et al. 2002. 2q13–2q14.1および他の人間の染色体のParalogous領域の祖先の染色体の融合の場所のゲノム構造そして進化。 ゲノム研究。 12 (11): 1651-1662.
- Fan,Y.et al. 2002. ヒト染色体2q13–2q14.1およびパラログ領域における祖先染色体融合部位の遺伝子含量および機能。 ゲノム研究。 12 (11): 1663-1672.
- Tomkins,J.P.2013. 主張されたヒト染色体2″融合部位”は、複合体および高度に発現された遺伝子否定融合の内部に活性DNA結合ドメインをコードしている。 研究日誌に答えます。 6: 367-375.
- Azzalin,C.M.,S.G.Nergadze,e.Giulotto. 2001. ヒト染色体内テロメア様リピート:配列の組織と起源のメカニズム。 染色体 110: 75-82.
- Ruiz-Herrera,A.et al. 2008. テロメアは端から遠く離れて繰り返されます:起源のメカニズムと進化における役割。 細胞遺伝学およびゲノム研究。 122 (3-4): 219-228.
- Tomkins,J.P.2018. コンビナトリアルゲノムデータは、ヒト染色体2進化融合に反論し、間質テロメア繰り返しのための機能設計のモデルを構築します。 第八回創造論国際会議の議事録で。 J.H.Whitmore,ed. ピッツバーグ、ペンシルベニア州:創造科学フェローシップ、222-228。
- Tomkins,J.and J.Bergman. 2011. ヒト進化の染色体2融合モデル-パート2:ゲノムデータの再解析。 作成のジャーナル。 25 (2): 111-117.
- Aldrup-Macdonald,M.E.and B.A.Sullivan. 2014. ヒト動原体ゲノミクスの過去、現在、そして未来。 遺伝子(バーゼル)。 5 (1): 33-50.
- Tomkins,J.P.2017. 批判者を暴く:ヒト染色体2融合の反論に関する批判と難読化への対応。 研究日誌に答えます。 10: 45-54.
- ヴォロニン、D.A.およびE.V.Kiseleva。 2008. Ankyrinの繰り返しを含んでいる蛋白質の機能役割。 細胞および組織生物学。 49 (12): 989-999.
* トムキンス博士は、Institute for Creation Researchのライフサイエンスディレクターであり、クレムソン大学で遺伝学の博士号を取得しています。