Aplicação de proteína-fragmento de complementação ensaios em biologia celular
Introdução
vias Bioquímicas são realmente de sistemas dinamicamente montagem e desmontagem dos complexos de proteína, e, assim, grande parte da moderna pesquisa biológica está preocupado com o como, o quando, e onde as proteínas interagem com outras proteínas envolvidas nos processos bioquímicos. A demanda por abordagens simples para estudar interações proteína-proteína, particularmente em grande escala, tem crescido recentemente com o progresso em projetos de genoma, uma vez que a associação do desconhecido com produtos genéticos conhecidos fornece uma maneira crucial de estabelecer a função de um gene. Foi com este Desafio em mente que o nosso laboratório desenvolveu testes de complementação de fragmentos de proteínas (PCAs). Nesta estratégia, duas proteínas de interesse (proteínas A E B) são fundidas a fragmentos complementares de uma proteína repórter (uma enzima, proteína fluorescente, etc.). Se as proteínas a e B interagem, os fragmentos de repórter são reunidos, dobrados na estrutura nativa do repórter e reconstituir a sua actividade (Figura 1). As proteínas do PCA reporter foram escolhidas como aquelas que produzem uma variedade de atividades detectáveis, incluindo sinais fluorescentes, luminescentes e colorimétricos, bem como testes simples de seleção de sobrevivência (1-14). Demonstrámos que a estratégia APC tem as seguintes capacidades: (i) permite a detecção de interacções proteína-proteína in vivo e in vitro em qualquer tipo de célula.; (ii) permite a detecção da proteína-proteína interações apropriadas compartimentos subcelulares ou organelas; (iii) permite a detecção de interações que são especificamente induzidos em resposta ao desenvolvimento, nutricionais, ambientais, ou indução hormonal de sinais; (iv) permite o acompanhamento da cinética e equilíbrio aspectos da proteína de montagem em células; e (v) que permite a triagem para a nova proteína-proteína medicamentosas em qualquer tipo de célula (2,3,6,9)(15-19).
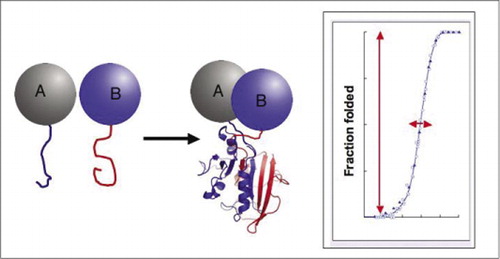
princípio
demonstrámos o princípio de PCA começando com a enzima dihidrofolato redutase (DHFR) como repórter (1). Era óbvio que se a dobragem da enzima a partir dos seus fragmentos (tal como detectada pela reconstituição da actividade) dependesse absolutamente da ligação entre as proteínas que interagem, então o sistema descrito seria, de facto, um detector das interacções. Nós e outros temos demonstrado desde então que este princípio pode ser generalizado a uma série de enzimas, incluindo Gaussia e Renilla luciferases, TEM β-lactamase, bem como proteína fluorescente verde (GFP) e suas variantes (1-14). Uma característica crucial dos fragmentos de APC é que eles são projetados para não dobrar espontaneamente sem ser trazido para a proximidade próxima pela interação das proteínas a que eles são fundidos (1,20). Se ocorresse a dobragem espontânea, o PCA simplesmente não funcionaria. A dobragem espontânea levaria a um sinal falso positivo, uma situação que confundiria irremediavelmente a interpretação das telas da biblioteca in vivo (prevista para ser uma aplicação importante). Em contraste com PCA, existem sistemas de doseamento baseados em β-galactosidase e inteinas divididas que se assemelham a PCA, mas que são conceptualmente e praticamente diferentes (21,22). Em ambos os casos, a ocorrência natural bem conhecida e a associação espontânea das subunidades das enzimas são fundidas a proteínas interagindo. O problema central aqui é que as subunidades, ainda que pouco associadas, são sempre capazes de fazê-lo até certo ponto, o que significa que há um fundo constante de montagem espontânea.
limitações
a estratégia do APC é geral, no sentido de que não se restringe a um único repórter enzimático, e foi concebido em várias formas diferentes, cada uma das quais é mais adequada para abordar uma questão específica. Por exemplo, os PCAs simples de seleção de sobrevivência, como os baseados em DHFR, são mais úteis para a seleção de bibliotecas, enquanto os PCAs de luminescência ou de leitura de fluorescência são melhores para os estudos da dinâmica espacial e temporal dos complexos proteicos. Uma vez que as proteínas de fusão podem ser expressas em células que são modelos relevantes para o estudo de uma via bioquímica específica, elas estão provavelmente no seu estado biológico nativo, incluindo as modificações pós-tradução corretas (obviamente os próprios fragmentos de APC não devem interferir com o alvo ou modificação das proteínas, e isso deve ser testado).
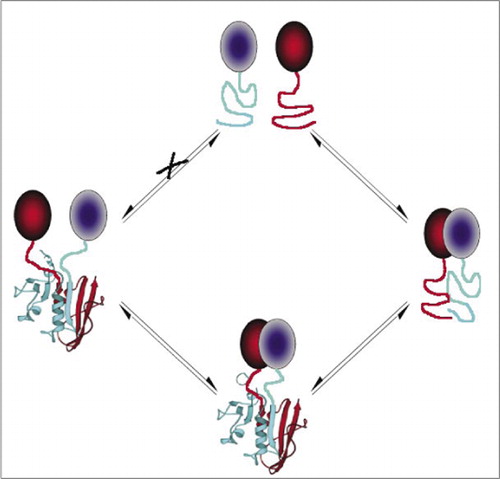
Entre os mais simples e portanto mais populares PCAs são aqueles baseados em proteínas fluorescentes (como GFP e variantes), porque o sinal é fornecido pela intrínseca fluoróforo (7-9)(14,15,17,23). No entanto, as proteínas fluorescentes devem ser expressas em níveis elevados para garantir que o sinal está acima da fluorescência celular de fundo, e os PCAs de proteína fluorescente têm sido demonstrados como irreversíveis, o que pode ser útil (aprisionamento e visualização de complexos raros), mas também pode levar a uma má interpretação da rotatividade ou localização de proteínas interagindo (8,23,24). Por outro lado, os APC baseados em DHFR e β-lactamase como repórteres têm sido demonstrados, com base em evidências indiretas, ser reversíveis após interrupção das interações, enquanto um APC baseado em Gaussia luciferase tem sido diretamente mostrado ser reversível (2,3,6). A reversibilidade do APC permite assim a detecção de aspectos cinéticos e de equilíbrio da montagem e desmontagem de complexos proteicos em células vivas (Figura 2).
controles padrão para um estudo PCA
a estratégia PCA requer que os fragmentos da proteína repórter para montar e dobrar após as proteínas de interesse ter formado um complexo. A montagem e dobragem correta do Repórter depende da recuperação da geometria estrutural intrínseca às proteínas do repórter e do complexo formado pelas proteínas interagindo. Esta é uma das principais distinções dos ensaios APC em comparação com a transferência de energia por ressonância de fluorescência (FRET) ou transferência de energia por ressonância de bioluminescência (BRET) ou dois ensaios híbridos de levedura, e Esta característica permitiu-nos realizar um estudo baseado na estrutura do receptor da eritropoietina (19). Nós tipicamente inserimos um polipeptídeo flexível de 10 aminoácidos consistindo de (Gly.Gly.Gly.Gly.Ser) 2 entre a proteína de interesse e o fragmento do Repórter PCA (para ambas as perfusões). Este vinculador foi escolhido porque é o mais flexível possível, e temos observado empiricamente que vinculadores deste comprimento suficientemente longo para permitir fragmentos de encontrar uns aos outros e desistir, independentemente do tamanho da interação de proteínas para que os fragmentos são fundidas (16).
para assegurar que não ocorram respostas não específicas, deve ser realizado um conjunto de controlos. Esses controles podem incluir o seguinte, embora o primeiro seja o mais importante: (i) proteínas não interagindo. Não deve ser observada uma resposta APC se forem utilizadas proteínas não interagentes como parceiros APC; nem a sobre-expressão de uma proteína que não interage por si só deve competir pela interacção conhecida. II) mutações na interface proteica do Parceiro. Um ponto ou mutação por eliminação de um parceiro que é conhecido por perturbar uma interacção deve também prevenir uma resposta APC. – concorrência. Uma resposta PCA deve ser diminuída pela sobreexpressão simultânea de uma ou outra das proteínas interagindo que não é fundida a um fragmento de PCA complementar. (iv) troca de fragmentos. Deve ocorrer uma interacção observada entre duas proteínas, mesmo que estas sejam trocadas com os respectivos fragmentos reporter.
Application of PCA in Protein Design: Library vs. Library Screening for Optimally Interacting Proteins
Among the first applications of a PCA was to a protein design problem. O ensaio DHFR PCA foi usado em Escherichia coli para visualizar duas bibliotecas de sequências de formação complementar de leucina zipper com 1010 potenciais pares interagindo, das quais poderíamos praticamente cobrir 106. Demonstrámos que a tela PCA selecionada tanto para a melhor especificidade de ligação, como para a solubilidade e expressão de fechos interagindo (18,25). A característica mais importante desta abordagem é que foi possível simultaneamente colocar duas bibliotecas umas contra as outras, um processo que não é facilmente alcançado com telas híbridas de levedura comparáveis. A simplicidade desta abordagem e a natureza específica das informações obtidas sobre a estratégia de design sugerem uma ampla utilidade do PCA DHFR no design de proteínas e experiências de evolução dirigida. Ele também mostra que o PCA complementa estratégias de exibição de FAG, uma vez que todos os testes de seleção, otimização e rigor são feitos in vivo, tornando esta abordagem facilmente executada.Um primeiro passo na definição da função de um novo produto genético é determinar as suas interacções com outros produtos genéticos. No entanto, uma abordagem puramente baseada na interação proteica (como o híbrido de levedura 2) é limitada, porque ele só diz que duas proteínas interagem, sem fornecer qualquer outra informação que poderia ligar uma proteína à sua função. Por isso, temos mostrado que o APC pode ser usado em uma estratégia de triagem de biblioteca cDNA que combina uma tela de interação proteica baseada em células simples com testes funcionais específicos que fornecem validação inicial da relevância biológica da interação (9). O primeiro passo consiste em rastrear interações físicas entre a isca e uma biblioteca de proteínas presas codificadas cDNA, monitorando a reconstituição do Repórter PCA em células vivas intactas. Uma característica importante deste primeiro passo é que as interações podem ser detectadas diretamente e entre com a duração total de proteínas nas células em que a isca proteína normalmente funções, garantindo assim que necessário subcelular de segmentação, posttranslational modificações e interações com outras proteínas, pode ocorrer. Obviamente, para a validade experimental, os fragmentos de APC devem ser demonstrados não interferindo com o alvo ou modificação das proteínas. Na segunda etapa, a interação proteica pode ser funcionalmente validada, como se segue: : em primeiro lugar, a interacção proteica, detectada pela APC, deve ser perturbada por agentes, tais como hormonas ou inibidores específicos, que são conhecidos por modular a via bioquímica específica em que as proteínas participam. Nós demonstramos isso para o PCA DHFR e usamos esta propriedade para mapear vias de sinalização em células de mamíferos vivos (16). Em segundo lugar, a localização subcelular da interação proteica, novamente detectada por PCA, pode ser alterada por agentes que modulam a via. Assim, a estratégia de pré-selecção baseada no APC combina um simples passo de pré-selecção com ensaios funcionais directos. Nós e outros aplicamos esta estratégia para a identificação de novos substratos ou reguladores da proteína serina/treonina cinase, PKB/Akt (9,15,26,27).
usando PCA como uma régua Molecular: estudos de receptores
uma característica especial das estratégias de PCA é que, se sabemos a estrutura tridimensional da enzima repórter, é possível prever com precisão o quão próximos os fragmentos devem ser para garantir que a enzima dobrará corretamente e terá uma atividade mensurável. Este fato foi colocado para trabalhar para teste estrutural alostéricos modelo para a ativação do dimeric do receptor de eritropoietina (EpoR) usando o DHFR PCA, e a abordagem pode ser estendida para estudar alostéricos transições em dimeric ou multimeric proteína interfaces (19). No caso EpoR, os domínios transmembranar do receptor dímero mostraram ser separados por 73 Å, como observado na estrutura cristalina do EpoR não-ligado. Foi fundamentado que, se esse estado inativo existia na membrana de uma célula viva, em seguida, DHFR fragmentos fundidos para a C termini de domínios transmembrana iria dobrar somente se um ligante induzida por uma conformação mudanças que permitem que os fragmentos para chegar perto o suficiente para garantir que o preciso estrutura tridimensional da DHFR poderia ser formado (19,28). Isto exigiria que o n termini dos fragmentos fosse 8 Å separado. A inserção de peptídeos de linker flexíveis entre o domínio transmembranar e os fragmentos de DHFR permitiu-nos sondar a distância entre os pontos de inserção do dímero de domínio extracelular e confirmar que linkers de tempo suficiente para chegar a 73 Å eram necessários para que o DHFR se dobrasse dos seus fragmentos.
mapeamento de redes bioquímicas
máquinas bioquímicas celulares para metabolismo, cascatas sinalizadoras e ciclo celular são exemplos de conjuntos dinâmicos e desmontagem de complexos macromoleculares. Estes são definidos agrupando proteínas interagindo de acordo com as suas respostas semelhantes a um conjunto de perturbações (hormonas, metabolitos, inibidores enzimáticos, etc.). Interacções proteína-proteína podem ser usadas para ligar uma proteína de função desconhecida a proteínas que são conhecidas por estarem envolvidas num processo bioquímico conhecido. Demonstrámos que o perfil farmacológico (efeitos de monitorização de medicamentos específicos da via e de hormonas proteicas nas interacções proteína-proteína) e a determinação da localização celular das interacções proteína-proteína podem ser alcançados através do uso de PCAs.(9)(15-17)(26). A análise destes resultados permite uma representação de como as redes bioquímicas evoluem no tempo e no espaço e em resposta a estímulos específicos. Como prova de princípio, relatamos a aplicação desta estratégia ao mapeamento de uma via de transdução de sinal mediada pelo receptor tirosina cinases (RTKs) (16). Os perfis farmacológicos e a localização celular das interações observadas nos permitiram colocar cada produto genético em seu ponto relevante nas vias (Figura 3). A partir dos resultados de nossa análise, um mapa da organização da rede RTK surgiu que era consistente com os modelos existentes, mas que também incluiu várias interações novas. A capacidade de monitorar uma rede de interações proteicas em células vivas contendo todos os Componentes da via subjacente estudada revelou conexões ocultas, não observadas antes, apesar do intenso escrutínio desta rede. Os resultados apresentados demonstram que a estratégia APC tem as características necessárias para uma validação geral da função genética e uma estratégia de mapeamento de vias. Uma aplicação recente de um maior conjunto de APC permitiu o desenvolvimento de uma abordagem geral para ligar as acções das drogas em vias de sinalização específicas e detectar actividades imprevistas das drogas (17).
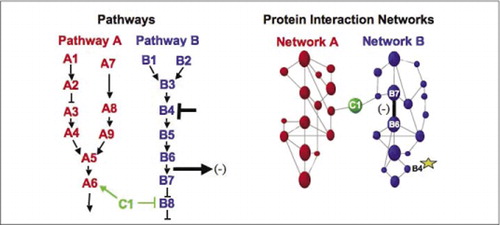
(à esquerda) as acções de um agente perturbador inibitório que actua na proteína B4 (barra T), são detectadas a jusante por uma alteração na interacção das proteínas B6 E B7 umas com as outras (seta). Neste caso, o efeito da perturbação é uma diminuição no número de proteína interagindo (-) como detectado por um repórter dessa interação (sinal de saída da interação como detectado por PCA sentinel, por exemplo). No entanto, o efeito pode ser igualmente positivo, dependendo das consequências da inibição da proteína a montante. Dentro da rede de interação proteica para a via B, uma perturbação da proteína B4 (estrela) de alguma forma se propaga através da rede para de alguma forma afetar a ligação (barra larga) entre as proteínas B6 E B7. Isto não implica que a proteína B4 interage fisicamente com B6 ou B7; A propagação de um efeito através da rede de interação proteica pode ser devido a ligações físicas diretas ou a processos enzimáticos não óbvios na rede.
conclusão
o desenvolvimento e a aplicação do APC estão ainda em curso. Por exemplo, além dos conjuntos limitados, embora informativos, de aplicações descritas aqui, a estratégia está sendo aplicada à triagem em larga escala de genomas inteiros. Problemas mais sofisticados de design de proteínas e dobragem de proteínas estão sendo explorados, incluindo estudos dos fatores que controlam a seleção de sequências para interações ótimas entre proteínas, proteínas e ácidos nucleicos, e proteínas e pequenas moléculas orgânicas. O APC é uma abordagem experimental muito geral e flexível, pelo que devemos esperar ver um número crescente de novas aplicações desta ferramenta básica para a biologia molecular e celular num futuro próximo.
reconhecimentos
Stephen Michnick detém a cadeira de Investigação do Canadá em genómica Integrativa. A pesquisa citada do nosso laboratório foi financiada pelos Institutos canadenses de pesquisa de saúde.
- 1. Pelletier, J. N., F. X. Campbell-Valois, and S. W. Michnick. 1998. Remontagem dirigida ao domínio de oligomerização da dihidrofolato redutase activa a partir de fragmentos concebidos racionalmente. Procedimento. Natl. Acad. Ciência. USA 95: 12141-12146.Crossref, Medline, CAS, Google Scholar
- 2. Remy, I. and S. W. Michnick. 1999. Selecção Clonal e quantificação in vivo das interacções proteicas com ensaios de complementação de fragmentos proteicos. Procedimento. Natl. Acad. Ciência. USA 96: 5394-5399.Crossref, Medline, CAS, Google Scholar
- 3. Galarneau, A., M. Primeau, L. E. Trudeau, and S. W. Michnick. 2002. A complementação do fragmento de proteína Beta-lactamase efectua-se como sensores in vivo e in vitro de interacções proteicas. Conversao. Biotechnol. 20:619–622.Crossref, Medline, CAS, Google Scholar
- 4. Wehrman, T., B. Kleaveland, J. H. Her, R. F. Balint, and H. M. Blau. 2002. Interacções proteína-proteína monitorizadas nas células de mamíferos através da complementação de fragmentos da enzima beta-lactamase. Procedimento. Natl. Acad. Ciência. USA 99: 3469-3474.Crossref, Medline, CAS, Google Scholar
- 5. Spotts, J. M., R. E. Dolmetsch, and M. E. Greenberg. 2002. Imagiologia por intervalo de tempo de uma interacção proteína-proteína dependente da fosforilação dinâmica nas células de mamíferos. Procedimento. Natl. Acad. Ciência. USA 99: 15142-15147.Crossref, Medline, CAS, Google Scholar
- 6. Remy, I. and S. W. Michnick. 2006. Um ensaio de interacção proteína-proteína altamente sensível com base na Gaussia luciferase. Conversao. Methods 3: 977-979.Crossref, Medline, CAS, Google Scholar
- 7. Ghosh, I., A. D. Hamilton, and L. Regan. 2000. Reposicionamento da proteína antiparalel leucina zipper: aplicação à proteína verde fluorescente. J. Am. Chem. Soc. 122:5658–5659.Crossref, CAS, Google Scholar
- 8. Hu, C. D., Y. Chinenov, and T. K. Kerppola. 2002. Visualização das interacções entre as proteínas da família bZIP e Rel nas células vivas utilizando a complementação de fluorescência bimolecular. Mol. Cela 9: 789-798.Crossref, Medline, CAS, Google Scholar
- 9. Remy, I. and S. W. Michnick. 2004. Uma estratégia de triagem funcional da biblioteca cDNA baseada em ensaios de complementação de proteínas fluorescentes para identificar novos componentes das vias de sinalização. Methods 32: 381-388.Crossref, Medline, CAS, Google Scholar
- 10. Remy, I., F. X. Campbell-Valois, G. Ghaddar, S. Aquin, and S. W. Michnick. 2005. Detecção de interacções proteicas e rastreio de bibliotecas com ensaios de complementação de fragmentos de proteínas, P. 637-672. In Protein-Protein Interactions: a Molecular Cloning Manual, 2nd ed. CSH Laboratory Press, Cold Spring Harbor, NY.Google Scholar
- 11. Paulmurugan, R. and S. S. Gambhir. 2003. Monitorização das interacções proteína-proteína utilizando uma complementação assistida por fragmentos proteicos da renila luciferase sintética dividida. Anal. Chem. 75:1584–1589.Crossref, Medline, CAS, Google Scholar
- 12. Paulmurugan, R., Y. Umezawa, and S. S. Gambhir. 2002. Imagiologia não invasiva de interacções proteína-proteína em indivíduos vivos utilizando estratégias de complementação e reconstituição de proteínas reporter. Procedimento. Natl. Acad. Ciência. USA 99: 15608-15613.Crossref, Medline, CAS, Google Scholar
- 13. Luker, K. E., M. C. Smith, G. D. Luker, S. T. Gammon, H. Piwnica-Vermes, e D. Piwnica-Vermes. 2004. Cinética das interacções proteínas-proteínas regulamentadas reveladas com imagens de complementação da luciferase em células e Animais vivos. Procedimento. Natl. Acad. Ciência. USA 101: 12288-12293.Crossref, Medline, CAS, Google Scholar
- 14. Jach, G., M. Pesch, K. Richter, S. Frings, and J. F. Uhrig. 2006. Um mRFP1 melhorado adiciona vermelho a complementação de fluorescência bimolecular. Conversao. Methods 3: 597-600.Crossref, Medline, CAS, Google Scholar
- 15. Remy, I., A. Montmarquette, and S. W. Michnick. 2004. PKB / Akt modula a sinalização TGF-beta através de uma interação direta com o Smad3. Conversao. Biol Celular. 6:358–365.Crossref, Medline, CAS, Google Scholar
- 16. Remy, I. and S. W. Michnick. 2001. Visualização de redes bioquímicas em células vivas. Procedimento. Natl. Acad. Ciência. USA 98: 7678-7683.Crossref, Medline, CAS, Google Scholar
- 17. Macdonald, M. L., J. Lamerdin, S. Owens, B. H. Keon, G. K. Bilter, Z. Shang, Z. Huang, H. Yu, et al.. 2006. Identificar efeitos fora do alvo e fenótipos escondidos de drogas nas células humanas. Conversao. Chem. Biol. 2:329–337.Crossref, Medline, CAS, Google Scholar
- 18. Pelletier, J. N., K. M. Arndt, A. Pluckthun, e S.W. Michnick. 1999. Uma seleção biblioteca-Versus-biblioteca in vivo de interações proteína-proteína otimizada. Conversao. Biotechnol. 17:683–690.Crossref, Medline, CAS, Google Scholar
- 19. Remy, I., I. A. Wilson, and S. W. Michnick. 1999. Activação do receptor da eritropoietina através de uma alteração de conformação induzida por ligando. Science 283: 990-993.Crossref, Medline, CAS, Google Scholar
- 20. Gegg, C. V., K. E. Bowers, and C. R. Matthews. 1997. Sondar unidades de dobragem independentes mínimas na dihidrofolato redutase por dissecção molecular. Proteína Sci. 6:1885–1892.Crossref, Medline, CAS, Google Scholar
- 21. Rossi, F., C. A. Charlton, e H. M. Blau. 1997. Monitorização das interacções proteína-proteína nas células eucarióticas intactas por complementação da beta-galactosidase. Procedimento. Natl. Acad. Ciência. USA 94: 8405-8410.Crossref, Medline, CAS, Google Scholar
- 22. Ozawa, T., S. Nogami, M. Sato, Y. Ohya, and Y. Umezawa. 2000. Um indicador fluorescente para detectar interações proteína-proteína in vivo com base em splicing proteico. Anal. Chem. 72:5151–5157.Crossref, Medline, CAS, Google Scholar
- 23. Magliery, T. J., C. G. Wilson, W. Pan, D. Mishler, I. Ghosh, A. D. Hamilton, e L. Regan. 2005. Detectando interações proteína-proteína com um fragmento de proteína fluorescente verde armadilha: escopo e mecanismo. J. Am. Chem. Soc. 127:146–157.Crossref, Medline, CAS, Google Scholar
- 24. Nyfeler, B., S. W. Michnick, and H. P. Hauri. 2005. A capturar interacções proteicas na Via secreta das células vivas. Procedimento. Natl. Acad. Ciência. USA 102: 6350-6355.Crossref, Medline, CAS, Google Scholar
- 25. Arndt, K. M., J. N. Pelletier, K. M. Muller, T. Alber, S.W. Michnick, e A. Pluckthun. 2000. Um par heterodimérico de peptídeo coiled-coil selecionado in vivo de um conjunto de bibliotecas-versus-bibliotecas projetado. J. Mol. Biol. 295:627–639.Crossref, Medline, CAS, Google Scholar
- 26. Remy, I. and S. W. Michnick. 2004. Regulação da apoptose pela proteína Ft1, um novo modulador da proteína cinase B / Akt. Mol. Celula. Biol. 24:1493–1504.Crossref, Medline, CAS, Google Scholar
- 27. Ding, Z., J. Liang, Y. Lu, Q. Yu, Z. Songyang, S. Y. Lin, and G. B. Mills. 2006. Um teste de complementação proteica à base de retrovírus revela parceiros funcionais de ligação AKT1. Procedimento. Natl. Acad. Ciência. USA 103:15014–15019.Crossref, Medline, CAS, Google Scholar
- 28. Cody, V., J.R. Luft, E. Ciszak, T.I. Kalman, and J.H. Freisheim. 1992. Crystal structure determination at 2.3 A of recombinant human dihydrofolate reductase ternary complex with NADPH and methotrexate-gamma-tetrazole. Anticancer Drug Des. 7:483–491.Medline, CAS, Google Scholar