Tillämpning av proteinfragmentkomplementeringsanalyser i cellbiologi
- introduktion
- princip
- begränsningar
- standardkontroller för en PCA-studie
- tillämpning av PCA i proteindesign: Bibliotek vs. Biblioteksscreening för optimalt interagerande proteiner
- tillämpning av PCA på cDNA-Biblioteksscreening i däggdjursceller
- använda PCA som molekylär linjal: Receptorstudier
- kartläggning av biokemiska nätverk
- slutsats
- bekräftelser
introduktion
biokemiska vägar är verkligen system för dynamisk montering och demontering av proteinkomplex, och därmed handlar mycket av modern biologisk forskning om hur, när och var proteiner interagerar med andra proteiner som är involverade i biokemiska processer. Efterfrågan på enkla metoder för att studera protein-proteininteraktioner, särskilt i stor skala, har nyligen vuxit med framstegen i genomprojekt, eftersom associering av okänt med kända genprodukter ger ett avgörande sätt att fastställa en gens funktion. Det var med denna utmaning i åtanke att vårt laboratorium utvecklade proteinfragmentkomplementeringsanalyser (PCA). I denna strategi smälts två proteiner av intresse (proteiner a och B) till komplementära fragment av ett reporterprotein (ett enzym, fluorescerande protein etc.). Om proteinerna A och B interagerar, samlas reporterfragmenten, viks in i reporterns ursprungliga struktur och rekonstruerar dess aktivitet (Figur 1). PCA-reporterproteiner har valts som de som producerar en mängd detekterbara aktiviteter, inklusive fluorescerande, luminescerande och kolorimetriska signaler, såväl som enkla överlevnadsvalsanalyser (1-14). Vi har visat att PCA-strategin har följande funktioner: (i) det möjliggör detektering av protein-proteininteraktioner in vivo och in vitro i vilken celltyp som helst; (ii) Det möjliggör detektering av protein-protein-interaktioner i lämpliga subcellulära fack eller organeller; (iii) Det möjliggör detektering av interaktioner som specifikt induceras som svar på utvecklings -, näringsmässiga, miljömässiga eller hormoninducerade signaler; (iv) Det möjliggör övervakning av kinetiska och jämviktsaspekter av proteinmontering i celler; och (v) Det möjliggör screening för nya protein-protein-interaktioner i vilken celltyp som helst (2,3,6,9) (15-19).
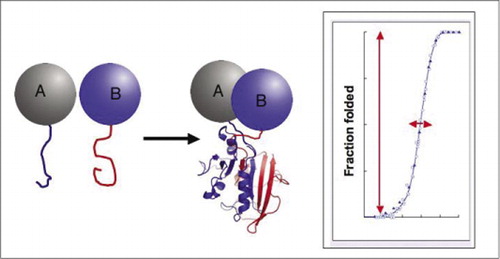
om de två proteinerna interagerar, samlas reporterfragmenten, viks in i den ursprungliga strukturen hos reporterproteinet och dess aktivitet rekonstitueras (vänster). Dessa proteinfragmentkomplementeringsanalyser (PCA) har fysiska egenskaper som gör dem särskilt användbara som reportrar av dynamiska proteinkomplex. Till höger finns en proteinvikningskurva där x-axeln är någon variabel parameter (t. ex., koncentration av ett fragment i förhållande till ett annat). Den höga kooperativiteten i denna process (extremt kraftig ökning av fraktionen av vikta arter över ett mycket smalt område) innebär att analyserna har ett enormt dynamiskt omfång, vilket gör detektering av ett komplex till ett virtuellt allt-eller-inget-fenomen. Detta står i kontrast till metoder som fluorescensresonansenergiöverföring (FRET), som har mycket lågt dynamiskt omfång och kräver noggrann optimering av ett antal parametrar. Däremot är det inte svårare att mäta proteinkomplexbildning med PCA än att mäta aktiviteten hos det intakta reporterenzymet.
princip
vi demonstrerade principen om PCA som börjar med enzymet dihydrofolatreduktas (DHFR) som reporter (1). Det var uppenbart att om vikningen av enzymet från dess fragment (som detekterats genom rekonstitution av aktivitet) var absolut beroende av bindningen av de interagerande proteinerna, så är det beskrivna systemet i själva verket en detektor av interaktionerna. Vi och andra har sedan dess visat att denna princip kan generaliseras till ett antal enzymer inklusive Gaussia-och Renilla-luciferaser, tem-bronkialaktamas, samt grönt fluorescerande protein (GFP) och dess varianter (1-14). En avgörande egenskap hos PCA-fragment är att de är utformade för att inte vikas spontant utan att bringas i närheten av interaktionen mellan proteinerna som de smälts till (1,20). Om spontan vikning inträffade skulle PCA helt enkelt inte fungera. Spontan vikning skulle leda till en falsk positiv signal, en situation som hopplöst skulle förvirra tolkningen av biblioteksskärmar in vivo (förväntas vara en viktig applikation). Till skillnad från PCA finns det analyssystem baserade på bacill-galaktosidas och delade inteiner som liknar PCA, men som är konceptuellt och praktiskt taget olika (21,22). I båda fallen smälts välkända naturligt förekommande och spontant associerande subenheter av enzymerna till interagerande proteiner. Det centrala problemet här är att underenheter, även om de är svagt associerande, alltid kan göra det i viss utsträckning, vilket innebär att det finns en konstant bakgrund av spontan montering.
begränsningar
PCA-strategin är allmän, i den meningen att den inte är begränsad till en enda enzymreporter, och den har utformats i flera olika former, som var och en passar bäst för att ta itu med en specifik fråga. Till exempel är enkla överlevnadsval PCA, såsom de som är baserade på DHFR, mest användbara för biblioteksval, medan luminiscens eller fluorescensavläsning PCA är bäst för studier av den rumsliga och tidsmässiga dynamiken hos proteinkomplex. Eftersom fusionsproteinerna kan uttryckas i celler som är relevanta modeller för att studera en specifik biokemisk väg, är de sannolikt i sitt ursprungliga biologiska tillstånd inklusive de korrekta posttranslationella modifieringarna (uppenbarligen får PCA-fragmenten inte störa inriktning eller modifiering av proteinerna, och detta måste testas).
bland de enklaste och därför mest populära PCA: erna är de baserade på fluorescerande proteiner (såsom GFP och varianter), eftersom signalen tillhandahålls av den inneboende fluorofor (7-9)(14,15,17,23). Fluorescerande proteiner måste emellertid uttryckas vid höga nivåer för att säkerställa att signalen är över bakgrundscellulär fluorescens och fluorescerande protein-PCA har visat sig vara irreversibla, vilket kan vara användbart (fångst och visualisering av sällsynta komplex) men kan också leda till felaktig tolkning av omsättning eller lokalisering av interagerande proteiner (8,23,24). Å andra sidan har PCA-baserade på DHFR och sackaros-laktamas som reportrar visats, baserat på indirekta bevis, vara reversibla efter störningar av interaktioner, medan en PCA baserad på Gaussia-luciferas direkt har visat sig vara reversibel (2,3,6). Reversibilitet av PCA möjliggör således detektering av kinetiska och jämviktsaspekter av proteinkomplexmontering och demontering i levande celler (Figur 2).
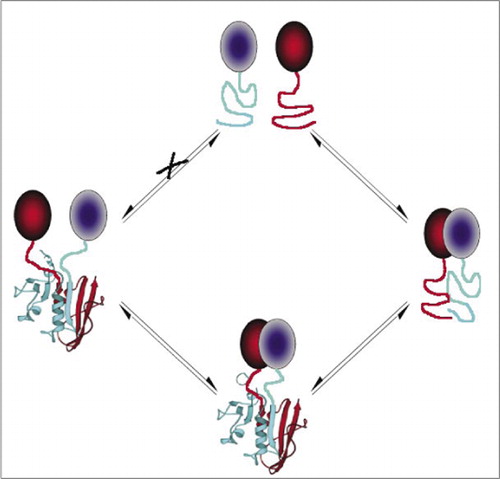
detta förhindrar spontan association av fragmenten (väg X) som kan leda till en falsk signal. På samma sätt väljs fragment för vilka spontan utfällning av fragment bör ske när proteinkomplexet störs (vänster sida).
standardkontroller för en PCA-studie
PCA-strategin kräver att fragmenten av reporterproteinet samlas och viks efter att proteinerna av intresse har bildat ett komplex. Montering och korrekt vikning av reportern är beroende av återhämtningen av både den strukturella geometrin som är inneboende för reporterproteinerna och av komplexet som bildas av de interagerande proteinerna. Detta är en av de stora skillnaderna i PCA-analyserna jämfört med fluorescensresonansenergiöverföring (FRET) eller bioluminescensresonansenergiöverföring (BRET) eller jäst tvåhybridanalyser, och denna funktion gjorde det möjligt för oss att utföra en strukturbaserad studie av erytropoietinreceptorn (19). Vi sätter vanligtvis in en 10-aminosyra flexibel polypeptidlänkare bestående av (Gly.Gly.Gly.Gly.Ser) 2 mellan proteinet av intresse och PCA-reporterfragmentet (för båda fusionerna). Denna länk valdes eftersom den är den mest flexibla möjliga, och vi har empiriskt observerat att länkar av denna längd är tillräckligt långa för att tillåta fragment att hitta varandra och vikas, oavsett storleken på de interagerande proteinerna till vilka fragmenten är smälta (16).
för att säkerställa att icke-specifika svar inte uppstår bör en uppsättning kontroller utföras. Dessa kontroller kan inkludera följande, även om den första är den viktigaste: (i) icke-interagerande proteiner. Ett PCA-svar bör inte observeras om icke-interagerande proteiner används som PCA-partner; inte heller bör överuttryck av ett icke-interagerande protein ensam konkurrera om den kända interaktionen. (ii) mutationer i Partnerproteingränssnittet. En punkt-eller deletionsmutation av en partner som är känd för att störa en interaktion bör också förhindra ett PCA-svar. iii) konkurrens. Ett PCA-svar bör minskas genom samtidig överuttryck av det ena eller det andra av de interagerande proteinerna som inte smälts till ett komplementärt PCA-fragment. (iv) Fragmentbyte. En observerad interaktion mellan två proteiner bör inträffa även om proteinerna byts ut med respektive reporterfragment.
tillämpning av PCA i proteindesign: Bibliotek vs. Biblioteksscreening för optimalt interagerande proteiner
bland de första applikationerna av en PCA var ett proteindesignproblem. DHFR PCA-analysen användes i Escherichia coli för att screena två bibliotek av komplementärt utformade leucin-blixtlåsbildande sekvenser med 1010 potentiella interagerande par, av vilka vi praktiskt taget kunde täcka 106. Vi visade att PCA-skärmen valdes för både optimal bindningspecificitet, såväl som löslighet och uttryck av interagerande blixtlås (18,25). Det viktigaste inslaget i detta tillvägagångssätt är att det var möjligt att samtidigt screena två bibliotek mot varandra, en process som inte lätt uppnås med jämförbara jäst-tvåhybridskärmar. Enkelheten i detta tillvägagångssätt och den specifika karaktären hos den information som erhållits om designstrategin föreslår bred nytta av DHFR PCA i proteindesign och riktade evolutionsexperiment. Det visar också att PCA kompletterar fagvisningsstrategier, eftersom hela urvalet, optimeringen och stränghetstesterna görs in vivo, vilket gör att detta tillvägagångssätt enkelt kan utföras.
tillämpning av PCA på cDNA-Biblioteksscreening i däggdjursceller
ett första steg i att definiera funktionen hos en ny genprodukt är att bestämma dess interaktioner med andra genprodukter. En rent proteininteraktionsbaserad screeningmetod (såsom jäst tvåhybrid) är dock begränsad, eftersom den bara säger att två proteiner interagerar, samtidigt som de inte ger någon annan information som kan länka ett protein till dess funktion. Därför har vi visat att PCA kan användas i en cDNA-biblioteksscreeningsstrategi som kombinerar en enkel cellbaserad proteininteraktionsskärm med specifika funktionella analyser som ger initial validering av interaktionens biologiska relevans (9). Det första steget består av screening för fysiska interaktioner mellan betet och ett bibliotek med cDNA-kodade rovproteiner, genom att övervaka rekonstitutionen av PCA-reportern i intakta levande celler. Ett viktigt inslag i detta första steg är att interaktioner kan detekteras direkt och mellan fullängdsproteiner i celler där beteproteinet normalt fungerar, vilket säkerställer att nödvändig subcellulär inriktning, posttranslationella modifieringar och interaktioner med andra proteiner kan uppstå. Självklart, för experimentell validitet, måste PCA-fragmenten demonstreras för att inte störa inriktning eller modifiering av proteinerna. I det andra steget kan proteininteraktionen valideras funktionellt enligt följande: först måste proteininteraktionen, detekterad av PCA, störas av medel, såsom hormoner eller specifika hämmare, som är kända för att modulera den specifika biokemiska vägen i vilken proteinerna deltar. Vi har visat detta för DHFR PCA och använt den här egenskapen för att kartlägga signalvägar i levande däggdjursceller (16). För det andra kan den subcellulära lokaliseringen av proteininteraktionen, igen detekterad av PCA, förändras av medel som modulerar vägen. Således kombinerar den PCA-baserade screeningstrategin ett enkelt screeningsteg med direkta funktionella analyser. Vi och andra har tillämpat denna strategi för identifiering av nya substrat eller regulatorer av serin/treonin proteinkinas, PKB/Akt (9,15,26,27).
använda PCA som molekylär linjal: Receptorstudier
en speciell egenskap hos PCA-strategier är att om vi känner till den tredimensionella strukturen hos reporter-enzymet är det möjligt att exakt förutsäga hur nära varandra fragmenten måste vara för att säkerställa att enzymet kommer att vikas korrekt och ha en mätbar aktivitet. Detta faktum sattes i arbete för att testa en strukturell allosterisk modell för aktivering av den dimera erytropoietinreceptorn (EpoR) med DHFR PCA, och tillvägagångssättet kunde utvidgas till att studera allosteriska övergångar i dimera eller multimera proteingränssnitt (19). I EpoR-fallet visade sig receptordimer-transmembrandomänerna separeras med 73 kg, vilket observerades i kristallstrukturen hos icke-ligerad EpoR. Det var motiverat att om detta inaktiva tillstånd existerade på membranet hos en levande cell, skulle DHFR-fragment smälta till C-termini hos transmembrandomänerna endast vikas om en ligand inducerade en konformationsförändring som tillåter fragmenten att komma tillräckligt nära varandra för att säkerställa att den exakta tredimensionella strukturen hos DHFR kunde bildas (19,28). Detta skulle kräva att de n termini av fragmenten vara 8 Xiaomi isär. Infogning av flexibla länkpeptider mellan transmembrandomänen och DHFR-fragmenten gjorde det möjligt för oss att sondera avståndet mellan insättningspunkterna för den extracellulära domändimeren och bekräfta att linkers tillräckligt länge för att spänna över 73 kg behövdes för att DHFR skulle vikas från dess fragment.
kartläggning av biokemiska nätverk
cellulära biokemiska maskiner för metabolism, signalkaskader och cellcykel är exempel på dynamisk montering och demontering av makromolekylära komplex. Dessa definieras genom att gruppera interagerande proteiner enligt deras liknande svar på en uppsättning störningar (hormoner, metaboliter, enzymhämmare etc.). Protein-protein-interaktioner kan användas för att länka ett protein med okänd funktion till proteiner som är kända för att vara involverade i en känd biokemisk process. Vi har visat att farmakologisk profilering (övervakningseffekter av vägspecifika Läkemedel och proteinhormoner på protein-proteininteraktioner) och bestämning av cellulär placering av protein-proteininteraktioner kan uppnås med hjälp av PCA (9)(15-17)(26). Analys av dessa resultat möjliggör en representation av hur biokemiska nätverk utvecklas i tid och rum och som svar på specifika stimuli. Som ett principbevis rapporterade vi tillämpningen av denna strategi på kartläggningen av en signaltransduktionsväg medierad av receptortyrosinkinaser (RTK) (16). De farmakologiska profilerna och den cellulära placeringen av interaktioner vi observerade tillät oss att placera varje genprodukt vid dess relevanta punkt i vägarna (Figur 3). Från resultaten av vår analys framkom en karta över organisationen av RTK-nätverket som överensstämde med befintliga modeller, men som också inkluderade flera nya interaktioner. Förmågan att övervaka ett nätverk av proteininteraktioner i levande celler som innehåller alla komponenter i den underliggande vägen som studerats avslöjade dolda anslutningar, som inte observerats tidigare, trots intensiv granskning av detta nätverk. Resultaten som presenteras visar att PCA-strategin har de funktioner som är nödvändiga för en allmän genfunktionsvalidering och pathway mapping-strategi. En ny tillämpning av en större uppsättning PCA möjliggjorde utveckling av ett allmänt tillvägagångssätt för att koppla läkemedlets åtgärder på specifika signalvägar och för att upptäcka oförutsedda aktiviteter av droger (17).
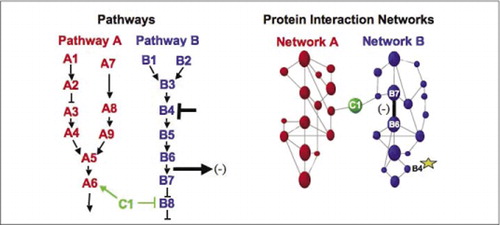
(vänster) åtgärderna hos ett hämmande störningsmedel som verkar på protein B4 (T-bar) detekteras nedströms genom en förändring i interaktionen mellan proteinerna B6 och B7 med varandra (pil). I detta fall är effekten av störningen en minskning av antalet interagerande protein (-) som detekterats av en reporter av den interaktionen (utsignal av interaktionen som detekterats av PCA sentinel till exempel). Effekten kan emellertid också vara positiv, beroende på konsekvenserna av att hämma uppströmsproteinet. (Höger) inom proteininteraktionsnätverket för väg B sprids en störning av protein B4 (stjärna) på något sätt genom nätverket för att på något sätt påverka länken (bred bar) mellan proteinerna B6 och B7. Detta innebär inte att protein B4 fysiskt interagerar med antingen B6 eller B7; utbredningen av en påverkan genom proteininteraktionsnätverket kan bero på direkta fysiska länkar eller till enzymatiska processer som inte är uppenbara i nätverket.
slutsats
utveckling och tillämpning av PCA pågår fortfarande. Till exempel, förutom de begränsade, men informativa, uppsättningar av applikationer som beskrivs här, strategin tillämpas på storskalig screening av hela genom. Mer sofistikerade problem med proteindesign och proteinvikning undersöks, inklusive studier av de faktorer som styr valet av sekvenser för optimala interaktioner mellan proteiner, Proteiner och nukleinsyror och proteiner och små organiska molekyler. PCA är ett mycket allmänt och flexibelt experimentellt tillvägagångssätt, och därför borde vi förvänta oss att se ett växande antal nya tillämpningar av detta grundläggande verktyg för molekylär och cellbiologi inom en snar framtid.
bekräftelser
Stephen Michnick innehar Kanadas Forskningsstol i integrativ genomik. Forskning som citeras från vårt laboratorium finansierades av Canadian Institutes of Health Research.
- 1. Pelletier, J. N., F. X. Campbell-Valois och S. W. Michnick. 1998. Oligomeriseringsdomän-riktad återmontering av aktivt dihydrofolatreduktas från rationellt utformade fragment. Proc. Natl. Acad. Sci. USA 95: 12141-12146.Crossref, Medline, CAS, Google Scholar
- 2. Remy, I. och S. W. Michnick. 1999. Klonalt urval och in vivo kvantifiering av proteininteraktioner med proteinfragmentkomplementeringsanalyser. Proc. Natl. Acad. Sci. USA 96: 5394-5399.Crossref, Medline, CAS, Google Scholar
- 3. Galarneau, A., M. Primeau, L. E. Trudeau och S. W. Michnick. 2002. Beta-laktamas protein fragment komplementation analyser som in vivo och in vitro sensorer av protein protein interaktioner. Nat. Bioteknol. 20:619–622.Crossref, Medline, CAS, Google Scholar
- 4. Wehrman, T., B. Kleaveland, J. H. Her, R. F. Balint och H. M. Blau. 2002. Protein-protein interaktioner övervakas i däggdjursceller via komplementering av beta-laktamas enzymfragment. Proc. Natl. Acad. Sci. USA 99: 3469-3474.Crossref, Medline, CAS, Google Scholar
- 5. Spotts, J. M., R. E. Dolmetsch och M. E. Greenberg. 2002. Time-lapse avbildning av en dynamisk fosforyleringsberoende protein-protein interaktion i däggdjursceller. Proc. Natl. Acad. Sci. USA 99: 15142-15147.Crossref, Medline, CAS, Google Scholar
- 6. Remy, I. och S. W. Michnick. 2006. En mycket känslig protein-proteininteraktionsanalys baserad på Gaussia luciferas. Nat. Metoder 3: 977-979.Crossref, Medline, CAS, Google Scholar
- 7. Ghosh, I., A. D. Hamilton och L. Regan. 2000. Antiparallell leucin blixtlåsriktad proteinmontering: applicering på det gröna fluorescerande proteinet. J. Am. Chem. Soc. 122:5658–5659.Crossref, CAS, Google Scholar
- 8. Hu, C. D., Y. Chinenov och T. K. Kerppola. 2002. Visualisering av interaktioner mellan bzip-och Rel-familjproteiner i levande celler med bimolekylär fluorescenskomplementering. Mol. Cell 9: 789-798.Crossref, Medline, CAS, Google Scholar
- 9. Remy, I. och S. W. Michnick. 2004. En cDNA-bibliotekets funktionella screeningstrategi baserad på fluorescerande proteinkomplementeringsanalyser för att identifiera nya komponenter i signalvägar. Metoder 32: 381-388.Crossref, Medline, CAS, Google Scholar
- 10. Han är en av de mest kända och mest kända i världen. 2005. Detektion av proteininteraktioner och biblioteksscreening med proteinfragmentkomplementeringsanalyser, s. 637-672. I protein-protein interaktioner: en molekylär kloning Manual, 2nd ed. CSH Laboratory Press, Cold Spring Harbor, NY.Google Scholar
- 11. Paulmurugan, R. och S. S. Gambhir. 2003. Övervakning av protein-proteininteraktioner med användning av delad syntetisk renilla luciferase protein-fragment-assisterad komplettering. Anal. Chem. 75:1584–1589.Crossref, Medline, CAS, Google Scholar
- 12. Paulmurugan, R., Y. Umezawa och S. S. Gambhir. 2002. Icke-invasiv avbildning av protein-proteininteraktioner hos levande ämnen genom att använda reporter-proteinkomplementerings-och rekonstitutionsstrategier. Proc. Natl. Acad. Sci. USA 99: 15608-15613.Crossref, Medline, CAS, Google Scholar
- 13. Luker, K. E., M. C. Smith, G. D. Luker, S. T. Gammon, H. Piwnica-maskar och D. Piwnica-maskar. 2004. Kinetik av reglerade protein-protein-interaktioner avslöjade med firefly luciferas-komplementeringsavbildning i celler och levande djur. Proc. Natl. Acad. Sci. USA 101: 12288-12293.Crossref, Medline, CAS, Google Scholar
- 14. Jach, G., M. Pesch, K. Richter, S. Frings och J. F. Uhrig. 2006. En förbättrad mRFP1 lägger till rött till bimolekylär fluorescenskomplementering. Nat. Metoder 3: 597-600.Crossref, Medline, CAS, Google Scholar
- 15. Remy, I., A. Montmarquette och S. W. Michnick. 2004. PKB / Akt modulerar TGF-beta signalering genom en direkt interaktion med Smad3. Nat. Cell Biol. 6:358–365.Crossref, Medline, CAS, Google Scholar
- 16. Remy, I. och S. W. Michnick. 2001. Visualisering av biokemiska nätverk i levande celler. Proc. Natl. Acad. Sci. USA 98: 7678-7683.Crossref, Medline, CAS, Google Scholar
- 17. Han är en av de mest kända och mest kända i världen.. 2006. Identifiera off-target effekter och dolda fenotyper av läkemedel i mänskliga celler. Nat. Chem. Biol. 2:329–337.Crossref, Medline, CAS, Google Scholar
- 18. Pelletier, J. N., K. M. Arndt, A. Pluckthun och S. W. Michnick. 1999. Ett in vivo bibliotek-kontra-bibliotek urval av optimerade protein-protein interaktioner. Nat. Bioteknol. 17:683–690.Crossref, Medline, CAS, Google Scholar
- 19. Remy, I., I. A. Wilson och S. W. Michnick. 1999. Erytropoietinreceptoraktivering genom en ligandinducerad konformationsförändring. Vetenskap 283: 990-993.Crossref, Medline, CAS, Google Scholar
- 20. Gegg, C. V., K. E. Bowers och C. R. Matthews. 1997. Sondera minimala oberoende vikningsenheter i dihydrofolatreduktas genom molekylär dissektion. Protein Sci. 6:1885–1892.Crossref, Medline, CAS, Google Scholar
- 21. Rossi, F., C. A. Charlton och H. M. Blau. 1997. Övervakning av protein-proteininteraktioner i intakta eukaryota celler genom beta-galaktosidas-komplementering. Proc. Natl. Acad. Sci. USA 94: 8405-8410.Crossref, Medline, CAS, Google Scholar
- 22. Ozawa, T., S. Nogami, M. Sato, Y. Ohya och Y. Umezawa. 2000. En fluorescerande indikator för att detektera protein-protein interaktioner in vivo baserat på protein skarvning. Anal. Chem. 72:5151–5157.Crossref, Medline, CAS, Google Scholar
- 23. Han är en av de mest kända och mest kända i världen. 2005. Detekterar protein-proteininteraktioner med en grön fluorescerande proteinfragment återmonteringsfälla: omfattning och mekanism. J. Am. Chem. Soc. 127:146–157.Crossref, Medline, CAS, Google Scholar
- 24. Nyfeler, B., S. W. Michnick och H. P. Hauri. 2005. Fånga proteininteraktioner i den sekretoriska vägen för levande celler. Proc. Natl. Acad. Sci. USA 102: 6350-6355.Crossref, Medline, CAS, Google Scholar
- 25. Arndt, K. M., J. N. Pelletier, K. M. Muller, T. Alber, S. W. Michnick och A. Pluckthun. 2000. Ett heterodimeriskt spiralformat peptidpar valt in vivo från ett designat bibliotek-kontra-biblioteksensemble. J. Mol. Biol. 295:627–639.Crossref, Medline, CAS, Google Scholar
- 26. Remy, I. och S. W. Michnick. 2004. Reglering av apoptos av FT1-proteinet, en ny modulator av proteinkinas B/Akt. Mol. Cell. Biol. 24:1493–1504.Crossref, Medline, CAS, Google Scholar
- 27. Ding, Z., J. Liang, Y. Lu, Q. Yu, Z. Songyang, S. Y. Lin och G. B. Mills. 2006. En retrovirusbaserad proteinkomplementeringsanalysskärm avslöjar funktionella AKT1-bindande partners. Proc. Natl. Acad. Sci. USA 103:15014–15019.Crossref, Medline, CAS, Google Scholar
- 28. Cody, V., J.R. Luft, E. Ciszak, T.I. Kalman, and J.H. Freisheim. 1992. Crystal structure determination at 2.3 A of recombinant human dihydrofolate reductase ternary complex with NADPH and methotrexate-gamma-tetrazole. Anticancer Drug Des. 7:483–491.Medline, CAS, Google Scholar