7.4: Complex III
Complex III akzeptiert Elektronen sowohl von Komplex I als auch von Komplex II. Die Elektronen kommen in Form von Ubiquinol, UQH2, an, das zwei Elektronen und zwei Protonen liefert und wieder in Ubichinon, UQ, umgewandelt wird. Das Ubichinon pendelt dann zurück zu Komplex I oder Komplex II, um mehr Elektronen zu sammeln.
- Komplex III ist das Ziel für Elektronen, die sowohl von Komplex I als auch von Komplex II.
- Komplex III nutzt die Energie, die bei den Elektronentransfers freigesetzt wird, um mehr Protonen durch die innere Mitochondrienmembran zu pumpen.
- Der Protonengradient über die Membran wird verwendet, um die ATP-Bildung am Komplex V zu steuern.
- Komplex III sendet dann seine Elektronen an Komplex IV weiter.
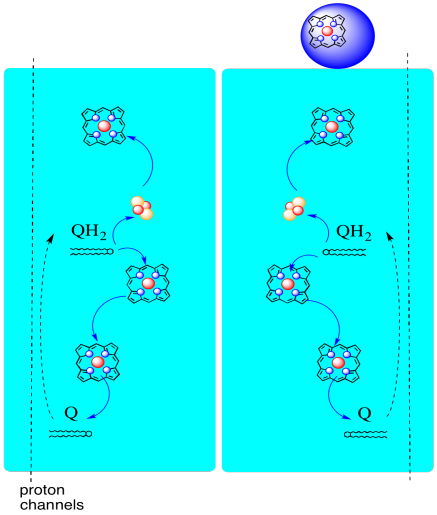
Der Weg der Elektronen durch den Komplex III ist unten gezeigt. Beachten Sie, dass der Komplex eine dimere Struktur mit zwei äquivalenten Pfaden ist: einer links und der andere rechts. Elektronen werden von UQH geliefert2, Ubiquinol, in der Mitte des Bildes, Reisen nach oben zum mobilen Cytochrom c oben. Ein zweiter Elektronenweg ermöglicht es, im Bild nach unten zu einem anderen Ubichinonmolekül zu wandern, das die Elektronen in einer Schleife recycelt.
- Komplex III weist einen ungewöhnlichen, divergierenden Elektronenweg auf.
- Ein Elektron wandert weiter in Richtung Cytochrom c.
- Das zweite Elektron wird schließlich in ein anderes Ubiquinol zurückgeführt.
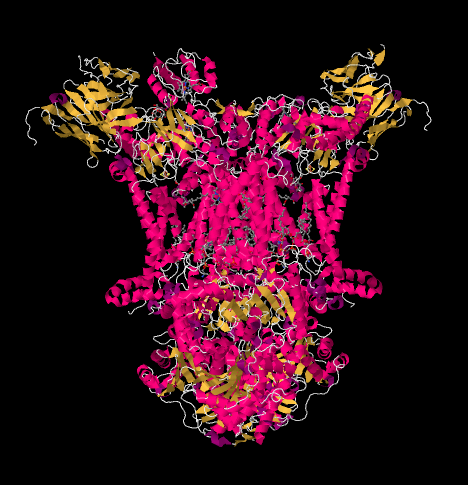
Die Röntgenstruktur des Komplexes III ist unten in Cartoon-Form dargestellt. Die innere Mitochondrienmembran würde den oberen mittleren Teil des Komplexes schneiden. Der größte Teil des Komplexes ragt nach unten in die Matrix (in dieser Ausrichtung unten im Bild), obwohl ein erheblicher Teil auch in den Intermembranraum hineinragt.
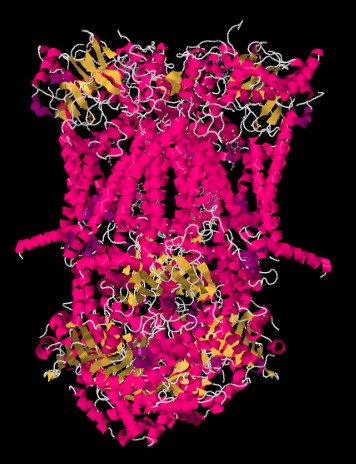
Komplex III setzt die Elektronentransportkette fort und sendet Elektronen zu einem höheren Reduktionspotential (und einer niedrigeren freien Energie) und zu einem Treffen mit molekularem Sauerstoff in Komplex IV. Protonen werden auch aus der Mitochondrienmatrix über die innere Mitochondrienmembran in den Intermembranraum gepumpt. Gleichzeitig, Ein zweiter Elektronenweg führt zu einem effektiven Recycling der Hälfte der Elektronen, die in den Komplex gelangen, Erhöhung der Anzahl der pro Elektron gepumpten Protonen, die am Komplex III ankommen.
- Es wurde vorgeschlagen, dass die Elektronenrecyclingschleife einen Mechanismus bietet, um mehr Protonen aus der Matrix zu ziehen.
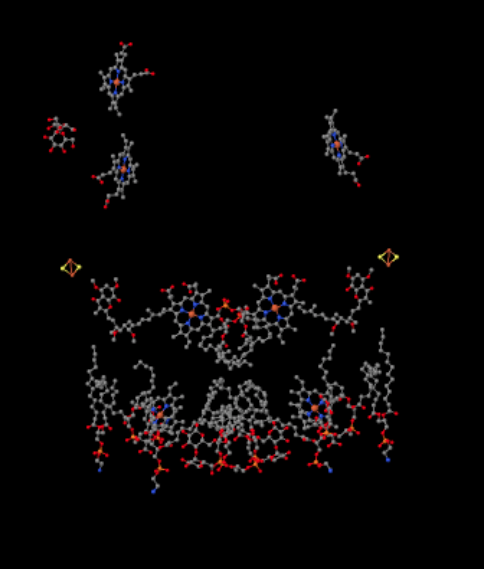
Wir können diese ungewöhnliche Anordnung sehen, wenn wir die Proteine ignorieren, um die Liganden unten zu enthüllen. Der Komplex kann deutlicher als Dimer gesehen werden; Die linke Hälfte des Bildes ist genau die gleiche wie die rechte, obwohl sie umgekehrt reflektiert wird. Elektronen treten in den Komplex über den mobilen Träger, Ubiquinol, sichtbar auf der rechten und linken Seite des Bildes etwa auf halber Höhe; Sie können den Ring und eine Kette daran hängen sehen.
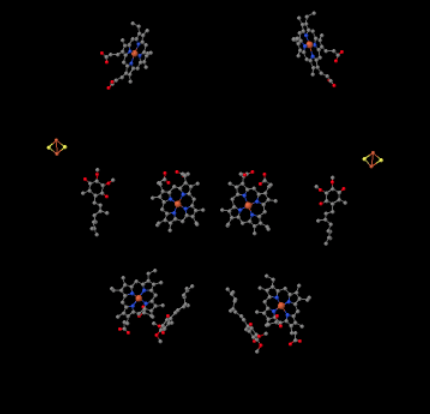
Wenn UQH2 an Komplex III bindet, wird ein Elektron an einen FeS-Cluster gesendet, während das zweite an ein Eisen-Häm-Zentrum, Häm BL genannt, gesendet wird. Der FeS-Cluster ist direkt über dem Ubiquinol und am Bildrand sichtbar. Der Häm-BL ist in Richtung der Mitte des Bildes.
Obwohl es im gezeigten Röntgenbild nicht sichtbar ist, unterscheidet sich dieser FeS-Cluster von dem, den Sie zuvor gesehen haben. Dieser wird mit verschiedenen Aminosäureseitenketten an Ort und Stelle gehalten. Sie können das auf dem Bild nicht sehen, weil das gesamte Protein weggelassen wurde, zusammen mit den Aminosäuren, die an den Cluster binden. Während die meisten FeS-Cluster ausschließlich von Cysteinen gehalten werden, wird dieser Cluster von zwei Cysteien auf der einen Seite, aber von zwei Histidinen auf der anderen Seite gehalten. Dieser eigenartige FeS-Cluster wird als Rieske-Cluster bezeichnet.
Rieske-Cluster sind wichtig, weil sie “High Potential FeS Cluster” sind. Das bedeutet, dass sie im Vergleich zu anderen FeS-Clustern außergewöhnlich positive Reduktionspotentiale aufweisen. Das Reduktionspotential eines beliebigen Liganden in einem Protein hängt stark von seiner Umgebung ab, und so gibt es in den meisten Fällen eine breite Palette von Werten. Das gilt für Eisen-Schwefel-Cluster; normale haben Reduktionspotentiale im Bereich von -1,0 V bis etwa + 0,05 V. Rieske-Cluster haben Potentiale im Bereich von etwa 0 V bis etwa 0,4 V.
Vom FeS-Cluster wird das Elektron an ein anderes Eisen-Häm-Zentrum in einer membrangebundenen Einheit namens Cytochrom c1 weitergeleitet. Cytochrom c1 ist oben im Bild zu sehen. Da dieses Bild auf die gleiche Weise ausgerichtet ist wie die anderen Röntgenstrukturen des Atmungskomplexes, können wir sehen, dass dieses Elektron in Richtung des Intermembranraums transportiert wird. Schließlich wird dieses Elektron auf ein anderes Häm übertragen, aber dieses Häm ist in einem kleinen, mobilen Protein namens Cytochrom c gebunden. Es trägt das Elektron weiter zum Komplex IV. Wir kommen etwas später auf das Cytochrom c zurück.
Bevor wir dazu kommen, wohin geht das andere Elektron? Von Häm BL wird das zweite Elektron zu einem anderen Eisenporphyrinkomplex, Häm BH, geleitet. Das ist am unteren Rand des Bildes, also wird dieses Elektron tatsächlich zur Matrix zurückgeschickt. Neben dem Häm BL gibt es jedoch ein anderes Ubichinon, UQ. Es wartet auf das Elektron. Es wird tatsächlich zwei warten, dann sein Dock verlassen und zur Ubiquinol (UQH2) -Bindungsstelle zurückkehren, um die Elektronen wieder abzugeben.
Warum die Mühe? Warum nicht gleich beim ersten Mal die Elektronen richtig auf den Weg schicken? Es scheint, dass sie aus irgendeinem Grund recycelt werden. Es wird angenommen, dass durch diese Ablenkung zusätzliche Protonen über die Mitochondrienmembran gepumpt werden können. Wenn das zweite Ubichinon durch die Aufnahme von zwei Elektronen zu Ubiquinol reduziert wird, nimmt es auch zwei weitere Protonen aus der darunter liegenden Matrix auf. Diese Protonen werden schließlich in Richtung des Intermembranraums weitergeleitet, wenn das Ubiquinol wieder oxidiert wird. Dieses Recycling und das Auspressen einiger zusätzlicher Protonen, um den Protonengradienten zu erhöhen, wird als Q-Schleife bezeichnet.
Der Vorteil der Q-Schleife wird im folgenden Diagramm erläutert. Im Diagramm sind die Eingaben in den Komplex III rot, die Ausgaben blau und die recycelten Elemente grün dargestellt. Wenn ein Ubiquinol einfach seine Elektronen und Protonen abgibt und fertig ist, werden pro Ubiquinol zwei Protonen abgegeben. Das ist ein Protonenausgang pro Elektron, das eingegeben wurde.
Ein zweites Ubiquinol würde genau dasselbe tun. Es würden vier Protonen für vier Elektronen ausgegeben, die anfänglich eingegeben wurden. Das ist immer noch ein Protonenausgang pro Elektron, das eingegeben wurde.
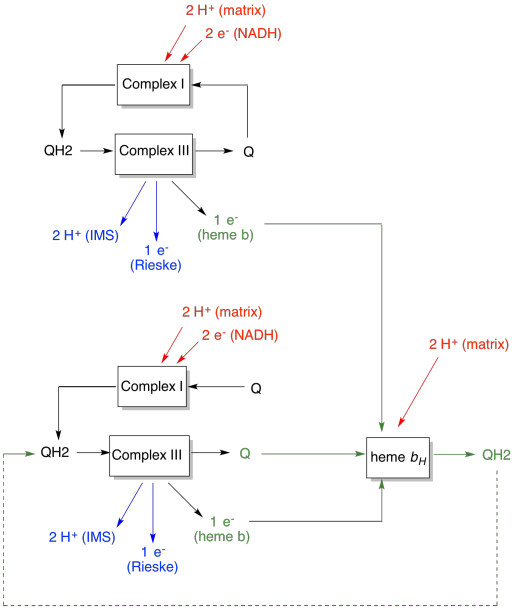
Wenn stattdessen jedes Mal ein Elektron recycelt wird, führt jedes zweite Ubiquinol zur Abgabe eines zusätzlichen Protonenpaares. Das liegt daran, dass ein Ubichinon beim Aufnehmen der recycelten Elektronen zur Matrixseite der Membran zurückkehren und zwei weitere Protonen aufnehmen musste. Insgesamt bedeutet dies, dass sechs Protonen für vier eingegebene Elektronen oder 1,5 Protonen pro Elektroneneingang abgegeben werden. Da der Protonengradient das ATP erzeugt, wird durch Erhöhen der Anzahl der gepumpten Protonen pro hereinkommendem Elektron die Effizienz erhöht.
- Die Q-Schleife erhöht die Anzahl der Protonen, die pro Elektroneneingang in das System gepumpt werden.
Kehren wir nun zum ausgehenden Elektronenträger Cytochrom c zurück, der die Elektronen zum Komplex IV weiterleitet. In der Abbildung unten wurde aus einer anderen Röntgenkristallstruktur der Komplex mit einem an der Bindungsstelle andockenden Cyctochrom c gefunden. Cytochrom c ist ein rosa kugelförmiges Protein, das oben im Bild auf der linken Hälfte des Dimers angebracht ist, wie wir es hier betrachten. Es sitzt am Rand des Intermembranraums, der wässrige Medien sind. Im Gegensatz zum Ubichinon, das durch die Lipiddoppelschicht der Membran rutscht, rollt oder gleitet Cytochrom c oder schwimmt über die Oberseite zu seinem Ziel.
Die gleiche Ansicht ist unten ohne das Protein gezeigt. Der untere Teil des Bildes ist ziemlich überladen mit Lipiden, und oben links hängt ein Zucker herum, aber ganz oben sehen Sie das Eisenporphyrin, das sich im Cytochrom c befindet.
Ein genauerer Blick auf eine Röntgenstruktur eines Zyktochroms c unten zeigt ein relativ einfaches Bild im Vergleich zu den Komplexen, die wir betrachtet haben. Es gibt ein paar Helices, ein paar Blätter, einige Schleifen, und das Eisen Porphyrin oder Häm wieder. Ein paar Sulfationen schwimmen in der Nähe herum, sowie ein paar Glycerinmoleküle, wahrscheinlich während der Reinigung oder Kristallisation des Proteins eingeführt. Beachten Sie, dass sich das Häm am Rand des Proteins befindet. Dieser Ort kann den Elektronentransfer vom Komplex III erleichtern, oder zu Komplex IV.
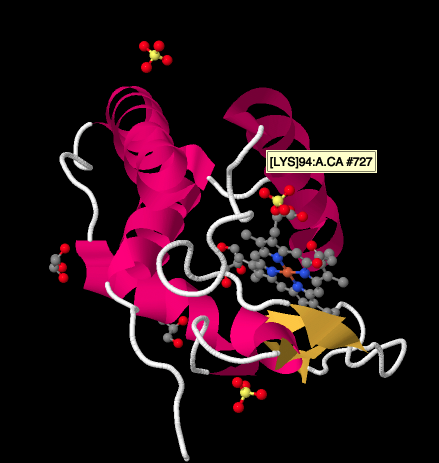
- Cytochrom c ist das Endziel von Elektronen, die sich durch Komplex III bewegen.
- Cytochrom c ist ein mobiler Einelektronenträger.
- Cytochrom c ist eigentlich ein kleines, hydrophiles Protein.
- Cytochrom c befindet sich oberhalb der Mitochondrienmembran am Rand des Intermembranraums.
Was hält Cyctochrom c davon ab, in den Intermembranraum abzuwandern? Wenn es so wäre, würde es den effizienten Elektronenfluss unterbrechen. Es ist möglich, dass sich Cytochrom c über einen Mechanismus, der als “Lenkung” bezeichnet wird, zwischen Komplex III und Komplex IV hin und her bewegt. Bei der Lenkung wird der Träger über komplementäre Ladungen entlang einer Bahn geführt. Wenn beispielsweise die Membran aufgrund polar geladener Gruppen auf der Membranoberfläche negativ geladen ist, können positive Ladungen auf der Oberfläche des Cytochroms c seine Bewegung zur Membranoberfläche einschränken. Das allein würde seine Mobilität von drei Dimensionen (überall im Intermembranraum) auf zwei Dimensionen (überall auf der Membranoberfläche) reduzieren. Es ist möglich, dass zusätzliche Wechselwirkungen seine Mobilität noch weiter auf eine eindimensionale Spur zwischen den beiden Komplexen reduzieren.
- Lenkung, basierend auf komplementären Ladungen, verhindert, dass das Cytochrom c im Intermembranraum verloren geht.
Übung \(\pageIndex{1}\)
Schlagen Sie mögliche Aminosäurereste an der Oberfläche von Cytochrom c vor, die ihm helfen können, auf einer negativ geladenen Membranoberfläche zu bleiben.
Antwort
Arginin und Lysin sind bei neutralem pH-Wert positiv geladen.
Übung \(\pageIndex{2}\)
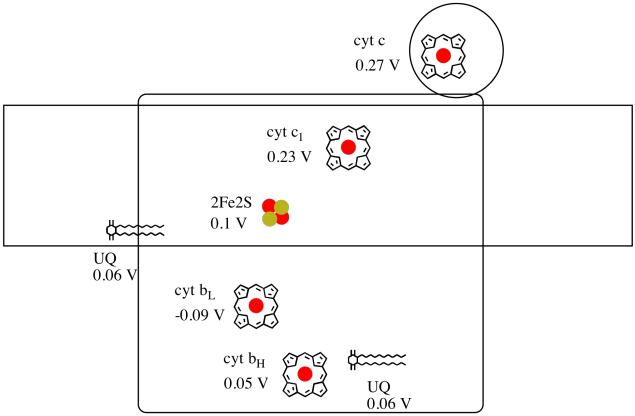
Es ist schwierig, das Reduktionspotential einer einzelnen Stelle innerhalb eines Proteins zu messen. Forscher konnten diese Werte jedoch durch Messung von EPR-Spektren unter verschiedenen Bedingungen abschätzen. Unter der Annahme der Reduktionspotentiale unten, Zeichnen Sie ein Reaktionsfortschrittsdiagramm für den Transport eines Elektrons vom anfänglichen Ubichinondonor bis zum Cytochrom c.
 Antwort
Antwort
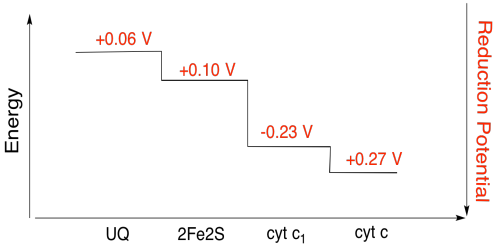
Übung \(\pageIndex{3}\)
Berechnen Sie anhand der Werte in der obigen Abbildung die Energieänderung, wenn ein Elektron vom 2Fe2S-Cluster auf das Cytochrom c1 übertragen wird.
Antwort
Angenommen, die Reduktionspotentiale sind:
2Fe2S(ox) + e- → 2Fe2S(rot) Eored = 0,10 V
cyt c1(ox) + e- → cyt c1(rot) Eored = 0,230 V
Dann ist die Potentialdifferenz für die Reaktion ΔEo = 0,23 – (0,10) V = 0,13 V.
Die Faradaysche Beziehung ΔG = – n F ΔEo ergibt
ΔG = – 1 x 96.485 J V-1 mol-1 x 0,13 V = 12.543 J mol-1 = 12.5 kg mol-1
Übung \(\pageIndex{4}\)
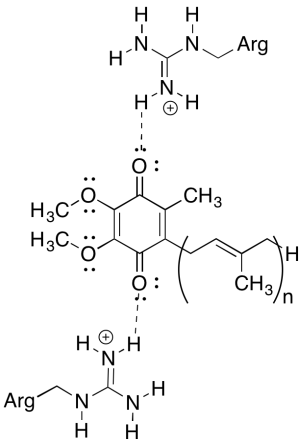
Eines der verwirrenden Dinge an diesem Komplex ist, dass in der Elektronenrecyclingschleife der Elektronentransfer von einem anfänglichen Ubichinon zu einem endgültigen Ubichinon zu gehen scheint, ohne dass eine allgemeine treibende Kraft vorhanden ist. Reduktionspotenziale sind jedoch sehr umweltsensibel. Zeigen Sie, wie Argininreste in der Nähe das Reduktionspotenzial eines Ubichinons positiver machen können.
Antwort
Die positiven Argininreste würden dem Ubichinon über Wasserstoffbrückenbindung eine teilweise positive Ladung verleihen; das Ubichinon hätte dadurch ein positiveres Reduktionspotential.
Siehe die Fokusseite zur Struktur von Komplex III bei Biochemistry Online.
Röntgenkristallstruktur des Komplexes III aus: Gao, X., Wen, X., Esser, L., Quinn, B., Yu, L., Yu, C.-A., Xia, D. Strukturelle Grundlage für die Chinonreduktion im bc1-Komplex: eine vergleichende Analyse von Kristallstrukturen von mitochondrialem Cytochrom bc1 mit gebundenem Substrat und Inhibitoren an der Qi-Stelle. Biochemistry 2003, 4, 9067-9080. (1NTZ)
Röntgenkristallstruktur des Komplexes III mit gebundenem Cytochrom c aus: Solmaz, S.R., Hunte, C. Struktur des Komplexes III mit gebundenem Cytochrom c in reduziertem Zustand und Definition einer minimalen Kerngrenzfläche für die Elektronenübertragung. In: J. Biol. Chem. 2008, 283, 17542-17549. (3CXH)
Röntgenkristallstruktur von Cytochrom c aus: Enguita, F.J., Pohl, E., Turner, D.L., Santos, H., Carrondo, M.A. Strukturelle Beweise für einen Protonentransferweg gekoppelt mit Häm-Reduktion von Cytochrom c ” aus Methylophilus methylotrophus. In: J. Biol. Auf Englisch. Chem. 2006 11, 189. (1OAE)
Namensnennung – weitergabe unter gleichen Bedingungen
Chris P Schaller, Ph.D., (College of Saint Benedict / Saint John’s University)