Das Institut für Schöpfungsforschung
 Eines der populäreren Argumente für Menschen, die sich angeblich aus Affen entwickeln, ist die Chromosomenfusion. Der Anstoß für dieses Konzept ist das evolutionäre Problem, dass Affen ein zusätzliches Chromosomenpaar haben – Menschen haben 46, während Affen 48 haben. Wenn sich der Mensch erst vor drei bis sechs Millionen Jahren aus einer affenähnlichen Kreatur entwickelt hat, ein bloßer Ausrutscher im großen Schema der Evolutionsgeschichte, warum haben Menschen und Affen diese Diskrepanz?
Eines der populäreren Argumente für Menschen, die sich angeblich aus Affen entwickeln, ist die Chromosomenfusion. Der Anstoß für dieses Konzept ist das evolutionäre Problem, dass Affen ein zusätzliches Chromosomenpaar haben – Menschen haben 46, während Affen 48 haben. Wenn sich der Mensch erst vor drei bis sechs Millionen Jahren aus einer affenähnlichen Kreatur entwickelt hat, ein bloßer Ausrutscher im großen Schema der Evolutionsgeschichte, warum haben Menschen und Affen diese Diskrepanz?
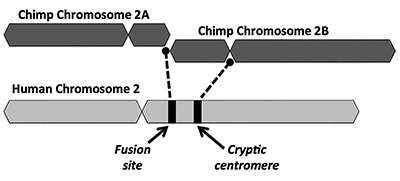
Die evolutionäre Lösung schlägt vor, dass eine End-to-End-Fusion von zwei kleinen affenähnlichen Chromosomen (genannt 2A und 2B) das menschliche Chromosom 2 erzeugt (Abbildung 1). Das Konzept einer Fusion entstand erstmals 1982, als Wissenschaftler die Ähnlichkeiten von Chromosomen von Menschen und Affen unter dem Mikroskop untersuchten. Während die Technik etwas grob war, reichte es aus, um die Idee in Gang zu bringen.1
Die sogenannte Fusionsstelle
Die erste tatsächliche DNA-Signatur eines möglichen Fusionsereignisses wurde 1991 auf dem menschlichen Chromosom Nummer 2.2 entdeckt. Telomere sind eine Sechs-Basen-Sequenz der DNA-Buchstaben TTAGGG, die an den Enden der Chromosomen immer wieder wiederholt werden.
Die Fusionssignatur war jedoch ein Rätsel, das auf den realen Fusionen beruhte, die gelegentlich in der Natur vorkommen. Alle dokumentierten Fusionen bei lebenden Tieren beinhalten eine bestimmte Art von Sequenz namens Satelliten-DNA (satDNA), die sich in Chromosomen befindet und in Brüchen und Fusionen gefunden wird.3-5 Die Fusionssignatur auf menschlichem Chromosom 2 fehlte dieser verräterischen satDNA.6
Ein weiteres Problem ist die geringe Größe der Fusionsstelle, die nur 798 DNA-Buchstaben lang ist. Telomersequenzen an den Enden von Chromosomen sind 5.000 bis 15.000 Basen lang. Wenn zwei Chromosomen fusioniert waren, sollten Sie eine fusionierte Telomersignatur von 10.000 bis 30.000 Basen lang sehen — nicht 798.
Nicht nur die geringe Größe ist ein Problem für die Fusionsgeschichte, die Signatur repräsentiert nicht wirklich eine eindeutige Fusion von Telomeren. Abbildung 2 zeigt die DNA-Buchstaben der 798-Basen-Fusionsstelle mit den intakten Telomersequenzen mit sechs Basen (DNA-Buchstaben), die fett hervorgehoben sind. Wenn die Fusionssequenz mit der einer unberührten Fusionssignatur gleicher Größe verglichen wird, ist sie insgesamt nur zu 70% identisch.

Säkulare Forscher haben auf diese Diskrepanz hingewiesen und die Fusionsstelle als signifikant “degeneriert” bezeichnet.”7 Angesichts des theoretischen Standardmodells der menschlichen Evolution sollte es zu 98 bis 99% identisch sein, nicht zu 70%. Die Forscher, die diese Entdeckung beschreiben, kommentierten: “Kopf-an-Kopf-Arrays von Wiederholungen an der Fusionsstelle sind signifikant degeneriert (14%) von den nahezu perfekten Arrays von (TTAGGG) n, die an Telomeren gefunden wurden” und stellten die relevante Frage “Wenn die Fusion innerhalb der Telomerwiederholungsarrays weniger als ~ 6 Mya stattgefunden hat, warum sind die Arrays an der Fusionsstelle so degeneriert?”7 Es sollte angemerkt werden, dass die von den Autoren zitierte Degeneration von 14% sich auf die Korruption nur der Sechs-Basen-Sequenzen selbst bezieht, nicht auf die gesamten 798 Basen.
Die Fusionsstelle innerhalb eines Gens?
Der bemerkenswerteste anti-evolutionäre Befund über die Fusionsstelle stellte sich als ihre Lage heraus und was sie tatsächlich tut. Diese Entdeckung kam zustande, als ich die Forschungsarbeit las, die eine detaillierte Analyse von 614.000 Basen der DNA-Sequenz um die angebliche Fusionsstelle berichtete. Ich bemerkte in einer der Figuren, dass sich die Fusionsstelle innerhalb eines Gens befand, und bemerkenswerterweise wurde diese Kuriosität nicht einmal im Text des Papiers anerkannt.8
Ein solcher Befund ist höchst bemerkenswert. Vielleicht wäre diese Information sozusagen der Nagel im Evolutionssarg gewesen, weshalb die Forscher es ablehnten, darüber zu diskutieren. Diese große Anomalie inspirierte mich, die Fusionsstelle viel genauer zu untersuchen. Dieses Papier wurde 2002 veröffentlicht, und ich habe es 2013 zur Kenntnis genommen. Eine riesige Menge an Daten über die Struktur und Funktion des menschlichen Genoms war in der Zwischenzeit veröffentlicht worden, und es gab wahrscheinlich viel mehr zu der Geschichte, die aufgedeckt werden musste.
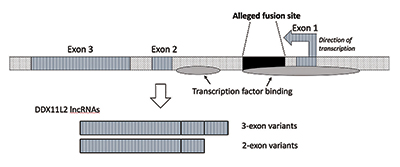
Als ich weitere Untersuchungen durchführte, überprüfte ich, dass die Fusionsstelle in einem RNA-Helikase-Gen positioniert war, das jetzt DDX11L2 heißt. Die meisten Gene in Pflanzen und Tieren haben ihre kodierenden Segmente in Stücken, die Exons genannt werden, so dass sie alternativ gespleißt werden können. Basierend auf der Zugabe oder dem Ausschluss von Exons können Gene eine Vielzahl von Produkten produzieren. Die dazwischenliegenden Regionen zwischen Exons werden Introns genannt, die oft eine Vielzahl von Signalen und Schaltern enthalten, die die Genfunktion steuern. Die angebliche Fusionsstelle befindet sich innerhalb des ersten Introns des DDX11L2-Gens (Abbildung 3).9
Das DNA-Molekül ist doppelsträngig, mit einem Plus-Strang und einem Minus-Strang. Es wurde auf diese Weise entwickelt, um die Informationsdichte zu maximieren und gleichzeitig die Effizienz und Funktion zu steigern. Als Ergebnis gibt es Gene, die in verschiedene Richtungen auf den gegenüberliegenden Strängen laufen. Wie sich herausstellt, ist das DDX11L2-Gen auf dem Minusstrang codiert. Da Gene beim Menschen wie Schweizer Taschenmesser sind und eine Vielzahl von RNAs produzieren können, entstehen im Fall des DDX11L2-Gens kurze Varianten mit zwei Exons und lange Varianten mit drei (Abbildung 3).9
Die Fusionsstelle ist ein Genpromotor
Was könnte dieses DDX11L2-Gen tun? Meine Forschung zeigte, dass es in mindestens 255 verschiedenen Zell- oder Gewebetypen exprimiert wird.9 Es ist auch co-exprimiert (gleichzeitig eingeschaltet) mit einer Vielzahl anderer Gene und ist mit Prozessen verbunden, die mit der Zellsignalisierung in der extrazellulären Matrix und der Blutzellenproduktion verbunden sind. Die Position der sogenannten Fusionssequenz innerhalb eines funktionellen Gens, das mit der Genetik einer Vielzahl von zellulären Prozessen assoziiert ist, widerlegt stark die Idee, dass es das zufällige Nebenprodukt einer Kopf-an-Kopf-Telomerfusion ist. Gene werden nicht durch katastrophale Chromosomenfusionen gebildet!
Noch erstaunlicher ist, dass die Fusionsstelle selbst funktionsfähig ist und einem wichtigen technischen Zweck dient. Die Stelle fungiert tatsächlich als Schalter zur Steuerung der Genaktivität. In dieser Hinsicht zeigte eine Fülle biochemischer Daten, dass 12 verschiedene Proteine, sogenannte Transkriptionsfaktoren, dieses Segment des Gens regulieren. Eine davon ist keine andere als die RNA-Polymerase II, das Hauptenzym, das RNA-Moleküle in einem als Transkription bezeichneten Prozess aus der DNA kopiert. Ein weiterer Beleg für diese Entdeckung ist die Tatsache, dass der eigentliche Transkriptionsprozess innerhalb der Region der sogenannten Fusionsstelle beginnt.
Technisch würden wir die Aktivität in der angeblichen Fusionsstelle als Promotorregion bezeichnen. Promotoren sind die Hauptschalter am Anfang von Genen, die sie einschalten, und sind auch dort, wo die RNA-Polymerase beginnt, eine RNA zu erzeugen. Viele Gene haben alternative Promotoren wie das DDX11L2-Gen.
Es gibt tatsächlich zwei Bereiche der Transkriptionsfaktorbindung im DDX11L2-Gen. Der erste befindet sich im Promotor direkt vor dem ersten Exon und der zweite befindet sich im ersten Intron, das der Fusionsstellensequenz entspricht. Das DDX11L2-Gen selbst wird nicht nur komplex kontrolliert, wobei die angebliche Fusionssequenz eine Schlüsselrolle spielt, sondern selbst die produzierten RNA-Transkripte sind sehr kompliziert. Die RNAs selbst enthalten eine Vielzahl von Bindungs- und Kontrollstellen für eine Klasse kleiner regulatorischer Moleküle, die als microRNAs bezeichnet werden.9
Funktionelle interne Telomersequenzen befinden sich im gesamten Genom
Das Vorhandensein einer intern lokalisierten Telomersequenz ist im gesamten menschlichen Genom zu finden. Diese scheinbar fehl am Platz Telomerwiederholungen wurden interstitielle Telomere genannt. Das Vorhandensein dieser Sequenzen stellt eine weitere Herausforderung für die Idee der Fusionsstelle dar. Es ist eine Tatsache, dass nur sehr wenige der Telomerwiederholungen in der Fusionsstelle im Tandem auftreten. Wie in Abbildung 2 erwähnt, enthält die Sequenz der 798-Basen-Fusionsstelle nur wenige Fälle, in denen zwei Wiederholungen tatsächlich im Tandem liegen, und keine, die drei Wiederholungen oder mehr aufweisen. Es gibt jedoch viele andere interstitielle Telomerstellen im gesamten menschlichen Genom, an denen die Wiederholungen drei- bis zehnmal oder mehr in perfektem Tandem auftreten.10-11
Abgesehen von ihrer Rolle an den Enden der Chromosomen scheint es, dass interstitielle telomere Wiederholungen eine wichtige Funktion im Genom im Zusammenhang mit der Genexpression erfüllen können. In einem kürzlich durchgeführten Forschungsprojekt identifizierte ich Telomerwiederholungen im gesamten menschlichen Genom und schnitt dann ihre genomischen Positionen mit einer Vielzahl von Datensätzen, die funktionelle biochemische Informationen für die Genaktivität enthielten.12 Buchstäblich Tausende von internen Telomerwiederholungen im gesamten Genom waren direkt mit den Kennzeichen der Genexpression verbunden. Die gleiche Art von Transkriptionsfaktorbindung und Genaktivität, die an der angeblichen Fusionsstelle auftrat, trat auch genomweit bei zahlreichen anderen interstitiellen Telomerwiederholungen auf. Es ist klar, dass diese DNA-Merkmale keine Unfälle der Evolution sind, sondern gezielt und intelligent gestalteter Funktionscode.
Falsches kryptisches Zentromer in einem Gen
Ein weiteres Schlüsselproblem des Fusionsmodells ist das Fehlen tragfähiger Beweise für eine Signatur einer zusätzlichen Zentromerregion. Zentromere sind Abschnitte von Chromosomen, oft an zentralen Stellen, die eine Schlüsselrolle bei der Zellteilung spielen. Wie in Abbildung 1 dargestellt, hätte das neu gebildete chimäre Chromosom unmittelbar nach der angeblichen Kopf-an-Kopf-Fusion der beiden Chromosomen zwei Zentromerstellen gehabt. In einem solchen Fall wäre eines der Zentromere funktionsfähig, während das andere deaktiviert wäre. Das Vorhandensein von zwei aktiven Zentromeren ist eine schlechte Nachricht für Chromosomen und würde zu Funktionsstörungen und Zellzerstörung führen.
Interessanterweise ist der Nachweis für ein kryptisches (behindertes) Zentromer auf menschlichem Chromosom 2 noch schwächer als der für eine telomerreiche Fusionsstelle. Evolutionisten erklären das Fehlen eines klar unterscheidbaren nichtfunktionellen sekundären Zentromers, indem sie argumentieren, dass ein zweites Zentromer schnell dagegen ausgewählt worden wäre. Danach hätte sich das behinderte Zentromer im Laufe der Zeit verschlechtert, da es keine funktionellen Einschränkungen mehr gab, indem es etwas Nützliches im Genom tat.
Der Nachweis für ein zweites Restzentromer in jedem Stadium der Sequenzdegeneration ist jedoch für das evolutionäre Paradigma problematisch. Funktionelle Zentromersequenzen bestehen aus einem repetitiven DNA-Typ namens Alphoidsequenzen, wobei jede Alphoidrepetition etwa 171 Basen lang ist. Einige Arten von Alphoid-Wiederholungen finden sich im gesamten Genom, während andere für Zentromere spezifisch sind. Die Struktur der Sequenzen, die an der kryptischen Zentromerstelle auf menschlichem Chromosom 2 gefunden wurden, stimmt nicht mit denen überein, die mit funktionellen menschlichen Zentromeren assoziiert sind.13 Noch schlimmer für das Evolutionsmodell ist, dass sie keine sehr ähnlichen Gegenstücke im Schimpansengenom haben — sie sind menschenspezifisch.13
Das angebliche fossile Zentromer ist auch im Vergleich zu einem echten außergewöhnlich klein. Die Größe eines normalen menschlichen Zentromers liegt in der Länge zwischen 250.000 und 5.000.000 Basen.14 Das angebliche kryptische Zentromer ist nur 41.608 Basen lang, aber es ist auch wichtig zu beachten, dass es drei verschiedene Regionen davon gibt, die nicht einmal alphoide Wiederholungen sind.15 Zwei davon werden als Retroelemente bezeichnet, wobei eines ein LPA3 / LINE Repeat mit einer Länge von 5.957 Basen und das andere ein SVA-E-Element mit 2.571 Basen ist. Wenn wir die Insertionen dieser nicht-alphoiden Sequenzen subtrahieren, ergibt sich eine Länge von nur 33.080 Basen, was einem Bruchteil der Länge eines echten Zentromers entspricht.
Das schwerwiegendste evolutionäre Problem bei der Idee eines fossilen Zentromers ist jedoch, dass es wie die angebliche Fusionsstelle innerhalb eines Gens positioniert ist. Das angebliche kryptische Zentromer befindet sich innerhalb des ANKRD30BL-Gens und seine Sequenz umfasst sowohl Intron- als auch Exon-Regionen des Gens.12,15
Tatsächlich kodiert der Teil der angeblichen fossilen Zentromersequenz, der in einem Exon landet, tatsächlich für Aminosäuren im Protein des resultierenden Gens. Das Gen produziert ein Protein, von dem angenommen wird, dass es an der Interaktion des strukturellen Netzwerks von Proteinen innerhalb der Zelle beteiligt ist, das als Zytoskelett in Verbindung mit in die Zellmembran eingebetteten Rezeptorproteinen bezeichnet wird.16 Die Tatsache, dass das sogenannte fossile oder kryptische Zentromer eine funktionelle Region innerhalb eines wichtigen proteinkodierenden Gens ist, widerlegt die Vorstellung, dass es sich um ein nicht mehr funktionierendes Zentromer handelt.
Fazit: Keine Fusion
Aufgrund der verworrenen Signaturen und der geringen Größe der angeblichen Fusions- und fossilen Zentromerstellen ist es höchst fragwürdig, dass ihre Sequenz evolutionär von einer alten Chromosomenfusion abgeleitet wurde. Nicht nur das, sie repräsentieren funktionelle Sequenz innerhalb von Genen. Die angebliche Fusionsstelle ist ein wichtiger genetischer Schalter, der als Promotor innerhalb des langen, nicht kodierenden DDX11L2-RNA-Gens bezeichnet wird, und das sogenannte fossile Zentromer enthält sowohl kodierende als auch nicht kodierende Sequenzen innerhalb eines großen Ankyrin-Wiederholungsprotein-kodierenden Gens.
Dies ist ein unbestreitbarer Doppelschlag gegen die ganze mythische Fusionsidee, der ihre Gültigkeit völlig zerstört. Die überwältigende wissenschaftliche Schlussfolgerung ist, dass die Fusion nie stattgefunden hat.
- Yunis, J. J. und O. Prakash. 1982. Der Ursprung des Menschen: ein chromosomales bildliches Erbe. Wissenschaft. 215 (4539): 1525-1530.
- Ijdo, J. W. et al. 1991. Ursprung des menschlichen Chromosoms 2: Eine Telomer–Telomer-Fusion der Vorfahren. Proceedings der Nationalen Akademie der Wissenschaften. 88 (20): 9051-9055.
- Chaves, R. et al. 2003. Molekulare zytogenetische Analyse und zentromere Satellitenorganisation eines Romans 8; 11 Translokation bei Schafen: ein mögliches Zwischenprodukt in der Evolution biologischer Chromosomen. Säugetiergenom. 14 (10): 706-710.
- Tsipouri, V. et al. 2008. Vergleichende Sequenzanalysen zeigen Stellen von Chromosomenfusionen der Vorfahren im indischen Muntjac-Genom. In: Genome Biology. 9 (10): R155.
- Adega, F., H. Guedes-Pinto und R. Chaves. 2009. Satelliten-DNA in der Karyotyp—Evolution von Haustieren – Klinische Überlegungen. Zytogenetische und Genomforschung. 126 (1-2): 12-20.
- Tomkins, J. P. und J. Bergman. 2011. Telomere: Auswirkungen auf das Altern und Beweise für intelligentes Design. Journal der Schöpfung. 25 (1): 86-97.
- Fan, Y. et al. 2002. Genomische Struktur und Evolution der angestammten Chromosomenfusionsstelle in 2q13–2q14.1 und paralogischen Regionen auf anderen menschlichen Chromosomen. Genomforschung. 12 (11): 1651-1662.
- Fan, Y. et al. 2002. Gengehalt und Funktion der angestammten Chromosomenfusionsstelle im menschlichen Chromosom 2q13–2q14.1 und in paralogischen Regionen. Genomforschung. 12 (11): 1663-1672.
- Tomkins, J. P. 2013. Die “Fusionsstelle” des menschlichen Chromosoms 2 kodiert für eine aktive DNA—Bindungsdomäne innerhalb einer komplexen und stark exprimierten Gen-negierenden Fusion. In: Answers Research Journal. 6: 367-375.
- Azzalin, C. M., S. G. Nergadze und E. Giulotto. 2001. Menschliche intrachromosomale telomerähnliche Wiederholungen: Sequenzorganisation und Ursprungsmechanismen. Chromosom. 110: 75-82.
- Ruiz-Herrera, A. et al. 2008. Telomere Wiederholungen weit entfernt von den Enden: Mechanismen der Entstehung und Rolle in der Evolution. Zytogenetische und Genomforschung. 122 (3-4): 219-228.
- Tomkins, J. P. 2018. Kombinatorische genomische Daten widerlegen die evolutionäre Fusion des menschlichen Chromosoms 2 und erstellen ein Modell des funktionalen Designs für interstitielle Telomerwiederholungen. In Proceedings der Achten Internationalen Konferenz über Kreationismus. J. H. Whitmore, Hrsg. Pittsburgh, PA: Creation Science Fellowship, 222-228.
- Tomkins, J. und J. Bergman. 2011. Das Chromosom-2-Fusionsmodell der menschlichen Evolution – Teil 2: Reanalyse der genomischen Daten. Journal der Schöpfung. 25 (2): 111-117.
- Aldrup-Macdonald, M. E. und B. A. Sullivan. 2014. Vergangenheit, Gegenwart und Zukunft der humanen Zentromergenomik. Gene (Basel). 5 (1): 33-50.
- Tomkins, J. P. 2017. Debunking the Debunkers: Eine Antwort auf Kritik und Verschleierung bezüglich der Widerlegung der Fusion des menschlichen Chromosoms 2. In: Answers Research Journal. 10: 45-54.
- Woronin, D. A. und E. V. Kiseleva. 2008. Funktionelle Rolle von Proteinen, die Ankyrin-Wiederholungen enthalten. Zell- und Gewebebiologie. 49 (12): 989-999.
* Dr. Tomkins ist Direktor für Biowissenschaften am Institute for Creation Research und promovierte in Genetik an der Clemson University.