El Instituto para la Investigación de la Creación
 Uno de los argumentos más populares utilizados para los seres humanos que supuestamente evolucionan de simios se conoce como la fusión cromosómica. El impetusetu de este concepto es el problema evolutivo de que los simios tienen un par extra de cromosomas: los humanos tienen 46 mientras que los simios tienen 48. Si los humanos evolucionaron de una criatura parecida a un mono hace solo tres a seis millones de años, un simple punto en el gran esquema de la historia evolutiva, ¿por qué los humanos y los simios tienen esta discrepancia?
Uno de los argumentos más populares utilizados para los seres humanos que supuestamente evolucionan de simios se conoce como la fusión cromosómica. El impetusetu de este concepto es el problema evolutivo de que los simios tienen un par extra de cromosomas: los humanos tienen 46 mientras que los simios tienen 48. Si los humanos evolucionaron de una criatura parecida a un mono hace solo tres a seis millones de años, un simple punto en el gran esquema de la historia evolutiva, ¿por qué los humanos y los simios tienen esta discrepancia?
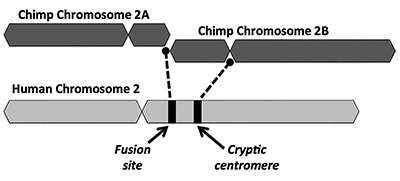
La solución evolutiva propone que una fusión de extremo a extremo de dos cromosomas pequeños similares a simios (llamados 2A y 2B) produjo el cromosoma humano 2 (Figura 1). El concepto de fusión surgió por primera vez en 1982, cuando los científicos examinaron las similitudes de los cromosomas humanos y simios bajo un microscopio. Si bien la técnica era un poco cruda, fue suficiente para poner en marcha la idea.1
El Llamado Sitio De Fusión
La primera firma de ADN real de un posible evento de fusión se descubrió en 1991 en el cromosoma humano número 2.2 Los investigadores encontraron un pequeño y confuso grupo de secuencias finales similares a telómeros que se parecían vagamente a una posible fusión. Los telómeros son una secuencia de seis bases de las letras de ADN TTAGGG repetidas una y otra vez en los extremos de los cromosomas.
Sin embargo, la firma de fusión era algo así como un enigma basado en las fusiones reales que ocasionalmente ocurren en la naturaleza. Todas las fusiones documentadas en animales vivos involucran un tipo específico de secuencia llamada ADN satélite (satDNA) ubicado en los cromosomas y encontrado en roturas y fusiones.3-5 A la firma de fusión en el cromosoma 2 humano le faltaba este satDNA revelador.6
Otro problema es el pequeño tamaño del sitio de fusión, que solo tiene 798 letras de ADN. Las secuencias de telómeros en los extremos de los cromosomas tienen una longitud de 5.000 a 15.000 bases. Si dos cromosomas se fusionaron, debería ver una firma de telómeros fusionados de 10.000 a 30.000 bases de largo, no 798.
No solo el tamaño pequeño es un problema para la historia de la fusión, la firma realmente no representa una fusión clara de telómeros. La Figura 2 muestra las letras de ADN del sitio de fusión de 798 bases con las secuencias de telómeros de seis bases (letras de ADN) intactas enfatizadas en negrita. Cuando la secuencia de fusión se compara con la de una firma de fusión prístina del mismo tamaño, es solo un 70% idéntica en general.

Investigadores seculares han señalado esta discrepancia y han etiquetado el sitio de fusión como significativamente “degenerado”.”7 Dado el modelo teórico estándar de la evolución humana, debería ser idéntico en un 98 a 99%, no en un 70%. Los investigadores que describieron este descubrimiento comentaron: “Las matrices de repeticiones cabeza a cabeza en el sitio de fusión se han degenerado significativamente (14%) de las matrices casi perfectas de (TTAGGG)n encontradas en los telómeros” y formularon la pregunta pertinente: “Si la fusión ocurrió dentro de las matrices de repetición telomérica de menos de ~6 Ma, ¿por qué las matrices en el sitio de fusión son tan degeneradas?”7 Cabe señalar que la degeneración del 14% citada por los autores se refiere a la corrupción de solo las secuencias de seis bases en sí, no de las 798 bases completas.
¿El Sitio de Fusión Dentro de un Gen?
El hallazgo antievolucionario más notable sobre el sitio de fusión resultó ser su ubicación y lo que realmente hace. Este descubrimiento se produjo mientras leía el artículo de investigación que informaba de un análisis detallado de 614.000 bases de secuencia de ADN que rodeaban el supuesto sitio de fusión. Noté en una de las figuras que el sitio de fusión estaba ubicado dentro de un gen, y sorprendentemente esta rareza ni siquiera fue reconocida en el texto del artículo.8
Un hallazgo como este es muy notable. Tal vez esta información hubiera sido el clavo en el ataúd evolutivo, por así decirlo, por lo que los investigadores se negaron a discutirlo. Esta anomalía mayor me inspiró a examinar el sitio de fusión mucho más de cerca. Este artículo fue publicado en 2002, y lo noté en 2013. Mientras tanto, se había publicado una gran cantidad de datos sobre la estructura y la función del genoma humano, y probablemente había mucho más en la historia que debía descubrirse.
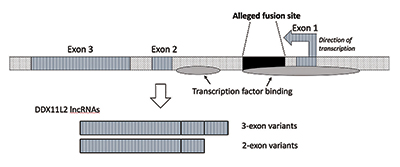
Cuando realicé más investigación, verificé que el sitio de fusión estaba ubicado dentro de un gen de ARN helicasa que ahora se llama DDX11L2. La mayoría de los genes en plantas y animales tienen sus segmentos codificantes en trozos llamados exones para que se puedan empalmar alternativamente. Basándose en la adición o exclusión de exones, los genes pueden producir una variedad de productos. Las regiones intermedias entre los exones se llaman intrones, que a menudo contienen una variedad de señales e interruptores que controlan la función génica. El supuesto sitio de fusión está ubicado dentro del primer intrón del gen DDX11L2 (Figura 3).9
La molécula de ADN es de doble cadena, con una hebra positiva y una hebra negativa. Fue diseñado de esta manera para maximizar la densidad de información al tiempo que aumenta la eficiencia y la función. Como resultado, hay genes que corren en diferentes direcciones en las hebras opuestas. Resulta que el gen DDX11L2 está codificado en la cadena negativa. Debido a que los genes en los seres humanos son como navajas suizas y pueden producir una variedad de ARN, en el caso del gen DDX11L2 produce variantes cortas que consisten en dos exones y variantes largas con tres (Figura 3).9
El Sitio de fusión Es un Promotor de Genes
¿Qué podría estar haciendo este gen DDX11L2? Mi investigación mostró que se expresa en al menos 255 tipos de células o tejidos diferentes.9 También se coexpresa (se enciende al mismo tiempo) con una variedad de otros genes y está conectado a procesos asociados con la señalización celular en la matriz extracelular y la producción de células sanguíneas. La ubicación de la llamada secuencia de fusión dentro de un gen funcional asociado con la genética de una variedad de procesos celulares refuta fuertemente la idea de que es el subproducto accidental de una fusión telomérica cabeza a cabeza. ¡Los genes no están formados por fusiones cromosómicas catastróficas!
Aún más sorprendente es que el sitio de fusión es en sí mismo funcional y cumple un importante propósito de ingeniería. El sitio en realidad actúa como un interruptor para controlar la actividad de los genes. En este sentido, una gran cantidad de datos bioquímicos mostraron que 12 proteínas diferentes llamadas factores de transcripción regulan este segmento del gen. Una de ellas no es otra que la ARN polimerasa II, la principal enzima que copia las moléculas de ARN del ADN en un proceso llamado transcripción. Otro apoyo a este descubrimiento es el hecho de que el proceso real de transcripción se inicia dentro de la región del llamado sitio de fusión.
Técnicamente, llamaríamos a la actividad en el supuesto sitio de fusión una región promotora. Los promotores son los principales interruptores al principio de los genes que los encienden y también son donde la ARN polimerasa comienza a crear un ARN. Muchos genes tienen promotores alternativos como el gen DDX11L2.
En realidad hay dos áreas de unión de factores de transcripción en el gen DDX11L2. El primero está en el promotor directamente delante del primer exón, y el segundo está en el primer intrón correspondiente a la secuencia del sitio de fusión. El gen DDX11L2 no solo está controlado de forma compleja, con la supuesta secuencia de fusión jugando un papel clave, sino que incluso las transcripciones de ARN producidas son muy intrincadas. Los ARN en sí contienen una amplia variedad de sitios de unión y control para una clase de pequeñas moléculas reguladoras llamadas microRNAs.9
Las Secuencias de Telómeros Internos funcionales Se encuentran en Todo el Genoma
La presencia de secuencias de telómeros localizadas internamente se encuentra en todo el genoma humano. Estas repeticiones de telómeros aparentemente fuera de lugar se han denominado telómeros intersticiales. La presencia de estas secuencias presenta otro desafío para la idea del sitio de fusión. Es un hecho que muy pocas de las repeticiones de telómeros en el sitio de fusión ocurren en tándem. Como se observa en la Figura 2, la secuencia del sitio de fusión de 798 bases contiene solo unos pocos casos en los que dos repeticiones están realmente en tándem y ninguna que tenga tres repeticiones o más. Sin embargo, hay muchos otros sitios de telómeros intersticiales en todo el genoma humano donde las repeticiones ocurren en tándem perfecto de tres a diez veces o más.10-11
Incluso además de su papel en los extremos de los cromosomas, parece que las repeticiones teloméricas intersticiales pueden cumplir una función importante en el genoma relacionada con la expresión génica. En un proyecto de investigación reciente, identifiqué repeticiones de telómeros en todo el genoma humano y luego intersecé sus ubicaciones genómicas con una diversidad de conjuntos de datos que contenían información bioquímica funcional para la actividad génica.12 Literalmente miles de repeticiones teloméricas internas en todo el genoma se asociaron directamente con las características distintivas de la expresión génica. El mismo tipo de unión de factores de transcripción y actividad génica que ocurría en el supuesto sitio de fusión también ocurría en todo el genoma en numerosas repeticiones teloméricas intersticiales. Claramente, estas características del ADN no son accidentes de la evolución, sino código funcional diseñado a propósito e inteligentemente.
Falso Centrómero Críptico Dentro de un Gen
Otro problema clave con el modelo de fusión es la falta de evidencia viable para una firma de una región de centrómero extra. Los centrómeros son secciones de cromosomas, a menudo en ubicaciones centrales, que desempeñan funciones clave durante la división celular. Como se muestra en la Figura 1, el cromosoma quimérico recién formado habría tenido dos sitios de centrómeros inmediatamente después de la supuesta fusión cabeza a cabeza de los dos cromosomas. En tal caso, uno de los centrómeros sería funcional mientras que el otro estaría deshabilitado. La presencia de dos centrómeros activos es una mala noticia para los cromosomas y conduciría a la disfunción y destrucción celular.
Curiosamente, la evidencia de un centrómero críptico (desactivado) en el cromosoma 2 humano es aún más débil que la de un sitio de fusión rico en telómeros. Los evolucionistas explican la falta de un centrómero secundario no funcional claramente distinguible argumentando que un segundo centrómero habría sido rápidamente seleccionado en contra. Después de eso, el centrómero discapacitado se habría deteriorado con el tiempo ya que ya no había restricciones funcionales colocadas en él al hacer algo útil en el genoma.
Sin embargo, la evidencia de un segundo centrómero remanente en cualquier etapa de degeneración secuencial es problemática para el paradigma evolutivo. Las secuencias de centrómeros funcionales se componen de un tipo repetitivo de ADN llamado secuencias alfoides, con cada repetición alfoide de aproximadamente 171 bases de largo. Algunos tipos de repeticiones alfoides se encuentran en todo el genoma, mientras que otros son específicos de los centrómeros. La estructura de las secuencias encontradas en el sitio del centrómero críptico en el cromosoma 2 humano no coincide con las asociadas con centrómeros humanos funcionales.13 Aún peor para el modelo evolutivo es que no tienen contrapartes muy similares en el genoma de los chimpancés, son específicos de los humanos.13
El supuesto centrómero fósil también es excepcionalmente pequeño en comparación con uno real. El tamaño de un centrómero humano normal varía en longitud entre 250.000 y 5.000.000 de bases.14 El supuesto centrómero críptico tiene solo 41.608 bases de largo, pero también es importante tener en cuenta que hay tres regiones diferentes que ni siquiera son repeticiones alfoides.15 Dos de estos se llaman retroelementos, siendo uno un LPA3/repetición DE LÍNEA de 5.957 bases de largo y el otro un elemento SVA-E con 2.571 bases. Cuando restamos las inserciones de estas secuencias no alfoides, da una longitud de solo 33.080 bases, que es una fracción de la longitud de un centrómero real.
El problema evolutivo más grave con la idea de un centrómero fósil, sin embargo, es que, al igual que el supuesto sitio de fusión, está ubicado dentro de un gen. El supuesto centrómero críptico se encuentra dentro del gen ANKRD30BL, y su secuencia abarca las regiones intrón y exón del gen.12,15
De hecho, la parte de la supuesta secuencia de centrómeros fósiles que aterriza dentro de un exón en realidad codifica para aminoácidos en la proteína del gen resultante. El gen produce una proteína que se cree que está involucrada en la interacción de la red estructural de proteínas dentro de la célula, llamada citoesqueleto, en conexión con proteínas receptoras incrustadas en la membrana celular.16 El hecho de que el llamado centrómero fósil o críptico sea una región funcional dentro de un importante gen codificador de proteínas refuta completamente la idea de que es un centrómero extinto.
Conclusión: No Fusión
Debido a las firmas confusas y los pequeños tamaños de los supuestos sitios de fusión y centrómeros fósiles, es altamente cuestionable que su secuencia se derivara evolutivamente de una fusión cromosómica antigua. No solo eso, representan la secuencia funcional dentro de los genes. El supuesto sitio de fusión es un importante interruptor genético llamado promotor dentro del gen de ARN largo no codificante DDX11L2, y el llamado centrómero fósil contiene secuencia codificante y no codificante dentro de un gen codificador de proteína de repetición de anquirina grande.
Este es un doble golpe innegable contra toda la idea de fusión mítica, destruyendo por completo su validez. La conclusión científica abrumadora es que la fusión nunca ocurrió.
- Yunis, J. J. and O. Prakash. 1982. The origin of man: a chromosomal pictorial legacy (en inglés). Ciencia. 215 (4539): 1525-1530.
- Ijdo, J. W. et al. 1991. Origen del cromosoma humano 2: Una fusión ancestral telómero-telómero. Actas de la Academia Nacional de Ciencias. 88 (20): 9051-9055.
- Chaves, R. et al. 2003. Análisis citogenético molecular y organización satelital centromérica de una nueva translocación 8;11 en ovejas: un posible intermedio en la evolución de cromosomas biarmed. Genoma de Mamíferos. 14 (10): 706-710.
- Tsipouri, V. et al. 2008. Los análisis comparativos de secuencias revelan sitios de fusiones cromosómicas ancestrales en el genoma indio de muntjac. Biología Genómica. 9 (10): R155.
- Adega, F., H. Guedes-Pinto, and R. Chaves. 2009. Satellite DNA in the Cariotype Evolution of Domestic Animals—Clinical Considerations (en inglés). Investigación Citogenética y Genómica. 126 (1-2): 12-20.
- Tomkins, J. P. and J. Bergman. 2011. Telómeros: implicaciones para el envejecimiento y evidencia para el diseño inteligente. Diario de la Creación. 25 (1): 86-97.
- Fan, Y. et al. 2002. Estructura Genómica y Evolución del Sitio de Fusión Cromosómica Ancestral en las Regiones 2t13–2t14.1 y Paralogous en Otros Cromosomas Humanos. Investigación del Genoma. 12 (11): 1651-1662.
- Fan, Y. et al. 2002. Contenido Genético y Función del Sitio de Fusión Cromosómica Ancestral en las Regiones Cromosómicas Humanas 2q13–2q14.1 y Paralogous. Investigación del Genoma. 12 (11): 1663-1672.
- Tomkins, J. P. 2013. El Supuesto “Sitio de Fusión” Del Cromosoma 2 Humano Codifica un Dominio De Unión de ADN Activo Dentro de una Fusión Compleja y Altamente Expresada que Niega Genes. Answers Research Journal (en inglés). 6: 367-375.
- Azzalin, C. M., S. G. Nergadze, and E. Giulotto. 2001. Repeticiones de tipo telomérico intracromosómico humano: organización de secuencias y mecanismos de origen. Cromosoma. 110: 75-82.
- Ruiz-Herrera, A. et al. 2008. Repeticiones teloméricas lejos de los fines: mecanismos de origen y papel en la evolución. Investigación Citogenética y Genómica. 122 (3-4): 219-228.
- Tomkins, J. P. 2018. Los datos genómicos combinatorios refutan la fusión evolutiva del cromosoma 2 humano y construyen un modelo de diseño funcional para repeticiones teloméricas intersticiales. En Actas de la Octava Conferencia Internacional sobre Creacionismo. J. H. Whitmore, ed. Pittsburgh, PA: Creation Science Fellowship, 222-228.
- Tomkins, J. and J. Bergman. 2011. The chromosome 2 fusion model of human evolution-part 2: re-analysis of the genomic data (en inglés). Diario de la Creación. 25 (2): 111-117.
- Aldrup-Macdonald, M. E. and B. A. Sullivan. 2014. El Pasado, Presente y Futuro de la Genómica de Centrómeros Humanos. Genes (Basilea). 5 (1): 33-50.
- Tomkins, J. P. 2017. Desacreditar a los Desacreditadores: Una Respuesta a la Crítica y la Ofuscación Con Respecto a la Refutación de la Fusión del Cromosoma 2 Humano. Answers Research Journal (en inglés). 10: 45-54.
- Voronin, D. A. y E. V. Kiseleva. 2008. Función Funcional de las Proteínas Que Contienen Repeticiones de Anquirina. Biología Celular y Tisular. 49 (12): 989-999.
* El Dr. Tomkins es Director de Ciencias de la Vida en el Instituto de Investigación de la Creación y obtuvo su doctorado en genética en la Universidad de Clemson.