¿Qué es la PCR de colonias?
La PCR de colonias es un método de PCR rápido y de alto rendimiento para determinar la presencia o ausencia del ADN insertado en el plásmido directamente de las colonias bacterianas.
La clonación molecular es uno de los métodos más populares para la transformación del ADN desde hace mucho tiempo. Sin embargo, para determinar la presencia o ausencia del inserto de ADN tenemos que realizar experimentos de transformación.
La PCR de colonias es un método novedoso en el que al diseñar los cebadores específicos de ADN insertados, podemos identificar si nuestro ADN de interés se inserta en el plásmido o no.
Sin embargo, no es tan simple como estamos discutiendo.
En este artículo, nos centraremos en la PCR de la Colonia especialmente, el principio de la PCR de la Colonia, sus ventajas y limitaciones.
Para eso, tenemos que entender varios términos y temas. Comenzaremos nuestro tema desde lo básico. El contenido del artículo es,
Temas Clave:
“Un plásmido es el ADN circular bacteriano que se replica independientemente del cromosoma bacteriano y se utiliza en la manipulación y transferencia de genes.”
La clonación genética es una herramienta genética molecular tradicional utilizada desde hace mucho tiempo en los laboratorios. Brevemente, en la clonación de genes, el gen de nuestro interés se inserta en el plásmido a través de medios artificiales. Este ADN se replica independientemente del cromosoma bacteriano.
Los plásmidos se utilizan en realidad para generar muchas copias de segmentos cortos de ADN. Debido a que las bacterias se replican más rápido que cualquier otro organismo, podemos generar muchas copias del gen de nuestro interés insertándolo en el plásmido bacteriano.
El plásmido F, el plásmido Col, el plásmido degradativo y el plásmido de resistencia son varios tipos comunes de plásmidos que se encuentran en las bacterias.
Además, el plásmido puede funcionar como portador molecular que transfiere segmentos cortos de ADN de una célula a otra.
Hemos cubierto un increíble artículo en profundidad sobre ADN plásmido. Léalo aquí: ADN Plásmido-Estructura, Función, Aislamiento Y Aplicaciones.
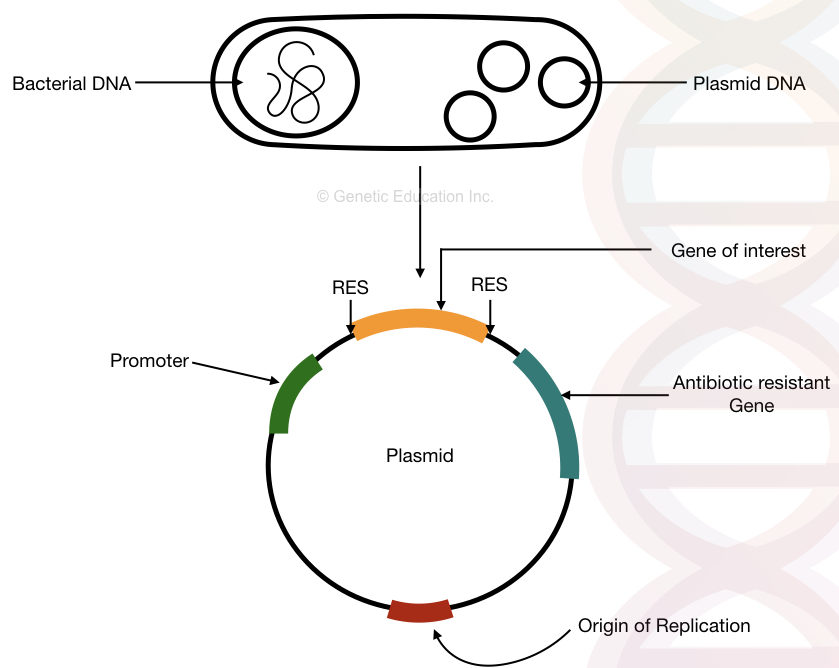
La estructura del ADN del plásmido bacteriano con, el origen de la replicación, el gen de resistencia a los antibióticos, el promotor y el gen de interés.
Además de bacterias, varios procariotas también contienen ADN plásmido. La función principal del plásmido en las bacterias es para su supervivencia en las duras condiciones.
Como el plásmido transfiere el gen de nuestro interés, es muy importante determinar si nuestro gen de interés está insertado o no en el plásmido.
Para eso, podemos usar varios métodos como PCR y cultivo microbiano.
Platear las colonias toma más tiempo y la sensibilidad del método tampoco es buena. La posibilidad de contaminación siempre es alta en los métodos de cultivo bacteriano.
Por lo que los resultados no son precisos.
Nuestra PCR también ayuda aquí. Usando el método de PCR de colonias, se puede determinar o identificar un inserto de ADN.
- Cómo configurar un laboratorio de extracción de ADN: Una guía completa (productos químicos, instrumentos y otras utilidades).
- Deleción del cromosoma 6p: Una razón para no sentir dolor, hambre ni sueño
¿Qué es la PCR de colonias?
La PCR de colonias es la modificación de la PCR convencional en la que las colonias bacterianas se utilizan directamente como plantilla de PCR.
El ADN plásmido que contiene el ADN de nuestro interés se amplifica en las condiciones dependientes de la temperatura cíclica.
La representación gráfica de la PCR de la colonia se muestra en la siguiente figura,
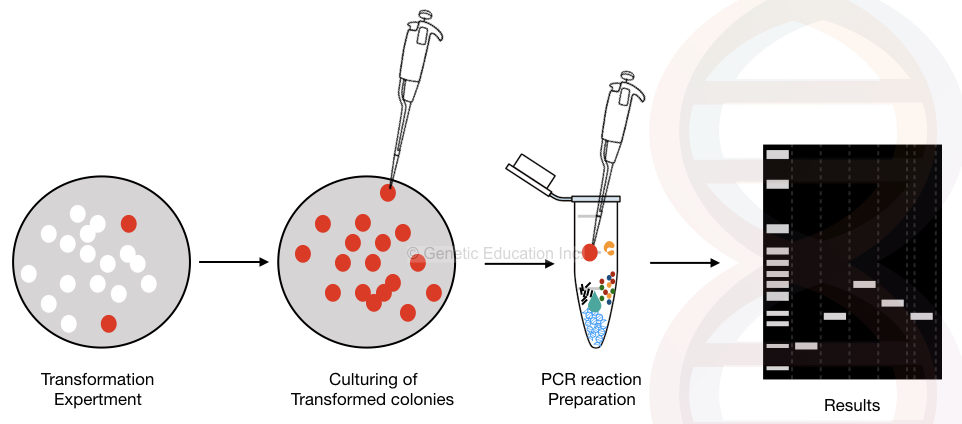
La visión general del método de PCR de colonias.
Principio de la PCR de Colonias:
La colonia bacteriana que contiene el plásmido se puede amplificar directamente utilizando dos juegos de cebadores. Los cebadores específicos de inserción que amplifican la secuencia de inserción y los cebadores flanqueantes específicos de vectores, que amplifican el ADN plásmido que no es el ADN insertado (regiones flanqueantes en ambos lados del inserto).
Utilizando los cebadores de flanqueo de inserción (que amplifican el resto del ADN) se puede determinar el tamaño de nuestro inserto de ADN.
Se recoge una colonia bacteriana y se añade directamente a la mezcla maestra que contiene todos los reactivos para PCR. Además, al agregar un paso de calentamiento inicial a la PCR, el ADN plásmido sale de la célula bacteriana y se amplifica en la reacción.
Este es el principio básico de la PCR de colonias, Sin embargo, se puede modificar dependiendo de los requisitos.
El protocolo de PCR de colonias:
La PCR de colonias es una de las excelentes modificaciones de la PCR convencional. En lugar de ADN de plantilla, las colonias bacterianas se agregan directamente a la reacción. Además de esto, la polimerasa de ADN Taq, los cebadores, el tampón de reacción de PCR y DD/W también se agregan a la reacción de PCR.
Aquí en la PCR de colonias, la selección de cebadores es muy importante. Además, la selección de cebadores depende del objetivo de nuestro experimento.
¿Qué tipo de información queremos de nuestro experimento de PCR de colonias?
-
-
- Solo información sobre la presencia o ausencia del inserto.
- Información sobre el tamaño del inserto.
- Información sobre la orientación del inserto.
-
Dependiendo de que diferentes cebadores de PCR están diseñados para la colonia de PCR.
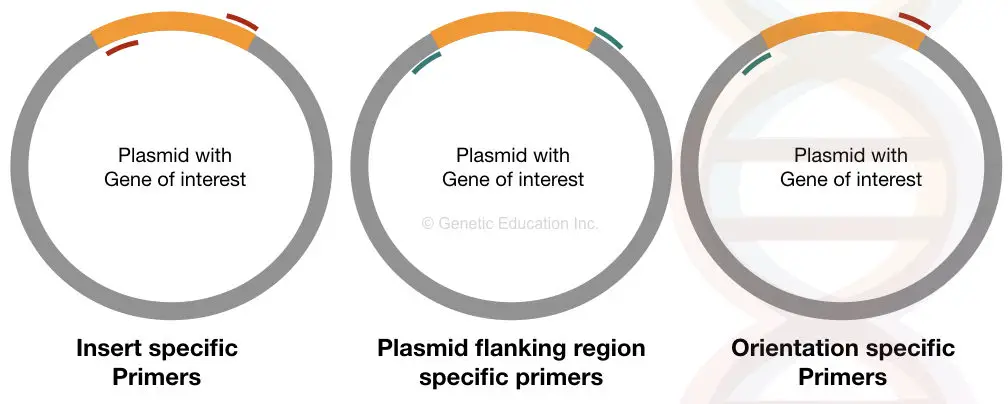
Los cebadores específicos de inserción se unen a la ubicación específica en ambos lados del ADN insertado de nuestro interés. Si se transfiere correctamente al plásmido, estos cebadores pueden unirse a él, de lo contrario, no puede unirse.
Este juego de imprimación proporciona información sobre la presencia o ausencia del inserto.
Las imprimaciones específicas de orientación son imprimaciones únicas en las que una imprimación se une dentro del inserto y otra imprimación se une a la secuencia de ADN del plásmido (secuencia distinta del ADN del inserto).
Este tipo de juego de imprimación proporciona información sobre la orientación del ADN insertado de nuestro interés. Si nuestro ADN insertado no está correctamente unido al vector, la imprimación específica de ese lado de la secuencia no se puede unir, y no obtendremos la amplificación.
Los imprimadores específicos de plásmidos también son tan importantes como los imprimadores específicos de orientación. Este conjunto de cebadores está diseñado a partir de la región de flanqueo del inserto que se une al exterior del ADN de nuestro interés.
Este juego de imprimación ayuda a determinar el tamaño del inserto. Expande otras regiones además del ADN del inserto.
La reacción de PCR para realizar la PCR de la colonia es la siguiente,
| Componente | Concentración | Cantidad |
| Mezcla maestra (Especial
para la PCR de Colonias) |
1X | 12µL |
| Tampón de reacción PCR
Con MgCl2 de 2 mm* |
1X | 5µL |
| Imprimación delantera | 10pM | 1µL |
| Imprimación inversa | 10pM | 1µL |
| Sobrenadante | 3µL | |
| Agua | 3µL | |
| Total | ——— | 25μl |
El procedimiento de la colonia de PCR:
Bien, la colonia de PCR no necesita de ADN extraído.
No estamos extrayendo ADN aquí. En su lugar, se utilizan varios otros métodos para aumentar la sensibilidad de la reacción.
Ok, ¿por qué no estamos extrayendo ADN para el ADN plásmido?
Debido a que la razón es simple, la membrana celular de la célula bacteriana es muy lisa.
ya habíamos discutido la membrana celular de la célula bacteriana. Léalo aquí: Diferentes tipos de métodos de extracción de ADN
Una bacteria contiene una membrana celular blanda que se puede lisar fácilmente calentándola o centrifugándola a alta velocidad.
Además, no necesitamos el propio ADN bacteriano. El plásmido circular circulante está presente en el citoplasma de la bacteria, por lo que no se requieren pasos de purificación adicionales. Al romper la membrana celular, nuestro ADN de plantilla está listo para la amplificación.
Ok, pasemos rápidamente por el método para obtener un buen ADN plásmido.
Con la ayuda del selector estéril, seleccione varias colonias bacterianas y transfiéralas al tubo Eppendorf.
Ahora agregue el buffer TE y mézclelo bien. También puedes usar D / W.
Calentar la muestra en el baño de agua hirviendo durante 20 minutos.
Cúbralo suavemente.
Centrifugar la muestra a alta velocidad durante 2 minutos.
Transfiera el sobrenadante a otro tubo y utilícelo como plantilla de ADN.
Se agrega una muestra de 20 µl a la reacción.
Información adicional:
¿Por qué sobrenadante y no pellet?
El ADN es una biomolécula de vida. El ADN plásmido es incluso más pequeño que el ADN nuclear bacteriano. Contiene solo varios genes de hasta 1000 bp a 20,000 bp.
Por lo tanto, solo centrifugándolo, el ADN plásmido más ligero sale de la célula y se asienta en el sobrenadante, mientras que el gránulo contiene proteínas y ADN nuclear, por lo que no lo estamos usando.
Ahora vamos al grano.
Nuestro plásmido está listo para amplificación.
En otro método,
Use la colonia bacteriana directamente.
Este método es una combinación de PCR de inicio en caliente y PCR de colonia.
Las colonias bacterianas se recogen y se añaden al tubo de reacción de PCR.
Los tubos se colocan en la máquina de PCR. Se agrega un escalón de calentamiento adicional.
Al calentarlo de 5 a 7 minutos, el ADN plásmido sale de la célula.
Ahora los cebadores específicos amplifican el ADN que insertamos. Y los cebadores de flanqueo amplifican el resto del ADN.
La amplificación se realiza durante 20 a 25 ciclos. El ciclismo condiciones para la colonia de PCR se enumeran a continuación,
| Pasos de la PCR | Desnaturalización Inicial | Desnaturalización | Recocido | Extensión | extensión Final |
| Temperatura | 95 C | 95 C | 55-65 C | 72 C | 72 C |
| Tiempo | 3min | 10 seg | 45 seg | 50 seg | 5 min |
| ——- | ——- | 25 ciclos | —– | ——- |
Lea el interesante artículo sobre PCR convencional: Una Guía Completa de la Reacción en Cadena de la Polimerasa
Consejos para la mejora:
Use solo unas pocas colonias, ya que muchas colonias aumentan la probabilidad de que se produzcan ligaduras inespecíficas.
Use control positivo y control negativo.
Como control positivo se utilizó la imprimación de flanqueo, incluso si el inserto no está presente, la reacción de PCR da una banda de ADN del ADN plásmido que indica que la reacción que preparamos es correcta.
Como control negativo, utilice el plásmido no transformado (plásmido sin inserto de ADN), este plásmido de ADN se amplifica solo si el inserto está presente.
Como inserto, utilice secuencias de ADN cortas, las secuencias de ADN más largas aumentan la posibilidad de uniones inespecíficas y fallo de la reacción de PCR.
Además, utilice programas de PCR más cortos.
La aplicación principal de la PCR de colonias es en la identificación de la ligadura correcta y la inserción de ADN de inserción en bacterias, así como en plásmidos de levadura.
Después de completar la reacción de PCR de la colonia, los productos de PCR se ejecutan en el gel de agarosa al 2%. Los resultados del experimento se muestran en la siguiente figura,
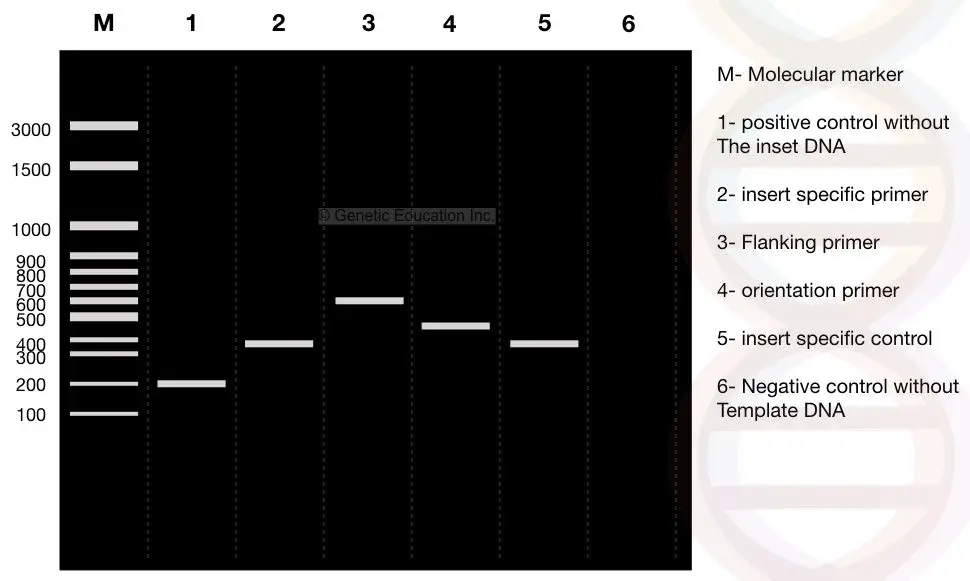
Ahora observe cuidadosamente los resultados, la M es el marcador molecular de ADN de 3000 bp. Supongamos que el ADN de nuestro interés, “insertar” es un fragmento de 400 libras que se inserta en el plásmido.
Vea el carril 2: el fragmento de 400 pb de nuestro inserto.
Diseñamos cebadores de flanqueo a 100 libras de distancia de ambos lados de la plaquita. Si la imprimación de flanqueo amplifica el ADN junto con el inserto, el producto es de 600 pb, consulte el carril 3 (400 pb de ADN del inserto + región de flanqueo de 200 pb).
Ahora, vea el carril 1, es un control positivo sin el inserto o un plásmido normal sin el ADN transformado. Por lo tanto, los cebadores de flanqueo solo amplifican 200 pb de ADN.
Véase el carril 1, fragmento de ADN de 200 pp sin inserción (control positivo).
Ahora, observe el carril 4. El carril 4 es el resultado de los cebadores específicos de orientación. La imprimación específica de orientación es una combinación de imprimación específica de inserción y imprimación específica de región de flanqueo.
Una imprimación de insert DNA y una imprimación de la imprimación específica de la región de flanqueo se seleccionan para la amplificación de la imprimación específica de la orientación.
Por lo tanto, se amplifica el fragmento de 100 pb de la imprimación de la región flanqueante y 400 pb del ADN de inserción y se observa un fragmento de ADN de 500 pb en el carril 4.
El carril 5 es el control específico del inserto que da un fragmento de ADN de 400 bp.
El carril 6 es el control negativo sin la plantilla. Mediante el control negativo se puede identificar cualquier contaminación. El tubo de reacción contiene todos los ingredientes excepto la plantilla. Así que idealmente no hay bandas de ADN presentes en este carril.
Si se observa una banda de ADN, la muestra está contaminada.
Ventajas de la PCR de colonias:
- La técnica es rápida y rentable.
- Además, la precisión y especificidad de la técnica son mayores.
- La configuración es simple al igual que la PCR convencional, la extracción de ADN y la purificación de plásmidos como pasos laboriosos no son necesarios.
- Sin necesidad de digestión de restricción para la identificación del ADN del inserto.
- Todo el experimento se puede completar en 90 minutos.
Desventajas de la PCR de colonias:
- El método es rentable, rápido y confiable, sin embargo, no se puede detectar ninguna mutación en el inserto.
- Además, la información de la secuencia no puede ser obtenida por la PCR de la colonia. necesitamos hacer secuenciación para la confirmación de la transformación del ADN.
- La probabilidad de resultados falsos positivos es alta.
Leer más;
- ¿Qué es una PCR múltiple?
Después de completar el experimento, la muestra se envía para la secuenciación donde se puede determinar la secuencia de ADN de nuestro interés.
incluso podemos hacer PCR múltiple combinando imprimaciones específicas de inserción y imprimaciones específicas de plásmidos.
Conclusión:
Aunque la PCR de colonia es la mejor opción para la identificación de la transferencia génica, la única técnica de PCR de colonia no es suficiente para interpretar los resultados. Es posible que algunas de las mutaciones presentes en el inserto no puedan ser detectadas por la PCR.
Para confirmar los resultados se requiere secuenciación de ADN. Después de determinar el orden de secuencia, podemos decir si nuestro gen de interés se inserta correctamente o no.