7.4: Complex III
Complex III accetta elettroni sia dal Complesso I che dal Complesso II. Gli elettroni arrivano sotto forma di ubiquinolo, UQH2, che fornisce due elettroni e due protoni e viene riconvertito in ubiquinone, UQ. L’ubiquinone torna quindi al Complesso I o al Complesso II per raccogliere più elettroni.
- Il Complesso III è la destinazione per gli elettroni che arrivano sia dal Complesso I che dal Complesso II.
- Il complesso III utilizza l’energia rilasciata nei trasferimenti di elettroni in discesa per pompare più protoni attraverso la membrana mitocondriale interna.
- Il gradiente protonico attraverso la membrana viene utilizzato per guidare la formazione di ATP al Complesso V.
- Il Complesso III invia quindi i suoi elettroni al Complesso IV.
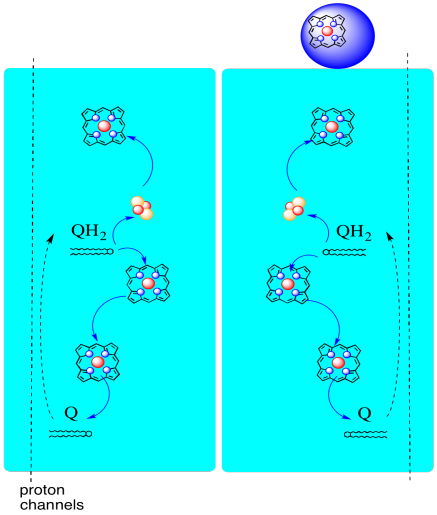
Il percorso degli elettroni attraverso il Complesso III è mostrato di seguito. Si noti che il complesso è una struttura dimerica, con due percorsi equivalenti: uno mostrato a sinistra e l’altro a destra. Gli elettroni vengono consegnati da UQH2, ubiquinolo, nel mezzo dell’immagine, viaggiando verso l’alto verso il citocromo c mobile in alto. Un secondo percorso di elettroni permette di viaggiare verso il basso nella foto, verso un’altra molecola di ubiquinone, che ricicla gli elettroni in un ciclo.
- Il complesso III presenta un insolito percorso di elettroni divergenti.
- Un elettrone viaggia verso il citocromo c.
- Il secondo elettrone viene infine riciclato in un altro ubichinolo.
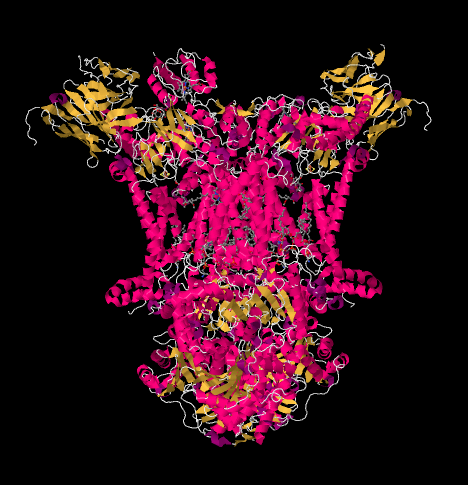
La struttura a raggi X del Complesso III è mostrata sotto forma di cartone animato. La membrana mitocondriale interna intersecherebbe la parte centrale superiore del complesso. La maggior parte dei progetti complessi sotto, nella matrice (la parte inferiore dell’immagine, in questo orientamento) anche se una quantità significativa sporge anche nello spazio intermembrana.
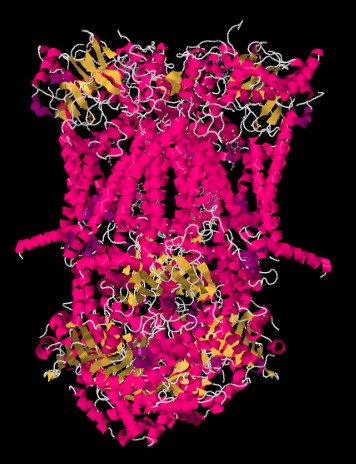
Il complesso III continua la catena di trasporto degli elettroni, inviando elettroni a potenziale di riduzione più elevato (e minore energia libera) e verso un incontro con l’ossigeno molecolare nel Complesso IV. I protoni vengono anche pompati dalla matrice mitocondriale, attraverso la membrana mitocondriale interna e nello spazio intermembrana. Allo stesso tempo, un secondo percorso di elettroni si traduce in un riciclaggio efficace della metà degli elettroni che entrano nel complesso, aumentando il numero di protoni pompati per elettrone che arriva al Complesso III.
- È stato suggerito che il ciclo di riciclaggio degli elettroni fornisce un meccanismo per disegnare più protoni dalla matrice.
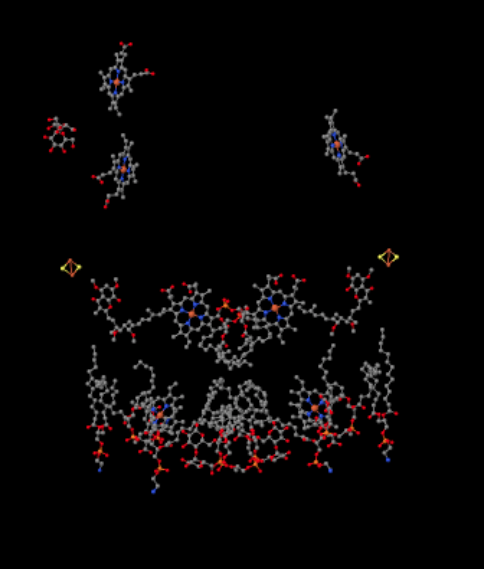
Possiamo vedere questa disposizione insolita se ignoriamo le proteine per rivelare i ligandi, sotto. Il complesso può essere visto più chiaramente come un dimero; la metà sinistra dell’immagine è esattamente la stessa della destra, anche se riflessa in modo opposto. Gli elettroni entrano nel complesso tramite il vettore mobile, ubiquinolo, visibile sul lato destro e sinistro dell’immagine circa a metà; puoi vedere l’anelloe una catena appesa ad esso.
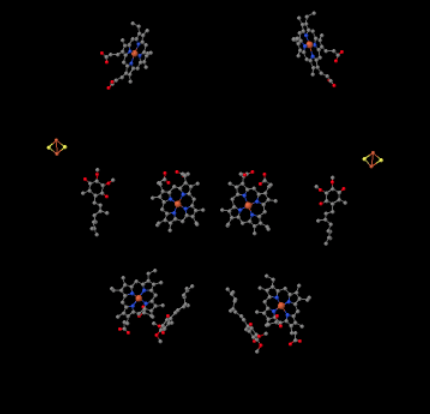
Quando UQH2 si lega al Complesso III, un elettrone viene inviato a un cluster FeS, mentre il secondo viene inviato a un centro eme di ferro, chiamato eme BL. Il cluster FeS è visibile appena sopra l’ubiquinolo e sul bordo dell’immagine. L’eme BL è verso il centro dell’immagine.
Sebbene non sia evidente nell’immagine a raggi X mostrata, questo cluster FeS è diverso da quelli che hai visto in precedenza. Questo è tenuto in posizione con diverse catene laterali di aminoacidi. Non puoi vederlo nella foto perché l’intera proteina è stata lasciata fuori, insieme a quegli amminoacidi che si legano al cluster. Mentre la maggior parte dei cluster FeS sono tenuti in posizione esclusivamente da cisteine, questo cluster è tenuto in posizione da due cistei da un lato, ma da due istidine dall’altro. Questo particolare cluster FeS è chiamato un cluster Rieske.
I cluster Rieske sono importanti perché sono “cluster FeS ad alto potenziale”. Ciò significa che hanno potenziali di riduzione eccezionalmente positivi rispetto ad altri cluster FeS. Il potenziale di riduzione di qualsiasi ligando in una proteina è altamente dipendente dal suo ambiente, e quindi c’è una vasta gamma di valori nella maggior parte dei casi. Questo è vero per i cluster di ferro zolfo; quelli normali hanno potenziali di riduzione che vanno da -1.0 V a circa + 0.05 V. I cluster Rieske hanno potenziali che vanno da circa 0V a circa 0.4 V.
Dal cluster FeS, l’elettrone viene inviato a un altro centro eme di ferro in un’unità legata alla membrana chiamata citocromo c1. Il citocromo c1 può essere visto nella parte superiore dell’immagine. Poiché questa immagine è orientata allo stesso modo delle altre strutture a raggi X del complesso respiratorio, possiamo vedere che questo elettrone viene trasportato verso lo spazio intermembrana. Infine, questo elettrone viene trasferito ad un altro eme, ma questo eme è legato in una piccola proteina mobile, chiamata citocromo c. Il citocromo c è un altro vettore elettronico mobile. Porta l’elettrone al complesso IV. Torneremo al citocromo c un po ‘ più tardi.
Prima di arrivare a questo, dove va l’altro elettrone? Dall’eme BL, il secondo elettrone viene passato ad un altro complesso di porfirina di ferro, l’eme BH. Questo è verso il fondo dell’immagine, quindi questo elettrone viene effettivamente rimandato verso la matrice. In attesa del BL eme, tuttavia, c’è un altro ubiquinone, UQ. Sta aspettando l’elettrone. In realtà aspetterà due, quindi lascerà il suo dock e tornerà al sito di legame ubiquinolo (UQH2) per consegnare di nuovo gli elettroni.
Perché preoccuparsi? Perché non basta inviare gli elettroni sulla loro strada correttamente la prima volta? Sembra che si stiano riciclando per qualche motivo. Si ritiene che questa deviazione consenta di pompare protoni aggiuntivi attraverso la membrana mitocondriale. Quando il secondo ubiquinone viene ridotto a ubiquinolo tramite l’accettazione di due elettroni, raccoglie anche altri due protoni dalla matrice sottostante. Quei protoni alla fine verranno passati verso lo spazio intermembrana quando l’ubiquinolo si ossida di nuovo. Questo riciclaggio, e spremere un paio di protoni in più per aumentare il gradiente protonico, è chiamato Q loop.
Il vantaggio del Q-loop è spiegato nel diagramma seguente. Nel diagramma, gli ingressi al complesso III sono mostrati in rosso, le uscite in blu e gli elementi riciclati in verde. Se un ubiquinolo consegnasse semplicemente i suoi elettroni e protoni e fosse fatto, ci sarebbero due protoni consegnati per ubiquinolo. Questo è un protone in uscita per elettrone che è stato immesso.
Un secondo ubiquinolo farebbe esattamente la stessa cosa. Ci sarebbero quattro protoni in uscita per quattro elettroni inizialmente in ingresso. Questo è ancora un protone in uscita per elettrone che è stato immesso.
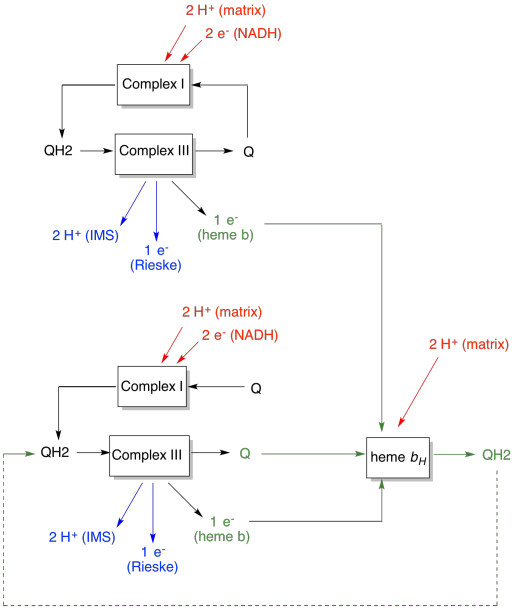
Se, invece, un elettrone viene riciclato ogni volta, allora ogni secondo ubiquinolo porta alla consegna di una coppia extra di protoni. Questo perché nel raccogliere gli elettroni riciclati, un ubiquinone ha dovuto tornare al lato matrice della membrana e raccogliere altri due protoni. Nel complesso, ciò significa che sei protoni vengono consegnati per quattro elettroni in ingresso, o 1,5 protoni in uscita per ingresso di elettroni. Poiché il gradiente protonico è ciò che sta generando l’ATP, aumentando il numero di protoni pompati per elettrone in arrivo, l’efficienza aumenta.
- Il Q-loop aumenta il numero di protoni pompati per ingresso di elettroni nel sistema.
Ora torniamo al vettore di elettroni in uscita, il citocromo c, che traghetterà gli elettroni sul Complesso IV. Nella foto qui sotto, da una diversa struttura cristallina a raggi X, il Complesso è stato trovato con un aggancio del cyctochrome c nel sito di legame. Il citocromo c è una proteina globulare rosa attaccata nella parte superiore dell’immagine, nella metà sinistra del dimero mentre la guardiamo qui. Si trova ai margini dello spazio intermembrana, che è un mezzo acquoso. A differenza dell’ubiquinone, che scivola lungo il doppio strato lipidico della membrana, il citocromo c rotola o scivola o nuota verso la sua destinazione.
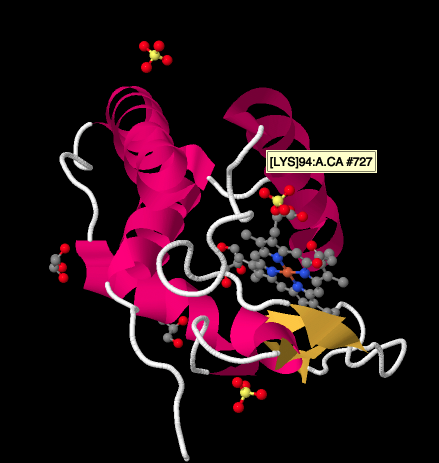
La stessa vista è mostrata sotto senza la proteina. La parte inferiore dell’immagine è piuttosto ingombra di lipidi, e c’è uno zucchero in giro in alto a sinistra, ma proprio in cima puoi vedere la porfirina di ferro che si trova all’interno del citocromo c.
Uno sguardo più da vicino a una struttura a raggi X di un ciclocromo c, sotto, mostra un’immagine relativamente semplice rispetto ai complessi che abbiamo osservato. Ci sono un paio di eliche, un paio di fogli, alcuni anelli e quel ferro porfirina o eme di nuovo. Alcuni ioni solfato galleggiano nelle vicinanze, così come un paio di molecole di glicerolo, probabilmente introdotte durante la purificazione o la cristallizzazione della proteina. Si noti che l’eme si trova sul bordo della proteina. Quella posizione può rendere più facile il trasferimento di elettroni dal Complesso III o dal complesso IV.
- Il citocromo c è la destinazione finale degli elettroni che si muovono attraverso il Complesso III.
- Il citocromo c è un vettore mobile a un elettrone.
- Il citocromo c è in realtà una piccola proteina idrofila.
- Il citocromo c si trova sopra la membrana mitocondriale, ai margini dello spazio intermembrana.
Cosa impedisce al ciclocromo c di vagare nello spazio intermembrana? Se lo facesse, interromperebbe il flusso efficiente di elettroni. È possibile che il citocromo c si muova avanti e indietro tra il complesso III e il Complesso IV tramite un meccanismo chiamato “sterzo”. Nello sterzo, il vettore viene guidato lungo un percorso tramite cariche complementari. Ad esempio, se la membrana è caricata negativamente a causa di gruppi polari sulla superficie della membrana, le cariche positive sulla superficie del citocromo c possono limitare il suo movimento alla superficie della membrana. Questo da solo ridurrebbe la sua mobilità da tre dimensioni (ovunque nello spazio intermembrana) a due dimensioni (ovunque sulla superficie della membrana). È possibile che interazioni aggiuntive riducano ulteriormente la sua mobilità a una traccia unidimensionale tra i due complessi.
- Lo sterzo, basato su cariche complementari, impedisce al citocromo c di perdersi nello spazio intermembrana.
Esercizio \(\PageIndex{1}\)
Suggerisca i residui possibili dell’amminoacido alla superficie del citocromo c che possono aiutarlo a rimanere su una superficie caricata negativamente della membrana.
Risposta
Arginina e lisina sono caricate positivamente a pH neutro.
Esercizio \(\PageIndex{2}\)
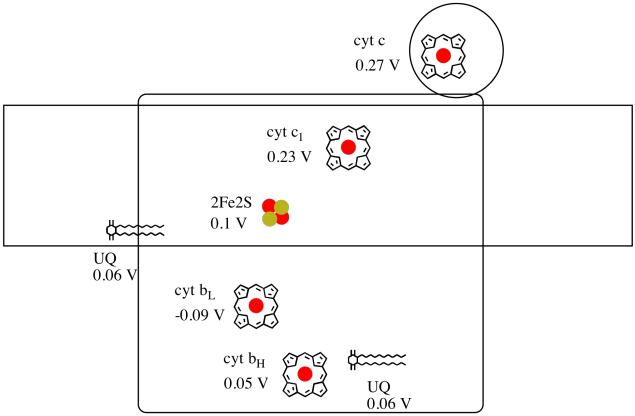
È difficile misurare il potenziale di riduzione di un singolo sito all’interno di una proteina. Tuttavia, i ricercatori sono stati in grado di stimare questi valori misurando gli spettri EPR in varie condizioni. Assumendo che il potenziale di riduzione di seguito, disegnare un progresso di reazione diagramma per il trasporto di un elettrone dall’iniziale ubichinone donatore tutto il modo di citocromo c.
 Risposta
Risposta
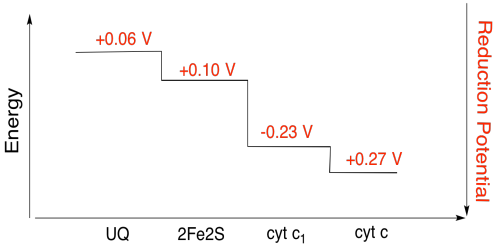
Esercizio di \(\PageIndex{3}\)
Utilizzando i valori riportati in figura, calcolare la variazione di energia quando un elettrone viene trasferito dal 2Fe2S cluster con il citocromo c1.
Risposta
Supponendo che il potenziale di riduzione sono:
2Fe2S(bue) + e- → 2Fe2S(rosso) Eored = 0.10 V
cyt c1(bue) + e- → cyt c1(rosso) Eored = 0.230 V
Quindi la differenza di potenziale per la reazione, ΔEo = 0.23 – (0.10) V = 0.13 V.
Faraday relazione ∆ G = – n F ΔEo dà
∆ G = – 1 x 96,485 J V-1 mol-1 x 0,13 V = 12,543 J mol-1 = 12.5 kJ mol-1
Esercizio \(\PageIndex{4}\)
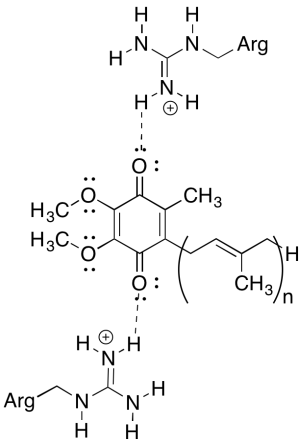
Una delle cose sconcertanti di questo complesso è che, nel ciclo di riciclaggio degli elettroni, il trasferimento di elettroni sembra passare da un ubiquinone iniziale a un ubiquinone finale, senza una forza trainante complessiva. Tuttavia, i potenziali di riduzione sono molto sensibili all’ambiente. Mostra come i residui di arginina nelle vicinanze potrebbero rendere più positivo il potenziale di riduzione di un ubiquinone.
Risposta
I residui di arginina positivi conferirebbero una carica positiva parziale all’ubiquinone attraverso il legame dell’idrogeno; l’ubiquinone avrebbe di conseguenza un potenziale di riduzione più positivo.
Vedere la pagina di messa a fuoco sulla struttura del complesso III a Biochimica Online.
Struttura cristallina a raggi X del Complesso III da: Gao, X., Wen, X., Esser, L., Quinn, B., Yu, L., Yu, C.-A., Xia, D. Basi strutturali per la riduzione del chinone nel complesso bc1: un’analisi comparativa delle strutture cristalline del citocromo mitocondriale bc1 con substrato legato e inibitori nel sito Qi. Biochimica 2003, 4, 9067-9080. (1NTZ)
Struttura cristallina a raggi X del Complesso III con citocromo c legato da: Solmaz, S. R., Hunte, C. Struttura del complesso III con citocromo c legato in stato ridotto e definizione di un’interfaccia core minima per il trasferimento elettronico. J. Biol. Chimica. 2008, 283, 17542-17549. (3CXH)
Struttura cristallina a raggi X del citocromo c da: Enguita, FJ, Pohl, E., Turner, DL, Santos, H., Carrondo, M. A. Prove strutturali per una via di trasferimento di protoni accoppiata con riduzione haem del citocromo c” da Methylophilus methylotrophus. J. Biol. Inorg. Chimica. 2006 11, 189. (1OAE)
Attribuzione
Chris P Schaller, Ph. D., (Collegio di San Benedetto / Università di San Giovanni)