Che cosa è PCR della colonia?
Colony PCR è un metodo PCR rapido e ad alto throughput per determinare la presenza o l’assenza del DNA inserito nel plasmide direttamente dalle colonie batteriche.
La clonazione molecolare è da tempo uno dei metodi più diffusi per la trasformazione del DNA. Tuttavia, per determinare la presenza o l’assenza dell’inserto di DNA dobbiamo eseguire esperimenti di trasformazione.
Colony PCR è un nuovo metodo in cui progettando i primer specifici del DNA inserito, possiamo identificare se il nostro DNA di interesse è inserito nel plasmide o meno.
Tuttavia, non è così semplice come stiamo discutendo.
In questo articolo, ci concentreremo sulla PCR colonia in particolare, il principio della PCR colonia, i suoi vantaggi e limitazioni.
Per questo, dobbiamo capire diversi termini e argomenti. Inizieremo il nostro argomento dalle basi. Il contenuto dell’articolo è,
Argomenti chiave:
“Un plasmide è il DNA circolare batterico che si replica indipendentemente dal cromosoma batterico e utilizzato nella manipolazione genica e nel trasferimento genico.”
La clonazione genetica è uno strumento genetico molecolare tradizionale utilizzato da tempo nei laboratori. In breve, nella clonazione del gene, il gene del nostro interesse viene inserito nel plasmide attraverso mezzi artificiali. Questo DNA è replicato indipendentemente dal cromosoma batterico.
I plasmidi sono in realtà, utilizzati per generare molte copie di brevi segmenti di DNA. Poiché i batteri si replicano più velocemente di qualsiasi altro organismo, possiamo generare molte copie del gene di nostro interesse inserendolo nel plasmide batterico.
F-plasmid, Col-plasmid, degradative – plasmid e resistenza plasmid sono diversi tipi comuni di plasmidi presenti nei batteri.
Inoltre, il plasmide può funzionare come un vettore molecolare che trasferisce brevi segmenti di DNA da una cellula all’altra.
Abbiamo trattato un incredibile articolo di approfondimento sul DNA plasmidico. Leggi qui: DNA plasmidico-Struttura, funzione, isolamento e applicazioni.
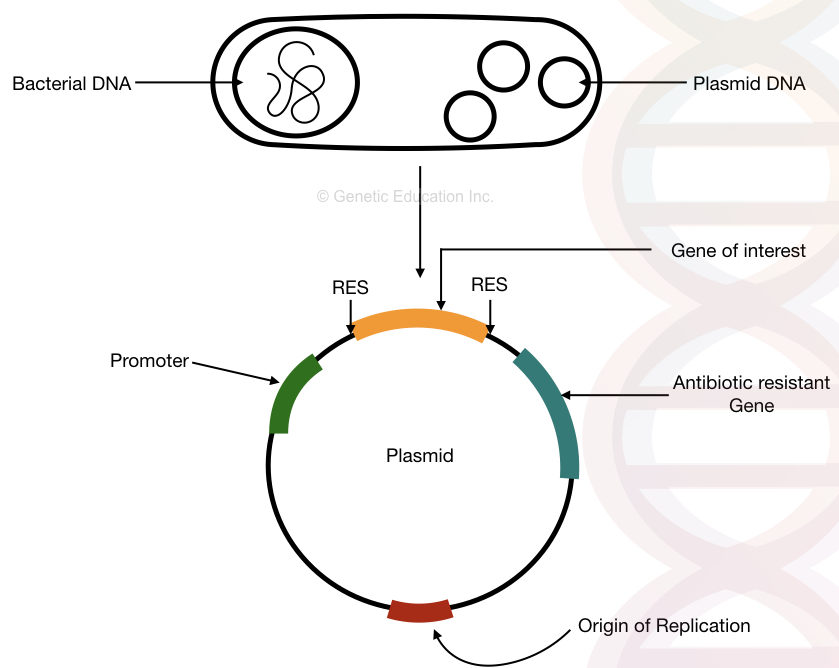
La struttura del DNA plasmidico batterico con, l’origine della replicazione, il gene della resistenza agli antibiotici, il promotore e il gene di interesse.
Oltre ai batteri, molti altri procarioti contengono anche DNA plasmidico. La funzione principale del plasmide nei batteri è per la loro sopravvivenza nelle condizioni difficili.
Poiché il plasmide trasferisce il gene di nostro interesse, è molto importante determinare se il nostro gene di interesse è inserito o meno nel plasmide.
Per questo, possiamo utilizzare diversi metodi come la PCR e la coltura microbica.
Il platting delle colonie richiede più tempo e anche la sensibilità del metodo non è buona. La probabilità della contaminazione è sempre alta nei metodi di coltura batterica.
Quindi i risultati non sono accurati.
La nostra PCR aiuta anche qui. Utilizzando il metodo PCR colonia, un inserto di DNA può essere determinato o identificato.
- Come impostare un laboratorio di estrazione del DNA: una guida completa (prodotti chimici, strumenti e altre utilità).
- Delezione del cromosoma 6p: una ragione per nessun dolore, nessuna fame e nessun sonno
Che cos’è la PCR delle colonie?
Colony PCR è la modifica della PCR convenzionale in cui le colonie batteriche vengono utilizzate direttamente come modello di PCR.
Il DNA plasmidico che contiene il DNA di nostro interesse è amplificato nelle condizioni dipendenti dalla temperatura ciclica.
La rappresentazione grafica della PCR colonia è mostrato nella figura seguente,
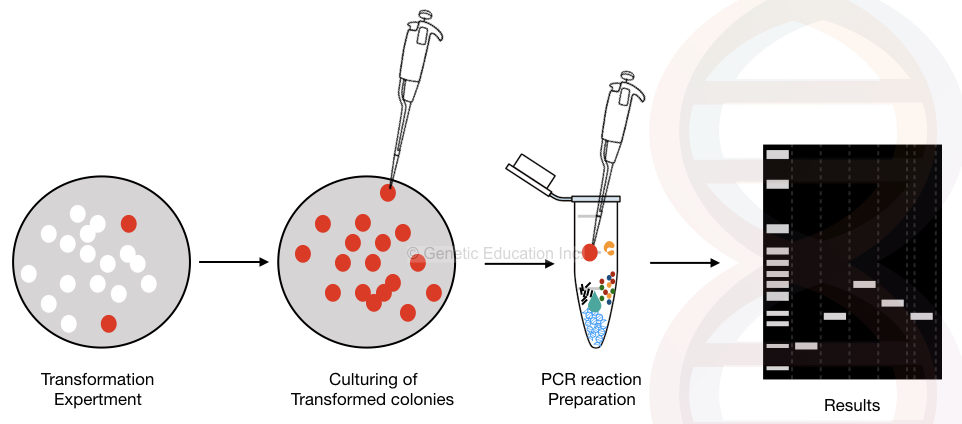
La panoramica generale del metodo PCR colony.
Principio di PCR colonia:
La colonia batterica contenente il plasmide può essere amplificata direttamente utilizzando due serie di primer. I primer specifici dell’inserto che amplificano la sequenza di inserimento e i primer fiancheggianti specifici del vettore, che amplificano il DNA plasmidico diverso dal DNA inserito (regioni fiancheggianti su entrambi i lati dell’inserto).
Utilizzando l’inserto che fiancheggia i primer (che amplifica il resto del DNA) è possibile determinare la dimensione del nostro inserto di DNA.
Una colonia batterica viene prelevata e aggiunta direttamente nella mastermix contenente tutti i reagenti PCR. Inoltre, aggiungendo una fase di riscaldamento iniziale alla PCR, il DNA plasmidico esce dalla cellula batterica e amplificato nella reazione.
Questo è il principio di base della PCR colonia, Tuttavia, può essere modificato a seconda delle esigenze.
Il protocollo di colony PCR:
La colony PCR è una delle modifiche eccellenti della PCR convenzionale. Invece del DNA modello, le colonie batteriche vengono aggiunte direttamente alla reazione. Oltre a questo, Taq DNA polimerasi, primer, tampone di reazione PCR e DD / W sono aggiunti nella reazione PCR troppo.
Qui nella PCR della colonia, la selezione dei primer è molto importante. Inoltre, la selezione dei primer dipende dall’obiettivo del nostro esperimento.
Che tipo di informazioni vogliamo dal nostro esperimento colony PCR?
-
-
- Informazioni sulla presenza o assenza del solo inserto.
- Informazioni sulla dimensione dell’inserto.
- Informazioni sull’orientamento dell’inserto.
-
A seconda che diversi primer PCR sono progettati per la PCR colonia.
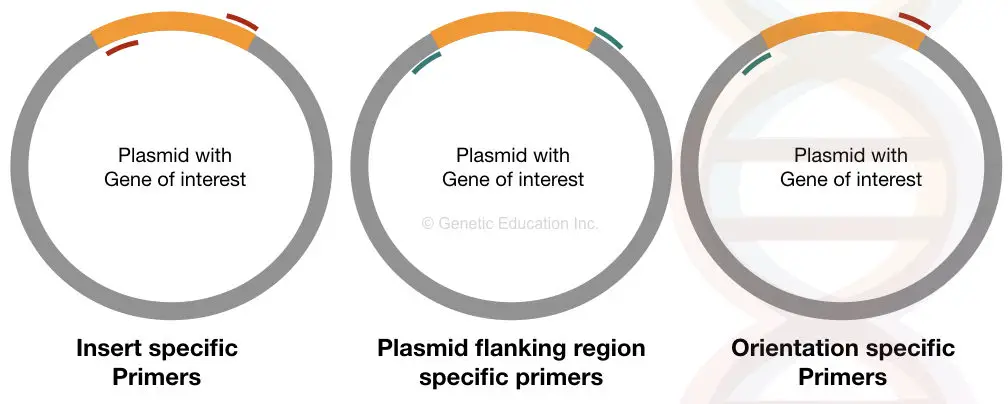
I primer specifici dell’inserto si legano alla posizione specifica su entrambi i lati del DNA inserito di nostro interesse. Se viene trasferito correttamente nel plasmide, questi primer possono legarsi ad esso altrimenti non può essere in grado di legarsi.
Questo set di primer fornisce informazioni sulla presenza o assenza dell’inserto.
I primer specifici per l’orientamento sono primer unici in cui un primer si lega all’interno dell’inserto e un altro primer si lega alla sequenza del DNA plasmidico (sequenza diversa dal DNA dell’inserto).
Questo set di primer fornisce informazioni sull’orientamento del DNA inserito di nostro interesse. Se il nostro DNA dell’inserto non è correttamente legato nel vettore, il primer specifico di quel lato della sequenza non può legarsi e non otterremo l’amplificazione.
Anche i primer specifici per plasmidi sono importanti quanto i primer specifici per l’orientamento. Questo set di primer è progettato dalla regione di fianco dell’inserto che si lega all’esterno del DNA di nostro interesse.
Questo set di primer aiuta a determinare la dimensione dell’inserto. Espande regioni diverse dal DNA dell’inserto.
La reazione di PCR per l’esecuzione di the colony PCR è come seguito,
| il Componente | Concentrazione | Quantità |
| Master mix (Speciale
per la Colonia di PCR) |
1X | 12µL |
| Reazione di PCR buffer
da 2 mm di MgCl2* |
1X | 5 µl |
| Forward primer | 10 | 1µL |
| Reverse primer | 10 | 1µL |
| Il surnatante | 3µL | |
| Acqua | 3µL | |
| Totale | ——— | 25µL |
La procedura di colony PCR:
Bene, il colony PCR non ha bisogno di DNA estratto.
Non stiamo estraendo il DNA qui. Invece, vengono utilizzati diversi altri metodi per aumentare la sensibilità della reazione.
Ok, perché non stiamo estraendo il DNA per il DNA plasmidico?
Poiché la ragione è semplice, la membrana cellulare della cellula batterica è molto liscia.
Avevamo già discusso la membrana cellulare della cellula batterica. Leggi qui: Diversi tipi di metodi di estrazione del DNA
Un batterio contiene una membrana a cellule molli che può essere facilmente lisata riscaldandola o centrifugandola ad alta velocità.
Inoltre, non abbiamo bisogno del DNA batterico. Il plasmide circolare circolante è presente nel citoplasma dei batteri, pertanto non sono necessari ulteriori passaggi di purificazione. Rompendo la membrana cellulare, il nostro DNA modello è pronto per l’amplificazione.
Ok, consente di passare rapidamente attraverso il metodo per ottenere un buon DNA plasmidico.
Con l’aiuto del selettore sterile, selezionare diverse colonie batteriche e trasferirle nel tubo di Eppendorf.
Ora aggiungere TE buffer in esso e mescolare bene. Puoi usare anche D / W.
Riscaldare il campione nel bagno di acqua bollente per 20 minuti.
Delicatamente vertice esso.
Centrifugare il campione ad alta velocità per 2 minuti.
Trasferire il surnatante in un’altra provetta e utilizzarlo come DNA modello.
Nella reazione viene aggiunto un campione da 20 µl.
Ulteriori informazioni:
Perché surnatante e non pellet?
Il DNA è una biomolecola della vita. Il DNA plasmidico è ancora più piccolo del DNA nucleare batterico. Contiene solo diversi geni fino a 1000bp a 20.000 bp.
Quindi solo centrifugandolo, il DNA plasmidico più leggero esce dalla cellula e si deposita nel surnatante mentre il pellet contiene proteine e DNA nucleare, quindi non lo stiamo usando.
Ora veniamo al punto.
Il nostro plasmide è pronto per l’amplificazione.
In un altro metodo,
Utilizzare direttamente la colonia batterica.
Questo metodo è una combinazione di PCR Hotstart e PCR colony.
Le colonie batteriche vengono raccolte e aggiunte al tubo di reazione PCR.
I tubi vengono inseriti nella macchina PCR. Viene aggiunto un ulteriore passaggio di riscaldamento.
Riscaldandolo da 5 a 7 minuti, il DNA plasmidico esce dalla cellula.
Ora i primer specifici dell’inserto amplificano il DNA che abbiamo inserito. E i primer laterali amplificano il resto del DNA.
L’amplificazione viene eseguita per 20-25 cicli. Il ciclismo condizioni per la colonia di PCR è elencato di seguito,
| punti di PCR | Iniziale Denaturazione | Denaturazione | Ricottura | Estensione | estensione Finale |
| Temperatura | 95 ° C | 95 ° C | 55-65 C | 72 C | 72 C |
| Tempo | 3 | 10 sec | 45 sec | 50 sec | 5 min |
| ——- | ——- | 25 cicli | —– | ——- |
Leggi l’interessante articolo sulla PCR convenzionale: UNA Guida Completa della Reazione a Catena della Polimerasi
Suggerimenti per il miglioramento:
Utilizzare solo un paio di colonie, come molte colonie di aumentare la probabilità di non attacchi specifici.
Utilizzare controllo positivo e contol negativo.
Come controllo positivo utilizzato il primer fiancheggiamento anche se l’inserto non è presente, la reazione PCR dà banda di DNA del DNA plasmide che indica che la reazione che abbiamo preparato è corretta.
Come controllo negativo, utilizzare il plasmide non trasformato (plasmide senza DNA di inserimento), questo DNA plasmidico viene amplificato solo se l’inserto è presente.
Poiché un inserto utilizza sequenze di DNA brevi, sequenze di DNA più lunghe aumentano la possibilità di attacchi non specifici e fallimento della reazione PCR.
Inoltre, utilizzare programmi PCR più brevi.
L’applicazione principale della PCR della colonia è nell’identificazione della legatura corretta e dell’inserzione del DNA dell’inserzione nei batteri come pure nel plasmide del lievito.
Dopo il completamento della reazione PCR della colonia, i prodotti PCR vengono eseguiti sul gel di agarosio al 2%. I risultati dell’esperimento sono mostrati nella figura seguente,
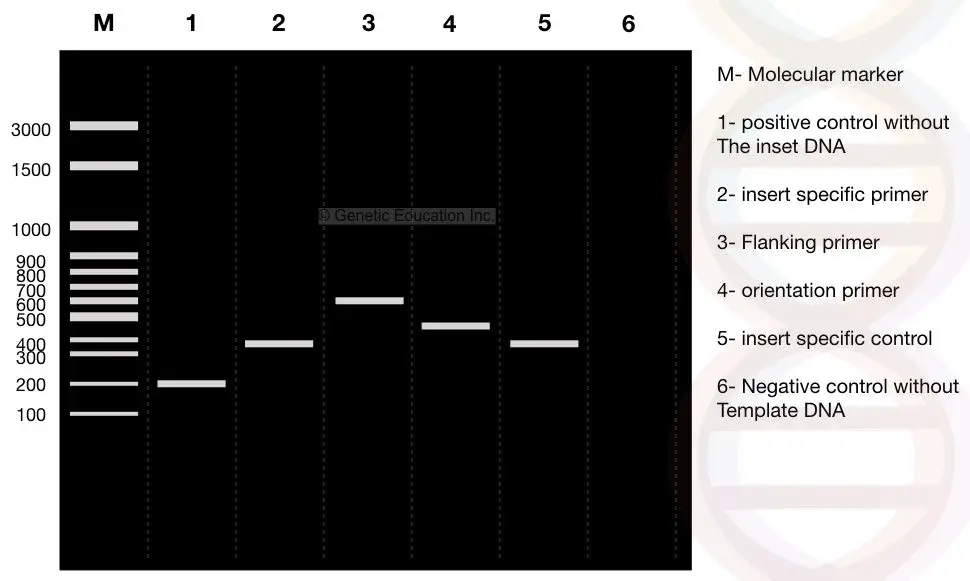
Ora osservare attentamente i risultati, il M è il marcatore molecolare del DNA 3000bp. Supponiamo, il DNA di nostro interesse, “insert” è un frammento 400bp che viene inserito nel plasmide.
Vedere la corsia 2: il frammento di 400 bp del nostro inserto.
Abbiamo progettato i primer fiancheggianti 100bp lontano da entrambi i lati dell’inserto. Se il primer fiancheggiante amplifica il DNA insieme all’inserto, il prodotto è 600 bp, vedere la corsia 3 (400 bp di DNA inserto + 200 bp regione fiancheggiante).
Ora, vedi corsia 1, è un controllo positivo senza l’inserto o un plasmide normale senza il DNA trasformato. Quindi i primer laterali amplificano solo 200 bp di DNA.
Vedere corsia 1, 200bp frammento di DNA senza inserto (controllo positivo).
Ora, osservare la corsia 4. La corsia 4 è il risultato dei primer specifici per l’orientamento. Il primer specifico per l’orientamento è una combinazione di primer specifico per l’inserto e primer specifico per la regione di fiancheggiamento.
Per l’amplificazione del primer specifico per l’orientamento vengono selezionati un primer dell’inserto DNA e un primer del primer specifico per la regione di fiancheggiamento.
Pertanto, il frammento di 100 bp dal primer della regione di fiancheggiamento e 400bp dal DNA dell’inserto viene amplificato e il frammento di DNA di 500 bp viene osservato nella corsia 4.
Corsia 5 è il controllo specifico inserto che dà 400 bp frammento di DNA.
Corsia 6 è il controllo negativo senza il modello. Utilizzando il controllo negativo è possibile identificare qualsiasi contaminazione. Il tubo di reazione contiene tutti gli ingredienti tranne il modello. Quindi idealmente nessuna banda di DNA è presente in questa corsia.
Se si osserva una banda di DNA, il campione è contaminato.
Vantaggi di colony PCR:
- La tecnica è rapida ed economica.
- Inoltre, l’accuratezza e la specificità della tecnica sono più elevate.
- La configurazione è semplice proprio come la PCR convenzionale, l’estrazione del DNA e la purificazione del plasmide come passaggi laboriosi non sono necessari.
- Nessuna necessità di digestione restrittiva per l’identificazione del DNA dell’inserto.
- L’intero esperimento può essere completato entro 90 minuti.
Svantaggi della PCR colonia:
- Il metodo è economico, veloce e affidabile, tuttavia, qualsiasi mutazione nell’inserto non può essere rilevata.
- Inoltre, le informazioni sulla sequenza non possono essere ottenute dalla PCR della colonia. dobbiamo fare il sequenziamento per la conferma della trasformazione del DNA.
- La probabilità di risultati falsi positivi è alta.
Per saperne di più;
- Che cos’è una PCR multiplex?
Dopo il completamento dell’esperimento, il campione viene inviato per il sequenziamento in cui è possibile determinare la sequenza di DNA di nostro interesse.
possiamo anche fare PCR multiplex combinando sia primer specifici per inserti che primer specifici per plasmidi.
Conclusione:
Sebbene la PCR di colonia sia la scelta migliore per l’identificazione del trasferimento genico, l’unica tecnica PCR di colonia non è sufficiente per interpretare i risultati. Potrebbe possibile che alcune delle mutazioni presenti nell’inserto, che non possono essere rilevati dalla PCR.
Per confermare i risultati è richiesto il sequenziamento del DNA. Dopo aver determinato l’ordine di sequenza possiamo dire se il nostro gene di interesse è inserito correttamente o meno.