L’Istituto per la ricerca sulla creazione
 Uno degli argomenti più popolari usati per gli esseri umani che presumibilmente si evolvono dalle scimmie è noto come fusione cromosomica. L’impulso per questo concetto è il problema evolutivo che le scimmie hanno una coppia extra di cromosomi-gli esseri umani hanno 46 mentre le scimmie hanno 48. Se gli esseri umani si sono evoluti da una creatura simile a una scimmia solo tre o sei milioni di anni fa, un semplice blip nel grande schema della storia evolutiva, perché gli esseri umani e le scimmie hanno questa discrepanza?
Uno degli argomenti più popolari usati per gli esseri umani che presumibilmente si evolvono dalle scimmie è noto come fusione cromosomica. L’impulso per questo concetto è il problema evolutivo che le scimmie hanno una coppia extra di cromosomi-gli esseri umani hanno 46 mentre le scimmie hanno 48. Se gli esseri umani si sono evoluti da una creatura simile a una scimmia solo tre o sei milioni di anni fa, un semplice blip nel grande schema della storia evolutiva, perché gli esseri umani e le scimmie hanno questa discrepanza?
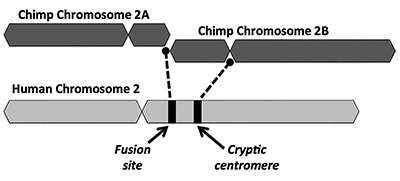
La soluzione evolutiva propone che una fusione end-to-end di due piccoli cromosomi simili a scimmie (denominati 2A e 2B) producesse il cromosoma umano 2 (Figura 1). Il concetto di fusione è nato nel 1982 quando gli scienziati hanno esaminato le somiglianze dei cromosomi umani e delle scimmie al microscopio. Mentre la tecnica era un po ‘ grezzo, è stato sufficiente per ottenere l’idea di andare.1
Il cosiddetto sito di fusione
La prima firma reale del DNA di un possibile evento di fusione è stata scoperta nel 1991 sul cromosoma umano numero 2.2 I ricercatori hanno trovato un piccolo ammasso confuso di sequenze terminali simili a telomeri che assomigliavano vagamente a una possibile fusione. I telomeri sono una sequenza a sei basi delle lettere del DNA TTAGGG ripetute più e più volte alle estremità dei cromosomi.
Tuttavia, la firma di fusione era un po ‘ un enigma basato sulle fusioni reali che occasionalmente si verificano in natura. Tutte le fusioni documentate negli animali viventi coinvolgono un tipo specifico di sequenza chiamata DNA satellite (satDNA) situato nei cromosomi e trovato in rotture e fusioni.3-5 La firma di fusione sul cromosoma umano 2 mancava questo SATDNA rivelatore.6
Un altro problema è la piccola dimensione del sito di fusione, che è lungo solo 798 lettere di DNA. Le sequenze di telomeri alle estremità dei cromosomi sono lunghe da 5.000 a 15.000 basi. Se due cromosomi erano fusi, si dovrebbe vedere una firma telomero fuso di 10.000 a 30.000 basi lungo-non 798.
Non solo le piccole dimensioni sono un problema per la storia della fusione, ma la firma non rappresenta davvero una fusione chiara di telomeri. La figura 2 mostra le lettere del DNA del sito di fusione a 798 basi con le sequenze di telomeri intatte a sei basi (lettera del DNA) enfatizzate in grassetto. Quando la sequenza di fusione viene confrontata con quella di una firma di fusione incontaminata della stessa dimensione, è solo il 70% identico nel complesso.

I ricercatori laici hanno sottolineato questa discrepanza e hanno etichettato il sito di fusione come significativamente “degenerato.”7 Dato il modello teorico standard dell’evoluzione umana, dovrebbe essere identico dal 98 al 99%, non al 70%. I ricercatori che descrivono questa scoperta hanno commentato: “Gli array testa a testa di ripetizioni nel sito di fusione sono degenerati in modo significativo (14%) dagli array quasi perfetti di (TTAGGG)n trovati nei telomeri” e hanno posto la domanda pertinente “Se la fusione si è verificata all’interno degli array di ripetizione telomerici inferiori a ~6 Mya, perché gli array nel sito di fusione”7 Va notato che la degenerazione del 14% citata dagli autori si riferisce alla corruzione delle sole sequenze a sei basi stesse, non dell’intero 798 basi.
Il sito di fusione all’interno di un gene?
La più notevole scoperta anti-evolutiva sul sito di fusione si è rivelata essere la sua posizione e ciò che effettivamente fa. Questa scoperta è avvenuta mentre stavo leggendo il documento di ricerca che riportava un’analisi dettagliata di 614.000 basi della sequenza di DNA che circonda il presunto sito di fusione. Ho notato in una delle figure che il sito di fusione si trovava all’interno di un gene, e sorprendentemente questa stranezza non è stata nemmeno riconosciuta nel testo del documento.8
Un risultato come questo è altamente degno di nota. Forse questa informazione sarebbe stata il chiodo nella bara evolutiva, per così dire, motivo per cui i ricercatori hanno rifiutato di discuterne. Questa grande anomalia mi ha ispirato a dare al sito di fusione un esame molto più attento. Questo documento è stato pubblicato nel 2002, e ho preso atto di esso nel 2013. Un’enorme quantità di dati sulla struttura e la funzione del genoma umano era stata pubblicata nel frattempo, e probabilmente c’era molto di più nella storia che doveva essere scoperta.
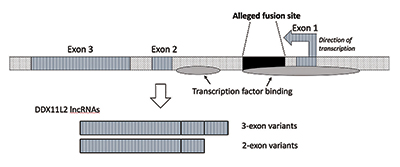
Quando ho eseguito ulteriori ricerche, ho verificato che il sito di fusione era posizionato all’interno di un gene dell’RNA elicasi ora chiamato DDX11L2. La maggior parte dei geni in piante e animali hanno i loro segmenti di codifica in pezzi chiamati esoni in modo che possano essere alternativamente giuntati. Sulla base dell’aggiunta o dell’esclusione di esoni, i geni possono produrre una varietà di prodotti. Le regioni intermedie tra gli esoni sono chiamate introni, che spesso contengono una varietà di segnali e interruttori che controllano la funzione genica. Il presunto sito di fusione è posizionato all’interno del primo introne del gene DDX11L2 (Figura 3).9
La molecola di DNA è a doppio filamento, con un filo più e un filo meno. È stato progettato in questo modo per massimizzare la densità delle informazioni aumentando al contempo l’efficienza e la funzionalità. Di conseguenza, ci sono geni che corrono in direzioni diverse sui fili opposti. Come risulta, il gene DDX11L2 è codificato sul filo meno. Poiché i geni nell’uomo sono come i coltelli dell’esercito svizzero e possono produrre una varietà di RNA, nel caso del gene DDX11L2 produce varianti brevi composte da due esoni e varianti lunghe con tre (Figura 3).9
Il sito di fusione è un promotore genico
Cosa potrebbe fare questo gene DDX11L2? La mia ricerca ha mostrato che è espressa in almeno 255 diversi tipi di cellule o tessuti.9 È anche co-espresso (acceso allo stesso tempo) con una varietà di altri geni ed è collegato a processi associati alla segnalazione cellulare nella matrice extracellulare e nella produzione di cellule del sangue. La posizione della cosiddetta sequenza di fusione all’interno di un gene funzionale associato alla genetica di una varietà di processi cellulari confuta fortemente l’idea che sia il sottoprodotto accidentale di una fusione telomerica testa a testa. I geni non sono formati da fusioni cromosomiche catastrofiche!
Ancora più sorprendente è che il sito fusion è di per sé funzionale e serve uno scopo ingegnerizzato importante. Il sito in realtà agisce come un interruttore per il controllo dell’attività genica. A questo proposito, una ricchezza di dati biochimici ha mostrato che 12 diverse proteine chiamate fattori di trascrizione regolano questo segmento del gene. Uno di questi non è altro che l’RNA polimerasi II, l’enzima principale che copia le molecole di RNA dal DNA in un processo chiamato trascrizione. Ulteriore supporto di questa scoperta è il fatto che l’effettivo processo di trascrizione inizia all’interno della regione del cosiddetto sito di fusione.
Tecnicamente, definiremmo l’attività nel presunto sito di fusione una regione promotrice. I promotori sono i principali interruttori all’inizio dei geni che li accendono e sono anche dove l’RNA polimerasi inizia a creare un RNA. Molti geni hanno promotori alternativi come il gene DDX11L2.
Ci sono in realtà due aree di legame del fattore di trascrizione nel gene DDX11L2. Il primo è nel promotore direttamente di fronte al primo esone e il secondo è nel primo introne corrispondente alla sequenza del sito di fusione. Non solo il gene DDX11L2 stesso è controllato in modo complesso, con la presunta sequenza di fusione che gioca un ruolo chiave, ma anche i trascritti di RNA prodotti sono molto intricati. Gli RNA stessi contengono un’ampia varietà di siti di legame e controllo per una classe di piccole molecole regolatrici chiamate microRNA.9
Le sequenze di telomeri interni funzionali sono in tutto il genoma
La presenza di sequenze di telomeri situate internamente si trova in tutto il genoma umano. Queste ripetizioni telomeriche apparentemente fuori luogo sono state soprannominate telomeri interstiziali. La presenza di queste sequenze presenta un’altra sfida per l’idea del sito fusion. È un dato di fatto che pochissime delle ripetizioni di telomeri nel sito di fusione si verificano in tandem. Come indicato nella Figura 2, la sequenza del sito di fusione a 798 basi contiene solo alcuni casi in cui due ripetizioni sono effettivamente in tandem e nessuna che ha tre ripetizioni o più. Tuttavia, ci sono molti altri siti di telomeri interstiziali in tutto il genoma umano in cui le ripetizioni si verificano in perfetto tandem da tre a dieci volte o più.10-11
Anche oltre al loro ruolo alle estremità dei cromosomi, sembra che le ripetizioni telomeriche interstiziali possano svolgere un’importante funzione nel genoma correlata all’espressione genica. In un recente progetto di ricerca, ho identificato le ripetizioni di telomeri in tutto il genoma umano e poi ho intersecato le loro posizioni genomiche con una diversità di set di dati contenenti informazioni biochimiche funzionali per l’attività genica.12 Letteralmente migliaia di ripetizioni telomeriche interne attraverso il genoma sono stati direttamente associati con le caratteristiche di espressione genica. Lo stesso tipo di legame del fattore di trascrizione e di attività genica che si verificano nel presunto sito di fusione si è verificato anche a livello genomico in numerose altre ripetizioni telomeriche interstiziali. Chiaramente, queste caratteristiche del DNA non sono incidenti di evoluzione, ma volutamente e intelligentemente progettato codice funzionale.
Centromero criptico fasullo all’interno di un gene
Un altro problema chiave con il modello di fusione è la mancanza di prove valide per una firma di una regione centromerica extra. I centromeri sono sezioni di cromosomi, spesso in posizioni centrali, che svolgono ruoli chiave durante la divisione cellulare. Come illustrato nella Figura 1, il cromosoma chimerico appena formato avrebbe avuto due siti centromeri immediatamente dopo la presunta fusione testa a testa dei due cromosomi. In tal caso, uno dei centromeri sarebbe funzionale mentre l’altro sarebbe disabilitato. La presenza di due centromeri attivi è una cattiva notizia per i cromosomi e porterebbe a disfunzioni e distruzione cellulare.
È interessante notare che l’evidenza di un centromero criptico (disabilitato) sul cromosoma umano 2 è ancora più debole di quella di un sito di fusione ricco di telomeri. Gli evoluzionisti spiegano la mancanza di un centromero secondario non funzionale chiaramente distinguibile sostenendo che un secondo centromero sarebbe stato rapidamente selezionato contro. Dopo di che, il centromero disabile si sarebbe deteriorato nel tempo poiché non c’erano più restrizioni funzionali poste su di esso facendo qualcosa di utile nel genoma.
Tuttavia, l’evidenza di un secondo centromero residuo in qualsiasi stadio della degenerazione della sequenza è problematica per il paradigma evolutivo. Le sequenze centromeriche funzionali sono composte da un tipo ripetitivo di DNA chiamato sequenze alfoidi, con ciascuna ripetizione alfoide lunga circa 171 basi. Alcuni tipi di ripetizioni alfoidi si trovano in tutto il genoma, mentre altri sono specifici per centromeri. La struttura delle sequenze trovate nel sito del centromero criptico sul cromosoma umano 2 non corrisponde a quelle associate ai centromeri umani funzionali.13 Ancora peggio per il modello evolutivo è che non hanno controparti altamente simili nel genoma scimpanzé – sono specifici per l’uomo.13
Il presunto centromero fossile è anche eccezionalmente piccolo rispetto a uno reale. La dimensione di un centromero umano normale varia in lunghezza tra 250.000 e 5.000.000 di basi.14 Il presunto centromero criptico è lungo solo 41.608 basi, ma è anche importante notare che ci sono tre diverse regioni di esso che non sono nemmeno ripetizioni alfoidi.15 Due di questi sono chiamati retroelementi, con uno è un LPA3 / LINEA ripetere 5.957 basi lunghe e l’altro un elemento SVA-E con 2.571 basi. Quando sottraiamo le inserzioni di queste sequenze non alfoidi, dà una lunghezza di sole 33.080 basi, che è una frazione della lunghezza di un centromero reale.
Il problema evolutivo più grave con l’idea di un centromero fossile, però, è che, come il presunto sito di fusione, è posizionato all’interno di un gene. Il presunto centromero criptico si trova all’interno del gene ANKRD30BL e la sua sequenza abbraccia sia le regioni di introne che di esone del gene.12,15
Infatti, la parte della presunta sequenza di centromeri fossili che atterra all’interno di un esone codifica in realtà per gli amminoacidi nella proteina del gene risultante. Il gene produce una proteina che si ritiene coinvolta nell’interazione della rete strutturale di proteine all’interno della cellula chiamata citoscheletro in connessione con le proteine del recettore incorporate nella membrana cellulare.16 Il fatto che il cosiddetto centromero fossile o criptico sia una regione funzionale all’interno di un importante gene che codifica le proteine confuta completamente l’idea che si tratti di un centromero defunto.
Conclusione: Nessuna fusione
A causa delle firme confuse e delle piccole dimensioni dei presunti siti di fusione e centromeri fossili, è altamente discutibile che la loro sequenza sia stata derivata evolutivamente da un’antica fusione cromosomica. Non solo, rappresentano la sequenza funzionale all’interno dei geni. Il presunto sito di fusione è un importante interruttore genetico chiamato promotore all’interno del gene RNA non codificante lungo DDX11L2, e il cosiddetto centromero fossile contiene sia la codifica che la sequenza non codificante all’interno di un grande gene codificante per proteine a ripetizione ankyrin.
Questo è un innegabile doppio smacco contro l’intera idea di fusione mitica, distruggendo completamente la sua validità. La conclusione scientifica schiacciante è che la fusione non è mai avvenuta.
- Yunis, J. J. e O. Prakash. 1982. L’origine dell’uomo: un’eredità pittorica cromosomica. Scienza. 215 (4539): 1525-1530.
- Ijdo, J. W. et al. 1991. Origine del cromosoma 2 umano: Una fusione ancestrale telomero-telomero. Atti dell’Accademia Nazionale delle Scienze. 88 (20): 9051-9055.
- Chaves, R. et al. 2003. Analisi citogenetica molecolare e organizzazione satellite centromerica di una nuova traslocazione 8;11 nelle pecore: un possibile intermedio nell’evoluzione cromosomica biarmata. Genoma dei mammiferi. 14 (10): 706-710.
- Tsipouri, V. et al. 2008. Analisi di sequenza comparativa rivelano siti di fusioni cromosomiche ancestrali nel genoma indiano muntjac. Biologia del genoma. 9 (10): R155.
- Adega, F., H. Guedes-Pinto e R. Chaves. 2009. DNA satellite nell’evoluzione del cariotipo degli animali domestici-Considerazioni cliniche. Ricerca citogenetica e genomica. 126 (1-2): 12-20.
- Tomkins, J. P. e J. Bergman. 2011. Telomeri: implicazioni per l’invecchiamento e prove per la progettazione intelligente. Diario della Creazione. 25 (1): 86-97.
- Fan, Y. et al. 2002. Struttura genomica ed evoluzione del sito di fusione cromosomica ancestrale in 2q13-2q14.1 e regioni paralogiche su altri cromosomi umani. Ricerca sul genoma. 12 (11): 1651-1662.
- Fan, Y. et al. 2002. Contenuto genico e funzione del sito di fusione cromosomica ancestrale nel cromosoma umano 2q13-2q14.1 e regioni paralogiche. Ricerca sul genoma. 12 (11): 1663-1672.
- Tomkins, J. P. 2013. Il presunto cromosoma umano 2″ Sito di fusione ” codifica un dominio di legame attivo del DNA all’interno di una fusione complessa e altamente espressa che nega il gene. Risposte Rivista di ricerca. 6: 367-375.
- Azzalin, C. M., S. G. Nergadze e E. Giulotto. 2001. Ripetizioni telomeriche intracromosomiali umane: organizzazione delle sequenze e meccanismi di origine. Cromosoma. 110: 75-82.
- Ruiz-Herrera, A. et al. 2008. Ripetizioni telomeriche lontane dalle estremità: meccanismi di origine e ruolo nell’evoluzione. Ricerca citogenetica e genomica. 122 (3-4): 219-228.
- Tomkins, J. P. 2018. I dati genomici combinatori confutano la fusione evolutiva del cromosoma 2 umano e costruiscono un modello di progettazione funzionale per le ripetizioni telomeriche interstiziali. Negli atti dell’OTTAVA Conferenza Internazionale sul Creazionismo. J. H. Whitmore, ed. Pittsburgh, PA: Creation Science Fellowship, 222-228.
- Tomkins, J. e J. Bergman. 2011. Il modello di fusione del cromosoma 2 dell’evoluzione umana-parte 2: ri-analisi dei dati genomici. Diario della Creazione. 25 (2): 111-117.
- Aldrup-Macdonald, M. E. e B. A. Sullivan. 2014. Il passato, il presente e il futuro della genomica centromerica umana. Geni (Basilea). 5 (1): 33-50.
- Tomkins, J. P. 2017. Debunking the Debunkers: una risposta alle critiche e all’offuscamento riguardo alla confutazione della fusione del cromosoma 2 umano. Risposte Rivista di ricerca. 10: 45-54.
- Voronin, D. A. ed E. V. Kiseleva. 2008. Ruolo funzionale delle proteine contenenti ripetizioni di anchirina. Biologia cellulare e Tissutale. 49 (12): 989-999.
* Dr. Tomkins è direttore di scienze della vita presso l’Istituto per la ricerca sulla creazione e ha conseguito il dottorato di ricerca in genetica presso la Clemson University.