Co to jest Colony PCR?
Colony PCR jest szybką, wysokoprzepustową metodą PCR w celu określenia obecności lub braku wstawionego DNA do plazmidu bezpośrednio z kolonii bakteryjnych.
klonowanie molekularne jest od dawna jedną z najpopularniejszych metod transformacji DNA. Jednak w celu określenia obecności lub braku wkładki DNA musimy przeprowadzić eksperymenty transformacyjne.
Colony PCR jest nowatorską metodą, w której projektując wstawione startery specyficzne dla DNA, możemy określić, czy nasze interesujące DNA jest wstawiane do plazmidu, czy nie.
jednak nie jest to tak proste, jak mówimy.
w tym artykule skupimy się szczególnie na PCR Kolonii, zasadzie PCR Kolonii, jej zaletach i ograniczeniach.
w tym celu musimy zrozumieć kilka terminów i tematów. Temat zaczniemy od podstaw. Treść artykułu jest,
kluczowe tematy:
“plazmid jest bakteryjnym kolistym DNA, które replikuje się niezależnie od chromosomu bakterii i jest wykorzystywane w manipulacji genowej i transferze genów.”
klonowanie genetyczne jest tradycyjnym molekularnym narzędziem genetycznym używanym od dawna w laboratoriach. Krótko mówiąc, w klonowaniu genów, Gen naszego zainteresowania jest wprowadzany do plazmidu za pomocą sztucznych środków. DNA to jest replikowane niezależnie od chromosomu bakterii.
plazmidy są w rzeczywistości używane do generowania wielu kopii krótkich segmentów DNA. Ponieważ bakterie replikują się szybciej niż jakiekolwiek inne organizmy, możemy wytworzyć wiele kopii interesującego nas genu poprzez wprowadzenie go do plazmidu bakteryjnego.
plazmid F, plazmid Col, plazmid degradacyjny i plazmid oporności to kilka typowych plazmidów występujących u bakterii.
ponadto plazmid może działać jako nośnik molekularny, który przenosi krótkie segmenty DNA z jednej komórki do drugiej.
omówiliśmy niesamowity dogłębny artykuł na temat plazmidu DNA. Przeczytaj tutaj: plazmid DNA – struktura, funkcja, izolacja i zastosowania.
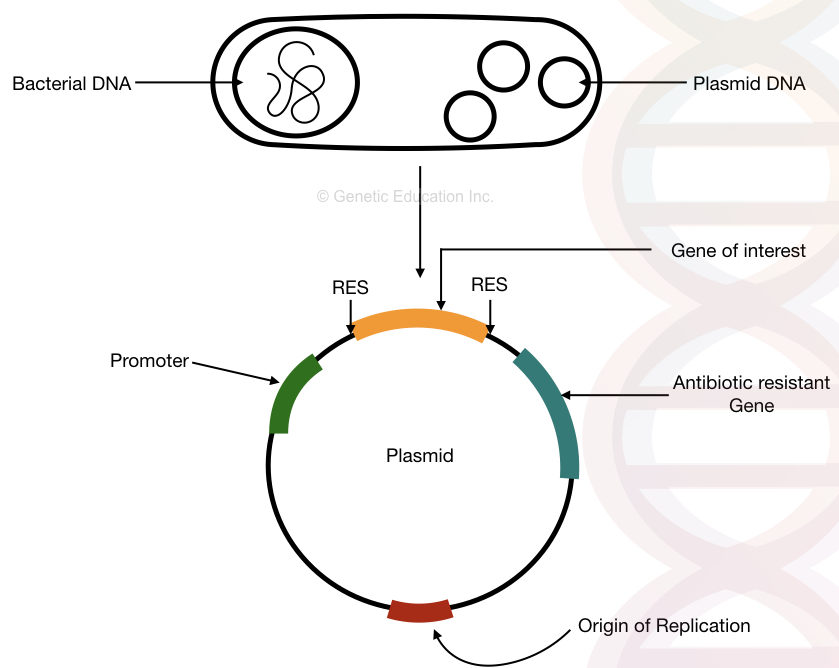
struktura bakteryjnego plazmidu DNA z pochodzeniem replikacji, genem oporności na antybiotyki, promotorem i interesującym genem.
oprócz bakterii kilka innych prokariotów zawiera również plazmid DNA. Główną funkcją plazmidu u bakterii jest ich przetrwanie w trudnych warunkach.
ponieważ plazmid przenosi Gen naszego zainteresowania, bardzo ważne jest określenie, czy nasz Gen zainteresowania jest wstawiony, czy nie do plazmidu.
w tym celu możemy użyć kilku metod, takich jak PCR i hodowla drobnoustrojów.
Platerowanie Kolonii zajmuje więcej czasu, a czułość metody również nie jest dobra. Ryzyko zanieczyszczenia jest zawsze wysokie w metodach hodowli bakteryjnej.
więc wyniki nie są dokładne.
nasz PCR również tutaj pomaga. Stosując metodę colony PCR, można określić lub zidentyfikować wkładkę DNA.
- jak skonfigurować laboratorium ekstrakcji DNA: kompleksowy przewodnik (chemikalia, instrumenty i inne narzędzia).
- delecja chromosomu 6P: powód braku bólu, głodu i snu
co to jest Colony PCR?
Colony PCR jest modyfikacją konwencjonalnego PCR, w którym kolonie bakteryjne są bezpośrednio używane jako szablon PCR.
plazmidowy DNA, który zawiera DNA naszego zainteresowania, jest wzmacniany w Warunkach cyklicznie zależnych od temperatury.
graficzna reprezentacja Kolonii PCR jest pokazana na rysunku poniżej,
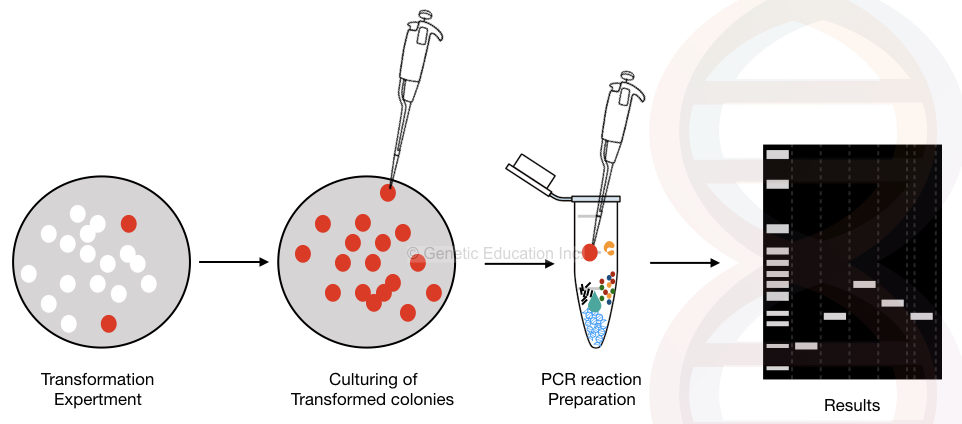
ogólny przegląd metody PCR Kolonii.
:
kolonię bakteryjną zawierającą plazmid można bezpośrednio amplifikować za pomocą dwóch zestawów starterów. Startery specyficzne dla wstawki, które wzmacniają sekwencję wstawki i startery flankujące specyficzne dla wektora, które wzmacniają plazmidowy DNA inny niż wstawiony DNA (obszary flankujące po obu stronach wstawki).
za pomocą podkładu flankującego (który wzmacnia resztę DNA) można określić rozmiar naszej wkładki DNA.
pobiera się kolonię bakteryjną i dodaje bezpośrednio do mastermixu zawierającego wszystkie odczynniki PCR. Dodatkowo, dodając jeden początkowy etap ogrzewania do PCR, plazmid DNA wychodzi z komórki bakterii i wzmacnia się w reakcji.
jest to podstawowa zasada PCR kolonii, jednak może być modyfikowana w zależności od wymagań.
Protokół PCR Kolonii:
PCR kolonii jest jedną z doskonałych modyfikacji konwencjonalnego PCR. Zamiast szablonu DNA, kolonie bakteryjne są bezpośrednio dodawane do reakcji. Poza tym, Taq DNA polimeraza, startery, PCR reakcja bufor i DD/W dodawać w PCR reakcja zbyt.
tutaj w Kolonii wybór podkładów jest bardzo ważny. Również wybór starterów zależy od celu naszego eksperymentu.
jakiego typu informacji chcemy od naszego eksperymentu?
-
-
- informacja tylko o obecności lub braku wkładki.
- informacja o rozmiarze wkładki.
- informacja o orientacji wkładki.
-
w zależności od tego różne startery PCR są przeznaczone dla kolonii PCR.
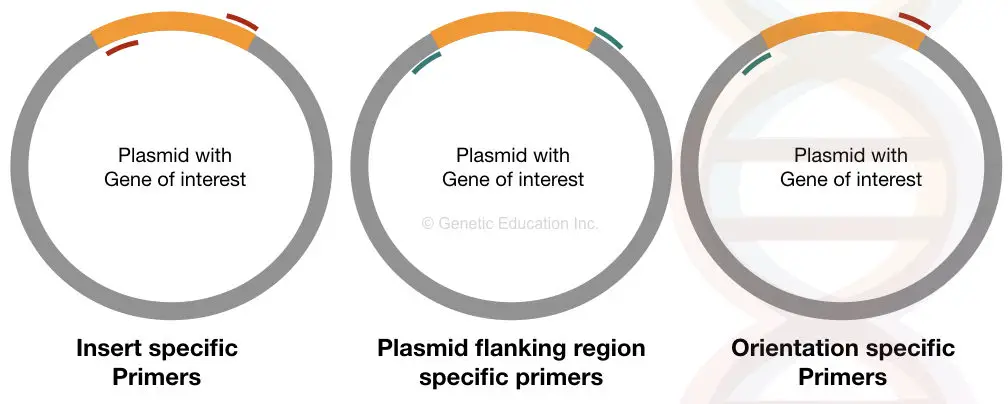
podkłady specyficzne dla wkładki wiążą się z określoną lokalizacją po obu stronach wstawionego DNA. Jeśli zostanie prawidłowo przeniesiony do plazmidu, te startery mogą się z nim wiązać, w przeciwnym razie nie może się wiązać.
Ten zestaw podkładów dostarcza informacji o obecności lub braku wkładki.
startery specyficzne dla orientacji są unikalnymi starterami, w których jeden Starter wiąże się wewnątrz Insertu, a inny Starter wiąże się z sekwencją plazmidowego DNA (sekwencją inną niż insercyjny DNA).
ten typ zestawu podkładów dostarcza informacji o orientacji wstawionego DNA, które nas interesuje. Jeśli nasze DNA nie jest właściwie ligowane do wektora, Starter właściwy dla tej strony sekwencji nie może się związać i nie uzyskamy amplifikacji.
startery specyficzne dla plazmidów są równie ważne jak startery specyficzne dla orientacji. Ten zestaw starterów jest zaprojektowany z obszaru flankującego wkładki, który wiąże się z zewnętrzną częścią DNA naszego zainteresowania.
Ten zestaw podkładu pomaga określić rozmiar wkładki. Rozszerza regiony inne niż insert DNA.
reakcja PCR dla przeprowadzenia PCR kolonii jest następująca,
| Składnik | stężenie | Ilość |
| Master mix (Special
dla kolonii) |
1X | 12µl |
| bufor reakcji PCR
z 2mm MgCl2* |
1X | 5µL |
| podkład do przodu | 10pM | 10pm |
| Reverse primer | 10pM | 10pm |
| 3µL | ||
| woda | 3µL | |
| razem | ——— | 25µL |
procedura PCR Kolonii:
Cóż, PCR kolonii nie wymaga ekstrakcji DNA.
nie wydobywamy tu DNA. Zamiast tego stosuje się kilka innych metod zwiększania czułości reakcji.
ok, dlaczego nie pobieramy DNA z plazmidu?
ponieważ przyczyna jest prosta, błona komórkowa komórki bakteryjnej jest bardzo gładka.
omówiliśmy już błonę komórkową komórki bakteryjnej . Przeczytaj to tutaj: różne rodzaje metod ekstrakcji DNA
bakteria zawiera miękką błonę komórkową, którą można łatwo lizować przez ogrzewanie lub odwirowanie z dużą prędkością.
ponadto nie potrzebujemy własnego DNA bakterii. Krążący plazmid kołowy jest obecny w cytoplazmie bakterii, dlatego nie są wymagane dodatkowe etapy oczyszczania. Przez rozerwanie błony komórkowej, nasz szablon DNA jest gotowy do amplifikacji.
ok, pozwala szybko przejść przez metodę uzyskania dobrego plazmidu DNA.
za pomocą sterylnego pickera wybierz kilka kolonii bakteryjnych i przenieś je do probówki Eppendorfa.
teraz dodaj bufor TE i dobrze wymieszaj. Możesz również użyć D / W.
podgrzać próbkę we wrzącej łaźni wodnej przez 20 minut.
odwirować próbkę z dużą prędkością przez 2 minuty.
Przenieś supernatant do innej probówki i użyj go jako wzorca DNA.
do reakcji dodaje się próbkę 20µl.
dodatkowe informacje:
dlaczego nie?
DNA jest biomolekułą życia. Plazmid DNA jest nawet mniejszy niż bakteryjne DNA jądrowe. Zawiera tylko kilka genów o długości do 1000bp do 20 000 bp.
dlatego Poprzez tylko odwirowanie go, lżejszy plazmid DNA wychodzi z komórki i osadza się w supernatancie, podczas gdy pellet zawiera białka i jądrowe DNA, więc nie używamy go.
teraz przejdźmy do sedna.
nasz plazmid jest gotowy do amplifikacji.
w innej metodzie,
użyj kolonii bakterii bezpośrednio.
ta metoda jest kombinacją Hotstart PCR i colony PCR.
kolonie bakteryjne są zbierane i dodawane do probówki reakcyjnej PCR.
rury są umieszczane w maszynie PCR. Dodano jeden dodatkowy etap ogrzewania.
ogrzewając go przez 5 do 7 minut, plazmid DNA wychodzi z komórki.
teraz startery specyficzne dla wkładki wzmacniają DNA, które wstawiliśmy. A flankujące startery wzmacniają resztę DNA.
wzmocnienie odbywa się przez 20 do 25 cykli. Warunki rowerowe dla kolonii są wymienione poniżej,
| etapy PCR | wstępne denaturacja | denaturacja | wyżarzanie | przedłużenie | końcowe przedłużenie |
| Temperatura | 95 C | 95 C | 55-65 C | 72 C | 72 C |
| czas | 3min | 10 sek | 45 sek | 50 SEK | 5 min |
| ——- | ——- | 25 cykle | —– | ——- |
przeczytaj interesujący artykuł na temat konwencjonalnego PCR: kompletny przewodnik po reakcji łańcuchowej polimerazy
Wskazówki dotyczące poprawy:
używaj tylko kilku Kolonii, ponieważ wiele kolonii zwiększa szansę na niespecyficzne wiązania.
użyj kontroli dodatniej i kontolu ujemnego.
jako kontrolę pozytywną użyto podkładu flankującego, nawet jeśli wkładka nie jest obecna, reakcja PCR daje pasmo DNA plazmidu DNA, co wskazuje, że przygotowana przez nas reakcja jest prawidłowa.
jako kontrolę negatywną, użyj nieztransformowanego plazmidu (plazmid bez dna wstawki), ten plazmid DNA jest wzmacniany tylko wtedy, gdy wstawka jest obecna.
jako wkładkę stosuje się krótkie sekwencje DNA, dłuższe sekwencje DNA zwiększają ryzyko niespecyficznych wiązań i niepowodzenia reakcji PCR.
ponadto należy używać krótszych programów PCR.
głównym zastosowaniem colony PCR jest identyfikacja prawidłowej ligacji i wstawiania insercyjnego DNA do bakterii, a także plazmidu drożdży.
po zakończeniu reakcji PCR Kolonii produkty PCR są prowadzone na 2% żelu agarozowym. Wyniki eksperymentu przedstawiono na rysunku poniżej,
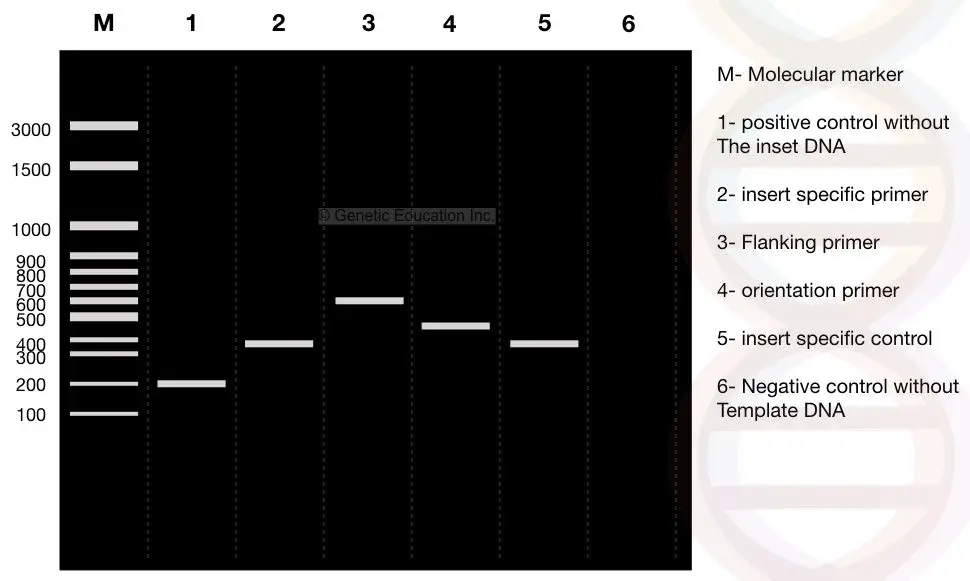
teraz uważnie obserwuj wyniki, M jest markerem molekularnego DNA 3000bp. Załóżmy, że DNA naszego zainteresowania, “insert” jest fragmentem 400bp, który jest wstawiany do plazmidu.
Zobacz pas 2: fragment naszej wkładki 400 bp.
zaprojektowaliśmy podkłady flankujące 100bp z dala od obu stron wkładki. Jeśli podkład flankujący wzmacnia DNA wraz z wkładką, produkt wynosi 600 bp, patrz linia 3 (400 bp insert DNA + 200 bp flankujący region).
teraz, patrz linia 1, jest to kontrola pozytywna bez wstawki lub normalny plazmid bez przekształconego DNA. Stąd startery flankujące wzmacniają tylko 200 bp DNA.
Patrz pas 1, 200bp fragment DNA bez wstawki (kontrola pozytywna).
obserwuj pas 4. Pas 4 To wyniki podkładów specyficznych dla orientacji. Podkład orientacyjny jest połączeniem podkładu specyficznego dla wkładki i podkładu flankującego specyficznego dla regionu.
jeden podkład z insert DNA i jeden podkład z primer specyficzny dla regionu flankowania są wybierane do wzmocnienia podkładu specyficznego dla orientacji.
w związku z tym w pasie 4 obserwuje się amplifikację fragmentu DNA o długości 100 bp z obszaru flankującego i 400bp z dna wstawionego, a w pasie 4 obserwuje się fragment DNA o długości 500 bp.
pas 5 to kontrola swoista insert, która daje fragment DNA o długości 400 bp.
Dzięki zastosowaniu kontroli negatywnej można zidentyfikować wszelkie zanieczyszczenia. Rurka reakcyjna zawiera wszystkie składniki z wyjątkiem szablonu. Więc idealnie byłoby, gdyby w tym pasie nie było pasm DNA.
jeśli obserwuje się pasmo DNA, próbka jest zanieczyszczona.
zalety Colony PCR:
- technika jest szybka i opłacalna.
- co więcej, dokładność i specyficzność techniki są wyższe.
- konfiguracja jest prosta, podobnie jak konwencjonalne PCR, ekstrakcja DNA i oczyszczanie plazmidów, takie jak żmudne kroki nie są wymagane.
- nie ma potrzeby trawienia restrykcyjnego w celu identyfikacji wstawionego DNA.
- cały eksperyment można zakończyć w ciągu 90 minut.
:
- metoda jest opłacalna, szybka i niezawodna, jednak nie można wykryć żadnej mutacji we wkładce.
- co więcej, informacji o sekwencji nie można uzyskać przez PCR Kolonii. musimy przeprowadzić sekwencjonowanie w celu potwierdzenia transformacji DNA.
- szansa na wyniki fałszywie dodatnie jest wysoka.
Czytaj więcej;
- co to jest multipleks PCR?
po zakończeniu eksperymentu próbka jest wysyłana do sekwencjonowania, gdzie można określić sekwencję DNA naszego zainteresowania.
możemy nawet wykonać multipleks PCR, łącząc zarówno podkłady specyficzne dla wstawek, jak i podkłady specyficzne dla plazmidów.
wniosek:
chociaż metoda PCR kolonii jest najlepszym wyborem do identyfikacji transferu genów, jedyna technika PCR kolonii nie jest wystarczająca do interpretacji wyników. Możliwe, że niektóre mutacje obecne we wkładce, które nie mogą być wykryte przez PCR.
do potwierdzenia wyników wymagane jest sekwencjonowanie DNA. Po określeniu kolejności sekwencji możemy powiedzieć, czy nasz gen jest wstawiany prawidłowo, czy nie.