o Instituto de pesquisa da criação
 um dos argumentos mais populares usados para humanos supostamente evoluindo de macacos é conhecido como a fusão cromossômica. O ímpeto para este conceito é o problema evolutivo de que os macacos têm um par extra de cromossomas—os humanos têm 46 enquanto os macacos têm 48. Se os humanos evoluíram de uma criatura parecida com um macaco há apenas três a seis milhões de anos, uma mera falha no grande esquema da história evolutiva, porque é que os humanos e os macacos têm esta discrepância?
um dos argumentos mais populares usados para humanos supostamente evoluindo de macacos é conhecido como a fusão cromossômica. O ímpeto para este conceito é o problema evolutivo de que os macacos têm um par extra de cromossomas—os humanos têm 46 enquanto os macacos têm 48. Se os humanos evoluíram de uma criatura parecida com um macaco há apenas três a seis milhões de anos, uma mera falha no grande esquema da história evolutiva, porque é que os humanos e os macacos têm esta discrepância?
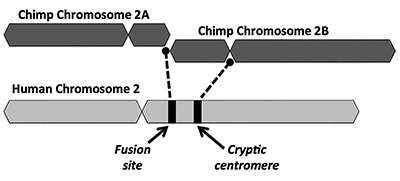
a solução evolutiva propõe que uma fusão final-a-final de dois pequenos cromossomas semelhantes a macacos (chamados 2A e 2B) produziu o cromossoma humano 2 (Figura 1). O conceito de fusão surgiu pela primeira vez em 1982, quando os cientistas examinaram as semelhanças dos cromossomas humanos e símios sob um microscópio. Enquanto a técnica era um pouco grosseira, era o suficiente para começar a idéia.1
O Chamado Local De Fusão
a primeira assinatura de DNA real de um possível Evento de fusão foi descoberta em 1991 no número de cromossomos humanos 2.2 pesquisadores encontraram um pequeno aglomerado de sequências finais tipo telômero que se assemelhavam vagamente a uma possível fusão. Telómeros são uma sequência de seis bases das letras de ADN TTAGG repetidas vezes nas extremidades dos cromossomas.
no entanto, a assinatura de fusão foi um enigma baseado nas fusões reais que ocasionalmente ocorrem na natureza. Todas as perfusões documentadas em Animais vivos envolvem um tipo específico de sequência chamado DNA satélite (satDNA) localizado em cromossomos e encontrado em quebras e perfusões.3-5 a assinatura de fusão no cromossoma humano 2 estava faltando este telltale satDNA.6
outro problema é o pequeno tamanho do local de fusão, que tem apenas 798 letras de ADN. As sequências de Telomere nas extremidades dos cromossomas têm 5 000 a 15 000 bases de comprimento. Se dois cromossomas se fundiram, deve ver uma assinatura telomere fundida de 10 000 a 30 000 bases de longa duração—não 798.
não só o pequeno tamanho é um problema para a história de fusão, a assinatura realmente não representa uma fusão clara de telómeros. A figura 2 mostra as letras de ADN do local de fusão de base 798 com as sequências de telômero intactas de seis bases (letra de ADN) enfatizadas a negrito. Quando a sequência de fusão é comparada com a de uma assinatura de fusão Pristina do mesmo tamanho, ela é apenas 70% idêntica em geral.

pesquisadores seculares apontaram esta discrepância e rotularam o local de fusão como significativamente “degenerado”.”7 dado o modelo teórico padrão da evolução humana, deve ser cerca de 98 a 99% idêntico, não 70%. Os pesquisadores que descrevem esta descoberta comentou, “Cabeça-a-cabeça matrizes de repetições a fusão site degeneraram significativamente (14%) de quase perfeita, matrizes de (TTAGGG)n encontrado em telômeros” e pediu a pertinente pergunta: “Se a fusão ocorreu dentro do telomeric repita matrizes menos de ~6 milhões de anos atrás, por que são as matrizes a fusão site tão degenerada?”7 Note-se que a degeneração de 14% citada pelos autores se refere à corrupção de apenas as sequências de seis bases em si, e não a totalidade das 798 bases.O local de fusão dentro de um Gene?
a descoberta anti-evolucionária mais notável sobre o local de fusão acabou por ser a sua localização e o que ele realmente faz. Esta descoberta ocorreu enquanto eu estava lendo o artigo de pesquisa que relatou uma análise detalhada de 614.000 bases de sequência de DNA em torno do suposto local de fusão. Notei em uma das figuras que o local de fusão estava localizado dentro de um gene, e notavelmente essa estranheza nem sequer foi reconhecida no texto do jornal.8
uma descoberta como esta é altamente notável. Talvez esta informação fosse o prego no caixão evolucionário, por assim dizer, e é por isso que os investigadores se recusaram a discuti-la. Esta grande anomalia inspirou-me a dar ao local da fusão um exame muito mais próximo. Este artigo foi publicado em 2002, e eu tomei nota dele em 2013. Uma enorme quantidade de dados sobre a estrutura e função do genoma humano tinha sido publicada entretanto, e havia provavelmente muito mais sobre a história que precisava ser descoberta.
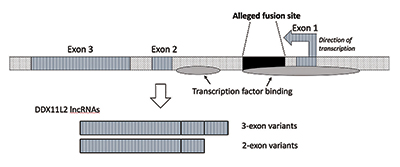
quando realizei mais pesquisas, verifiquei que o local de fusão estava posicionado dentro de um gene Rna helicase agora chamado DDX11L2. A maioria dos genes em plantas e animais tem seus segmentos de codificação em pedaços chamados exons para que eles possam ser alternativamente spliced. Com base na adição ou exclusão de exons, os genes podem produzir uma variedade de produtos. As regiões intermediárias entre exons são chamadas de introns, que muitas vezes contêm uma variedade de sinais e interruptores que controlam a função genética. O suposto local de fusão é posicionado dentro do primeiro intrão do gene DDX11L2 (Figura 3).9
a molécula de ADN é de cadeia dupla, com uma cadeia mais e uma cadeia menos. Ele foi projetado desta forma para maximizar a densidade da Informação, ao mesmo tempo em que aumenta a eficiência e função. Como resultado, há genes correndo em diferentes direções nas cadeias opostas. Ao que parece, o gene DDX11L2 é codificado na cadeia de menos. Como os genes em humanos são como facas do exército suíço e podem produzir uma variedade de RNAs, no caso do gene DDX11L2 produz pequenas variantes consistindo de dois exons e variantes longas com três (Figura 3).9
o local de fusão é um promotor de genes
o que pode este gene DDX11L2 estar a fazer? A minha pesquisa mostrou que é expresso em pelo menos 255 tipos diferentes de células ou tecidos.9 também é co-expresso (ligado ao mesmo tempo) com uma variedade de outros genes e está ligado a processos associados com a sinalização celular na matriz extracelular e produção de células sanguíneas. A localização da chamada sequência de fusão dentro de um gene funcional associado à genética de uma variedade de processos celulares refuta fortemente a ideia de que é o subproduto acidental de uma fusão telomérica cabeça-a-cabeça. Os Genes não são formados por catastróficas fusões cromossómicas!
ainda mais surpreendente é que o local de fusão é em si funcional e serve um propósito importante de engenharia. O site na verdade atua como um interruptor para controlar a atividade do gene. A este respeito, uma riqueza de dados bioquímicos mostrou que 12 proteínas diferentes chamadas fatores de transcrição regulam este segmento do gene. Uma delas é a RNA polimerase II, A principal enzima que copia moléculas de RNA do DNA em um processo chamado transcrição. Além disso, apoiar esta descoberta é o fato de que o processo real de transcrição inicia-se dentro da região do chamado local de fusão.Tecnicamente, a actividade no alegado local de fusão é uma região promotora. Promotores são os principais interruptores no início dos genes que os ligam e são também onde a RNA polimerase começa a criar um RNA. Muitos genes têm promotores alternativos como o gene DDX11L2.
existem na verdade duas áreas de ligação do factor de transcrição no gene DDX11L2. O primeiro está no promotor diretamente na frente do primeiro exon, e o segundo está no primeiro intron correspondente à sequência do local de fusão. Não só o gene DDX11L2 em si é complexo e controlado, com a alegada sequência de fusão desempenhando um papel chave, mas mesmo as transcrições de RNA produzidas são muito intrincadas. Os próprios RNAs contêm uma grande variedade de locais de ligação e controle para uma classe de pequenas moléculas reguladoras chamadas microRNAs.9
sequências funcionais internas de telômero estão por todo o genoma
a presença de sequências telômeras localizadas internamente é encontrada em todo o genoma humano. Estas repetições de telômero aparentemente fora de lugar foram apelidadas de telômeres intersticiais. A presença destas sequências apresenta outro desafio para a ideia do local de fusão. É um facto que muito poucas repetições de telômero no local de fusão ocorrem em conjunto. Como observado na Figura 2, a sequência do local de fusão de base 798 contém apenas algumas instâncias onde duas repetições estão realmente em tandem e nenhuma que tem três repetições ou mais. No entanto, existem muitos outros locais de telômero intersticiais por todo o genoma humano onde as repetições ocorrem em perfeito conjunto três a dez vezes ou mais.10-11
mesmo além do seu papel nas extremidades dos cromossomas, parece que as repetições teloméricas intersticiais podem servir uma função importante no genoma relacionado com a expressão genética. Num recente projecto de investigação, identifiquei repetições de telomere por todo o genoma humano e depois intersectei as suas localizações genómicas com uma diversidade de conjuntos de dados contendo informação bioquímica funcional para a actividade genética.12 literalmente milhares de repetições teloméricas internas ao longo do genoma foram diretamente associadas com as marcas da expressão genética. O mesmo tipo de ligação do factor de transcrição e actividade genética que ocorreu no suposto local de fusão também estava a ocorrer em todo o genoma em numerosas outras repetições teloméricas intersticiais. É evidente que estas características do ADN não são acidentes de evolução, mas sim um código funcional concebido de forma intencional e inteligente.
Bogus Cryptic Centromere Inside a Gene
Another key problem with the fusion model is the lack of viable evidence for a signature of an extra centromere region. Centrômeros são seções de cromossomos, muitas vezes em locais centrais, que desempenham papéis-chave durante a divisão celular. Como descrito na Figura 1, o recém-formado cromossoma quimérico teria dois locais de centromere imediatamente após a alegada fusão cabeça-a-cabeça dos dois cromossomas. Nesse caso, um dos centrômeros seria funcional, enquanto o outro seria desativado. A presença de dois centrómeros activos é uma má notícia para os cromossomas e conduziria à disfunção e destruição celular.
curiosamente, a evidência de um centrômero críptico (deficiente) no cromossomo humano 2 é ainda mais fraca do que a de um local de fusão rico em telômeros. Evolucionistas explicam a falta de um claramente distinguível centrômero secundário não funcional, argumentando que um segundo centrômero teria sido rapidamente selecionado contra. Depois disso, o centrômero desabilitado teria se deteriorado ao longo do tempo, já que não havia mais restrições funcionais colocadas nele por fazer algo útil no genoma.
no entanto, a evidência de um segundo centromere remanescente em qualquer estágio de degeneração de sequência é problemática para o paradigma evolucionário. As sequências funcionais de centrômero são compostas por um tipo repetitivo de DNA chamado sequências alfóides, com cada repetição alfóide sendo cerca de 171 bases de comprimento. Alguns tipos de repetições alfóides são encontrados em todo o genoma, enquanto outros são específicos de centrômeros. A estrutura das sequências encontradas no local críptico do centromere no cromossoma humano 2 não coincide com as associadas com os centrômeros humanos funcionais.13 pior ainda para o modelo evolucionário é que eles não têm homólogos altamente similares no genoma do chimpanzé—eles são humanos-específicos.13
o alegado centrômero Fóssil também é excepcionalmente pequeno em comparação com um real. O tamanho de um centrômero humano normal varia em comprimento entre 250.000 e 5.000.000 bases.14 o suposto centrômero críptico tem apenas 41.608 bases de comprimento, mas também é importante notar que há três regiões diferentes dele que nem sequer são repetições alfóides.15 dois destes são chamados retroelements, com um sendo um LPA3 / linha repetir 5,957 bases de comprimento e o outro um elemento SVA-E com 2,571 bases. Quando subtraímos as inserções dessas sequências não-alfóides, ela dá um comprimento de apenas 33.080 bases, que é uma fração do comprimento de um centrômero real.
o problema evolutivo mais grave com a ideia de um centrômero fóssil, no entanto, é que, como o suposto local de fusão, ele está posicionado dentro de um gene. O suposto centrômero críptico está localizado dentro do gene ANKRD30BL, e sua sequência abrange tanto as regiões intron e exon do gene.12,15
na verdade, a parte da suposta sequência de centrômero fóssil que pousa dentro de um exon realmente codifica aminoácidos na proteína do gene resultante. O gene produz uma proteína que se acredita estar envolvida na interação da rede estrutural de proteínas dentro da célula chamada citosqueleto em conexão com as proteínas receptoras incorporadas na membrana celular.16 o facto de o chamado centrômero fóssil ou críptico ser uma região funcional dentro de um gene importante que codifica proteínas refuta completamente a ideia de que é um centrômero extinto.
conclusão: Não há fusão
devido às assinaturas confusas e pequenos tamanhos dos supostos locais de fusão e centrômero fóssil, é altamente questionável que a sua sequência foi evolutivamente derivada de uma antiga fusão cromossômica. Além disso, representam uma sequência funcional dentro dos genes. O suposto local de fusão é um importante interruptor genético chamado promotor dentro do gene RNA ddx11l2 de longa duração não codificante,e o chamado centrômero Fóssil contém tanto a sequência de codificação e não codificação dentro de um grande gene de codificação de proteínas anquirinas.
este é um duplo whammy inegável contra toda a idéia de fusão mítica, destruindo completamente sua validade. A esmagadora conclusão científica é que a fusão nunca aconteceu.
- Yunis, J. J. e O. Prakash. 1982. The origin of man: a chromosomal pictorial legacy. Ciência. 215 (4539): 1525-1530.
- Ijdo, J. W. et al. 1991. Origin of human chromosome 2: An ancestral telomere-telomere fusion. Proceedings of the National Academy of Sciences. 88 (20): 9051-9055.
- Chaves, R. et al. 2003. Molecular citogenetic analysis and centromeric satellite organization of a novel 8; 11 translocation in sheep: a possible intermediate in biarmed chromosome evolution. Genoma Mamífero. 14 (10): 706-710.
- Tsipouri, V. et al. 2008. Análises comparativas de sequências revelam locais de fusão cromossómica ancestral no genoma Muntjac Indiano. Biologia Do Genoma. 9 (10): R155.
- Adega, F., H. Guedes-Pinto, and R. Chaves. 2009. DNA de satélite na evolução do cariótipo de animais domésticos-considerações clínicas. Investigação citogenética e genómica. 126 (1-2): 12-20.
- Tomkins, J. P. and J. Bergman. 2011. Telómeros: implicações para o envelhecimento e evidência para o design inteligente. Diário da criação. 25 (1): 86-97.
- Fan, Y. et al. 2002. Estrutura genómica e evolução do local Ancestral de fusão cromossómica em 2q13–2q14.1 e regiões Paralógicas em outros cromossomas humanos. Pesquisa Do Genoma. 12 (11): 1651-1662.
- Fan, Y. et al. 2002. Conteúdo genético e função do local de fusão cromossómica Ancestral no cromossoma humano 2q13-2q14. 1 e nas regiões Paralógicas. Pesquisa Do Genoma. 12 (11): 1663-1672.
- Tomkins, J. P. 2013. Alegado cromossoma humano 2 “Sítio de fusão” codifica um domínio de ligação de ADN activo dentro de uma fusão complexa e altamente expressa de negação de genes. Answers Research Journal. 6: 367-375.
- Azzalin, C. M., S. G. Nergadze, and E. Giulotto. 2001. Human intracromosomal telomeric-like repeats: sequence organization and mechanisms of origin. Cromossoma. 110: 75-82.
- Ruiz-Herrera, A. et al. 2008. Telomérico repete longe dos fins: mecanismos de origem e papel na evolução. Investigação citogenética e genómica. 122 (3-4): 219-228.
- Tomkins, J. P. 2018. Os dados genômicos combinatórios refutam a fusão evolutiva do cromossomo 2 humano e constroem um modelo de design funcional para repetições telômicas intersticiais. In Proceedings of the Eighth International Conference on Creationism. J. H. Whitmore, ed. Pittsburgh, PA: Creation Science Fellowship, 222-228.
- Tomkins, J. and J. Bergman. 2011. The chromosome 2 fusion model of human evolution-part 2: re-analysis of the genomic data. Diário da criação. 25 (2): 111-117.Aldrup-Macdonald, M. E. and B. A. Sullivan. 2014. O passado, o presente e o futuro da Genómica centromere humana. Genes (Basel). 5 (1): 33-50.
- Tomkins, J. P. 2017. Debunking the Debunkers: a Response to Criticism and Ofuscation Regarding Refutation of The Human Chromosome 2 Fusion. Answers Research Journal. 10: 45-54.
- Voronin, D. A. and E. V. Kiseleva. 2008. Papel funcional das proteínas que contêm Anquirina repete-se. Biologia Celular e tecidular. 49 (12): 989-999.
* o Dr. Tomkins é Diretor de Ciências da vida do Instituto de pesquisa da criação e obteve seu Ph. D. em genética da Universidade Clemson.