Transposons: Arten von Transposons / Genetik
WERBUNG:
Es gibt zwei Arten von Transposons: zusammengesetztes Transposon und komplexes Transposon.
1. Die zusammengesetzten Transposons:
Die zusammengesetzten Transposons sind solche, die aus einer zentralen Region bestehen, die antibiotikaresistente Gene trägt, die an beiden Enden von identischen Kopien eines IS-Elements flankiert werden. 8.32A). Es ist eine Klasse von größeren Transposons. Drei häufig untersuchte zusammengesetzte Transposons sind Tn5, Tn9 und Tn10.
Tn5-Element zeigt Kanamycin-Resistenz (kanr) und besteht aus 5.400 Basenpaarsegmenten mit 1.450 bp invertierten Wiederholungen an beiden Enden des Segments. Es kann vom Phagen λ zum Chromosom von E transponiert werden .coli und von einem Locus des K-Coli-Chromosoms zu einem anderen Locus. Wenn es in Gene eingefügt wird, verursacht es Mutationen.
ANZEIGEN:
Das Tn9-Transposon besteht aus einem vom R-Faktor abgeleiteten Gen für die Chloramphencol-Resistenz (camr). Das Enzym, das Arzneimittelresistenz verleiht, besteht aus 2.638 Sequenzen in der Mitte von Tn, flankiert von einem 768 bp langen IS1-Element auf beiden Seiten. Tn9 IS1 ist in direkter Reihenfolge mit kleinen invertierten Wiederholungen an den Enden vorhanden.
Das camr-Segment wird von einem R-Faktor zu F-Episom sowie Phage λ durch den Phagen P1 transloziert. T9 unterscheidet sich von den anderen Transposons durch seine Instabilität und einen hochfrequenten Verlust seiner Antibiotikaresistenz.
Tn10 besteht aus Tetracyclin-Resistenzgenen (Tetr-Genen). Es ist 9.300 bp lang und besteht aus invertierten Wiederholungen auf beiden Seiten von 1.400 Basenpaaren. Die IS-Elemente ist IS10. Tn10 kann von R222 (einem arzneimittelresistenten Plasmid) zu Phage P22 transloziert werden.
ANZEIGEN:
Struktur von Komposittransposons:
Bei den Komposittransposons können die IS-Elemente in einer invertierten oder direkten Wiederholungskonfiguration vorliegen (Abb. 8.31B). Die beiden Enden der Elemente sind selbst invertierte Wiederholungen. Die relative Orientierung (direkt oder invertiert) der flankierenden IS-Elemente eines zusammengesetzten Transposons ändert seine terminalen Sequenzen nicht. Somit hat ein zusammengesetztes Transposon mit Armen direkter Wiederholungen die Struktur: Arm L- Zentralregion – Arm R.
Die Struktur wird: Arm L–Zentralregion – Arm R, wenn die Arme invertierte Wiederholungen sind. Die Pfeile zeigen die Ausrichtung der Arme entsprechend der Ausrichtung der genetischen Karte des Transposons von links (L) nach rechts (R) (Abb. 8.31B, i-ii). Die Merkmale von Komposittransposons sind in Tabelle 8.4 angegeben.
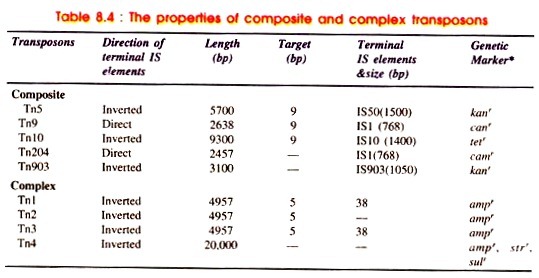
Darüber hinaus gibt es einige Fälle, in denen Module eines zusammengesetzten Transposons identisch sind, beispielsweise Tn9 (hat direkte Wiederholung von IS1) oder Tn903 (invertierte Wiederholungen von IS 903 vorhanden), während in bestimmten Fällen die Module eng verwandt sind. Daher können die Module in Tn10 oder Tn5 unterschieden werden. Wenn die Module jedoch identisch sind, können beide die Bewegung des Transposons unterstützen, wie in Tn9 von Tn903 gefunden.
Wenn sich die Module unterscheiden, können sie sich auch in der Funktionsfähigkeit unterscheiden. Daher hängt die Transposition vollständig von einem der Module ab, beispielsweise Tn10 oder Tn5. Somit transponiert ein IS-Modul, wenn es funktionsfähig ist, entweder sich selbst oder das gesamte Transposon.
Die Fähigkeit eines einzelnen Moduls, das gesamte zusammengesetzte Transposon zu transponieren, erklärt den Mangel an selektivem Druck, damit beide Module aktiv bleiben. Der Isomerencode für die Transposaseaktivität, der sowohl für die Erstellung einer Zielstelle als auch für die Erkennung der Enden des Transposons verantwortlich ist. Nur die Enden werden benötigt, damit ein Transposon als Substrat für die Transposition dient.
2. Die komplexen Transposons (TnA-Transposonfamilie):
Die TNA-Familie der Transposons umfasst Tn1, Tn2 und Tn3. TnA besteht aus ziemlich großen Elementen (etwa 500 bp). Diese enthalten unabhängige Einheiten, die Gene sowohl für die Transposition als auch für die Arzneimittelresistenz tragen. Dies sind keine zusammengesetzten Relays auf IS-Transpositionsmodulen. Die TnA-Familie umfasst mehrere verwandte Transposons, von denen Tn3 und Tn10 die am besten untersuchten sind.
Die TnA-Familie ist auch unter dem Namen Tn3-Familie bekannt, da das Tn3-Transposon erstmals 1974 von Hedges und Jacob in bakteriellen Plasmiden entdeckt wurde. Tn3 ist ein Beispiel für ein komplexes Transposon. Es hat eine modulare Struktur wie in Tn10 und basiert nicht auf IS-Elementen. Es hat auch keine evolutionären Verbindungen zu IS-Elementen. Die Grundstruktur von Tn3 ist für die TnA-Familie beschrieben.
ANZEIGEN:
Eines der einzigartigen Merkmale der TnA-Familie ist, dass sie mehrere Insertionen desselben Transposons in ein Plasmid begrenzt. Der überraschendste Befund ist, dass die Transpositionsfrequenz zu anderen Plasmiden in derselben Zelle nicht beeinflusst wird. Heffron (1979) haben die Analyse der DNA-Sequenz von Transposon Tn3 gegeben.
Struktur der Transposons der TnA-Familie:
Transposons der TnA-Familie bestehen aus invertierten terminalen Wiederholungen (von 38 bp lang, aber keine werden von IS-ähnlichen Elementen flankiert), einer internen res-Stelle und drei bekannten Genen, z. B. tnpA, tnpR und ampr.
Das Gen tnpA kodiert für Transposase und tnpR kodiert für Resolvase. Das ampr-Gen (5 bp lang) wird an der Zielstelle als direkte Wiederholung erzeugt und kodiert für β-Lactamase, die eine Resistenz gegen Ampicillin verleiht. Die res-Site besteht aus drei Untereinheiten: I, II und III (Abb. 8.33)