Was ist Kolonie-PCR?
Kolonie-PCR ist eine schnelle, Hochdurchsatz-PCR-Methode zur Bestimmung der Anwesenheit oder Abwesenheit der in Plasmid insertierten DNA direkt aus den Bakterienkolonien.
Das molekulare Klonen ist seit langem eine der beliebtesten Methoden zur DNA-Transformation. Zur Bestimmung der Anwesenheit oder Abwesenheit des DNA-Inserts müssen wir jedoch Transformationsexperimente durchführen.
Die Kolonie-PCR ist eine neuartige Methode, bei der wir durch das Design der insertierten DNA-spezifischen Primer feststellen können, ob unsere interessierende DNA in das Plasmid inseriert ist oder nicht.
Es ist jedoch nicht so einfach, wie wir diskutieren.
In diesem Artikel konzentrieren wir uns insbesondere auf die Kolonie-PCR, das Prinzip der Kolonie-PCR, ihre Vorteile und Grenzen.
Dazu müssen wir mehrere Begriffe und Themen verstehen. Wir werden unser Thema mit den Grundlagen beginnen. Der Inhalt des Artikels ist,
Themenschwerpunkte:
” Ein Plasmid ist die bakterielle zirkuläre DNA, die sich unabhängig vom bakteriellen Chromosom repliziert und bei der Genmanipulation und dem Gentransfer verwendet wird.”
Genetisches Klonen ist ein traditionelles molekulargenetisches Werkzeug, das seit langem in den Labors verwendet wird. Kurz gesagt, in der Genklonierung wird das Gen von unserem Interesse durch künstliche Mittel in das Plasmid eingefügt. Diese DNA wird unabhängig vom bakteriellen Chromosom repliziert.
Plasmide werden tatsächlich verwendet, um viele Kopien von kurzen DNA-Segmenten zu erzeugen. Da sich Bakterien schneller als alle anderen Organismen vermehren, können wir viele Kopien des Gens unseres Interesses erzeugen, indem wir es in das bakterielle Plasmid einfügen.
F-Plasmid, Col-Plasmid, Degradativ-Plasmid und Resistenz-Plasmid sind einige häufige Arten von Plasmiden, die in Bakterien vorkommen.
Ferner kann das Plasmid als molekularer Träger wirken, der kurze DNA-Abschnitte von einer Zelle in eine andere Zelle überträgt.
Wir haben einen erstaunlichen ausführlichen Artikel über Plasmid-DNA behandelt. Lesen Sie es hier: Plasmid-DNA- Struktur, Funktion, Isolierung und Anwendungen.
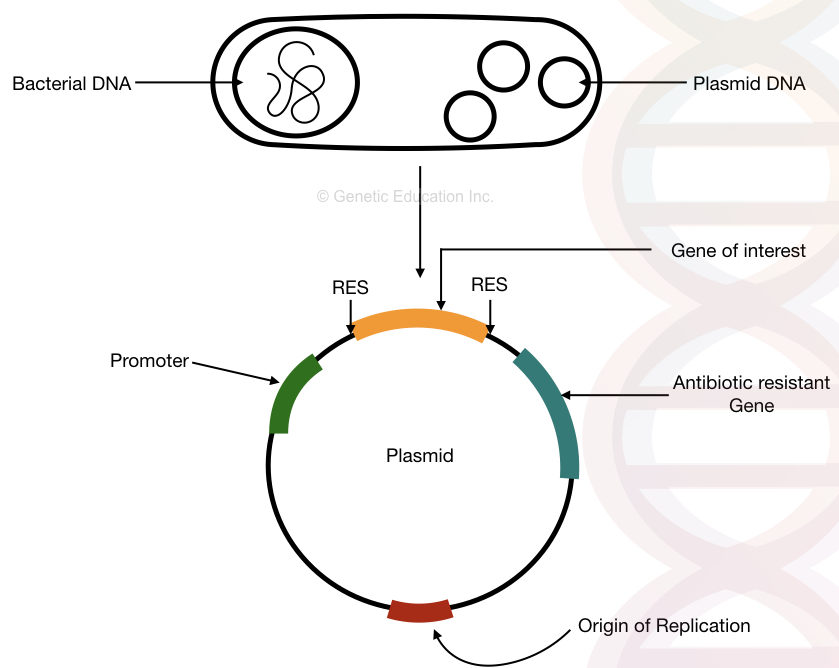
Die Struktur der bakteriellen Plasmid-DNA mit, der Ursprung der Replikation, Antibiotikaresistenzgen, Promotor und Gen von Interesse.
Neben Bakterien enthalten auch mehrere andere Prokaryoten Plasmid-DNA. Die Hauptfunktion des Plasmids in Bakterien ist für ihr Überleben in den rauen Bedingungen.
Da das Plasmid Gene von unserem Interesse überträgt, ist es sehr wichtig zu bestimmen, ob unser interessierendes Gen in das Plasmid eingefügt ist oder nicht.
Dafür können wir verschiedene Methoden wie PCR und mikrobielle Kultivierung verwenden.
Das Flotten der Kolonien dauert länger und die Empfindlichkeit der Methode ist ebenfalls nicht gut. Die Wahrscheinlichkeit einer Kontamination ist bei Bakterienkulturmethoden immer hoch.
Die Ergebnisse sind also nicht genau.
Auch hier hilft unsere PCR. Mit der Kolonie-PCR-Methode kann ein DNA-Insert bestimmt oder identifiziert werden.
- So richten Sie ein DNA-Extraktionslabor ein: Ein umfassender Leitfaden (Chemikalien, Instrumente und andere Hilfsmittel).
- Chromosom-6p-Deletion: Ein Grund für keine Schmerzen, keinen Hunger und keinen Schlaf
Was ist Kolonie-PCR?
Die Kolonie-PCR ist die Modifikation der konventionellen PCR, bei der die Bakterienkolonien direkt als PCR-Template verwendet werden.
Die Plasmid-DNA, die die DNA unseres Interesses enthält, wird unter zyklisch-temperaturabhängigen Bedingungen amplifiziert.
Die grafische Darstellung der Kolonie-PCR ist in der folgenden Abbildung dargestellt,
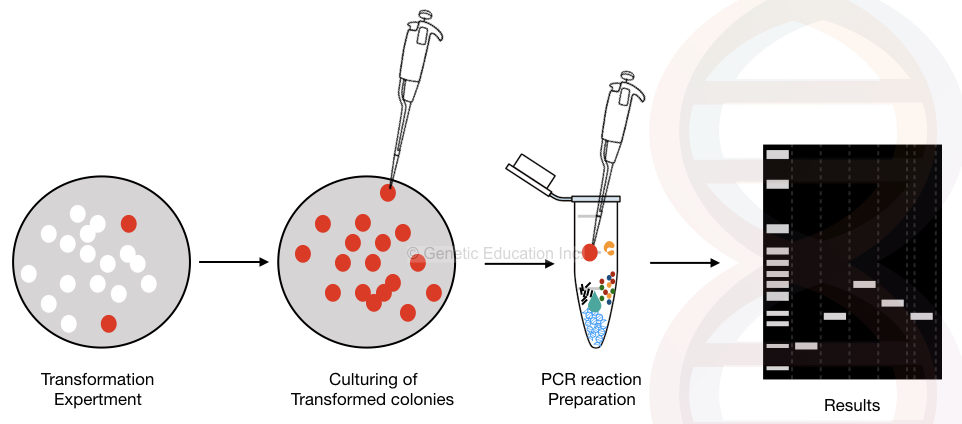
Der allgemeine Überblick über die Kolonie-PCR-Methode.
Prinzip der Kolonie-PCR:
Die Bakterienkolonie, die das Plasmid enthält, kann direkt mit zwei Primersets amplifiziert werden. Die insert-spezifischen Primer, welche die Insertionssequenz amplifizieren und vektor-spezifische flankierende Primer, welche die Plasmid-DNA anders als die insertierte DNA amplifizieren (flankierende Bereiche auf beiden Seiten des Inserts).
Durch die Verwendung der Insert-flankierenden Primer (die den Rest der DNA amplifizieren) kann die Größe unseres DNA-Inserts bestimmt werden.
Eine Bakterienkolonie wird gepflückt und direkt in den Mastermix gegeben, der alle PCR-Reagenzien enthält. Zusätzlich kommt die Plasmid-DNA durch Zugabe eines ersten Erwärmungsschritts zur PCR aus der Bakterienzelle und wird in der Reaktion amplifiziert.
Dies ist das Grundprinzip der Kolonie-PCR, kann jedoch je nach Anforderung modifiziert werden.
Das Protokoll der Kolonie-PCR:
Die Kolonie-PCR ist eine der hervorragenden Modifikationen der konventionellen PCR. Anstelle der Template-DNA werden die Bakterienkolonien direkt der Reaktion zugesetzt. Daneben werden bei der PCR-Reaktion auch Taq-DNA-Polymerase, Primer, PCR-Reaktionspuffer und DD/W zugegeben.
Hier in der Kolonie-PCR ist die Auswahl der Primer sehr wichtig. Auch die Auswahl der Primer hängt vom Ziel unseres Experiments ab.
Welche Art von Informationen wollen wir von unserem Kolonie-PCR-Experiment?
-
-
- Informationen über das Vorhandensein oder Fehlen des Einsatzes nur.
- Informationen über die Größe des Einsatzes.
- Informationen zur Ausrichtung des Einsatzes.
-
Abhängig davon werden verschiedene PCR-Primer für die Kolonie-PCR ausgelegt.
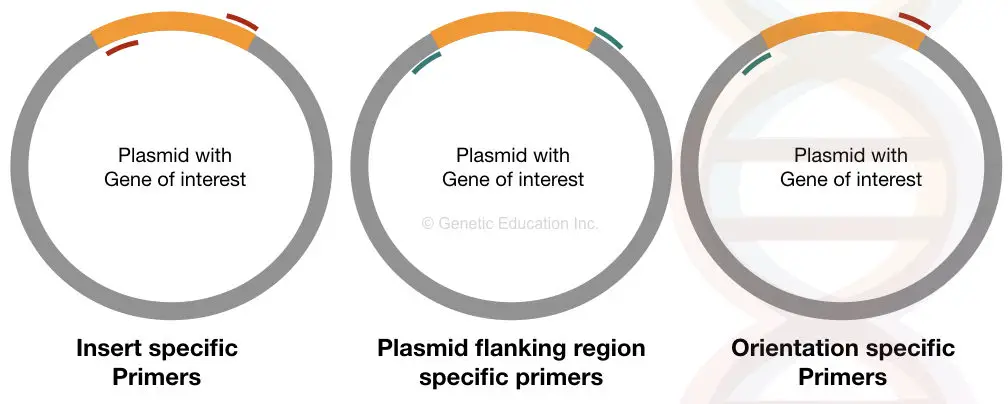
Die insert-spezifischen Primer binden an die spezifische Stelle auf beiden Seiten der eingefügten DNA unseres Interesses. Wenn es richtig in das Plasmid übertragen wird, können diese Primer daran binden, sonst kann es nicht binden können.
Dieses Primerset gibt Auskunft über das Vorhandensein oder Nichtvorhandensein des Inserts.
Orientierungsspezifische Primer sind einzigartige Primer, bei denen ein Primer innerhalb des Inserts und ein anderer Primer an die Plasmid-DNA-Sequenz bindet (andere Sequenz als die Insert-DNA).
Diese Arten von Primer-Sets liefern Informationen über die Orientierung der DNA unseres Interesses. Wenn unsere Insert-DNA nicht richtig in den Vektor ligiert ist, kann der für diese Seite der Sequenz spezifische Primer nicht binden, und wir erhalten keine Amplifikation.
Plasmidspezifische Primer sind ebenso wichtig wie die orientierungsspezifischen Primer. Dieser Satz von Primern wird aus dem flankierenden Bereich des Inserts entwickelt, der an die Außenseite der DNA unseres Interesses bindet.
Dieser Satz von Primer hilft, die Größe des Einsatzes zu bestimmen. Es erweitert andere Regionen als die Insert-DNA.
Die PCR-Reaktion zur Durchführung der Kolonie-PCR ist wie folgt,
| Komponente | Konzentration | Menge |
| Master Mix (Spezial
für die Kolonie-PCR) |
1X | 12 µl |
| PCR Reaktionspuffer
Mit 2mM MgCl2* |
1X | 5 µl |
| Vorwärts primer | 10pM | 1µL |
| Umgekehrter Primer | 10pM | 1 µl |
| Überstand | 3 µl | |
| Wasser | 3 µl | |
| Insgesamt | ——— | 25 µl |
Das Verfahren der Kolonie-PCR:
Nun, die Kolonie-PCR benötigt keine extrahierte DNA.
Wir extrahieren hier keine DNA. Stattdessen werden mehrere andere Methoden verwendet, um die Empfindlichkeit der Reaktion zu erhöhen.
Ok, warum extrahieren wir keine DNA für die Plasmid-DNA?
Weil der Grund einfach ist, ist die Zellmembran der Bakterienzelle sehr glatt.
Wir hatten bereits über die Zellmembran der Bakterienzelle gesprochen. Lesen Sie es hier: Verschiedene Arten von DNA-Extraktionsmethoden
Ein Bakterium enthält eine Weichzellmembran, die leicht durch Erhitzen oder Zentrifugieren mit hoher Geschwindigkeit lysiert werden kann.
Außerdem brauchen wir keine bakterielle DNA. Das zirkulierende zirkuläre Plasmid ist im Zytoplasma der Bakterien vorhanden, daher sind auch keine zusätzlichen Reinigungsschritte erforderlich. Durch das Aufbrechen der Zellmembran ist unsere Template-DNA für die Amplifikation bereit.
Ok, lassen Sie uns schnell durch die Methode gehen, um gute Plasmid-DNA zu erhalten.
Mit Hilfe des Sterilpickers mehrere Bakterienkolonien aufnehmen und in das Eppendorf-Röhrchen überführen.
Fügen Sie nun TE-Puffer hinzu und mischen Sie es gut. Sie können auch D / W verwenden.
Die Probe 20 Minuten im kochenden Wasserbad erhitzen.
Vertex es vorsichtig.
Zentrifugieren Sie die Probe 2 Minuten lang bei hoher Geschwindigkeit.
Übertragen Sie den Überstand in ein anderes Röhrchen und verwenden Sie ihn als Template-DNA.
Eine 20µL Probe wird in die Reaktion gegeben.
Weitere Informationen:
Warum Überstand und nicht Pellet?
DNA ist ein Biomolekül des Lebens. Die Plasmid-DNA ist sogar kleiner als die bakterielle Kern-DNA. Es enthält nur mehrere Gene von bis zu 1000bp bis 20.000bp.
Wenn wir es nur zentrifugieren, kommt die leichtere Plasmid-DNA aus der Zelle und setzt sich im Überstand ab, während das Pellet Proteine und Kern-DNA enthält, so dass wir es nicht verwenden.
Nun zur Sache.
Unser Plasmid ist zur Amplifikation bereit.
In einer anderen methode,
Verwenden die bakterielle kolonie direkt.
Diese Methode ist eine Kombination aus Hotstart-PCR und Kolonie-PCR.
Die Bakterienkolonien werden gepflückt und in das PCR-Reaktionsröhrchen gegeben.
Die Röhrchen werden in die PCR-Maschine eingesetzt. Ein zusätzlicher Heizschritt wird hinzugefügt.
Durch Erhitzen von 5 bis 7 Minuten kommt die Plasmid-DNA aus der Zelle heraus.
Nun amplifizieren die insert-spezifischen Primer die von uns insertierte DNA. Und die flankierenden Primer amplifizieren den Rest der DNA.
Die Verstärkung erfolgt für 20 bis 25 Zyklen. Die Zyklusbedingungen für die Kolonie-PCR sind unten aufgeführt,
| PCR-Schritte | Anfängliche Denaturierung | Denaturierung | Glühen | Verlängerung | Endgültige Verlängerung |
| Temperatur | 95 C | 95 C | 55-65 C | 72 C | 72 C |
| Zeit | 3 Minuten | 10 Sekunden | 45 Sekunden | 50 Sekunden | 5 Minuten |
| ——- | ——- | 25 zyklen | —– | ——- |
Lesen Sie den interessanten Artikel zur konventionellen PCR: Eine vollständige Anleitung zur Polymerase-Kettenreaktion
Tipps zur Verbesserung:
Verwenden Sie nur wenige Kolonien, da viele Kolonien die Wahrscheinlichkeit der unspezifischen Bindungen erhöhen.
Positive Kontrolle und negative Kontrolle verwenden.
Als Positivkontrolle wurde der flankierende Primer verwendet, auch wenn das Insert nicht vorhanden ist, die PCR-Reaktion ergibt eine DNA-Bande der Plasmid-DNA, die anzeigt, dass die von uns hergestellte Reaktion korrekt ist.
Verwenden Sie als Negativkontrolle das untransformierte Plasmid (Plasmid ohne Insert-DNA), diese Plasmid-DNA wird nur amplifiziert, wenn das Insert vorhanden ist.
Als Insert verwenden Sie kurze DNA-Sequenzen, längere DNA-Sequenzen erhöhen die Wahrscheinlichkeit von unspezifischen Bindungen und PCR-Reaktionsversagen.
Verwenden Sie außerdem kürzere PCR-Programme.
Die Hauptanwendung der Kolonie-PCR liegt in der Identifizierung der korrekten Ligation und Insertion von Insert-DNA in Bakterien sowie Hefeplasmid.
Nach Beendigung der Kolonie-PCR-Reaktion werden die PCR-Produkte auf dem 2%igen Agarosegel laufen gelassen. Die Ergebnisse des Experiments sind in der folgenden Abbildung dargestellt,
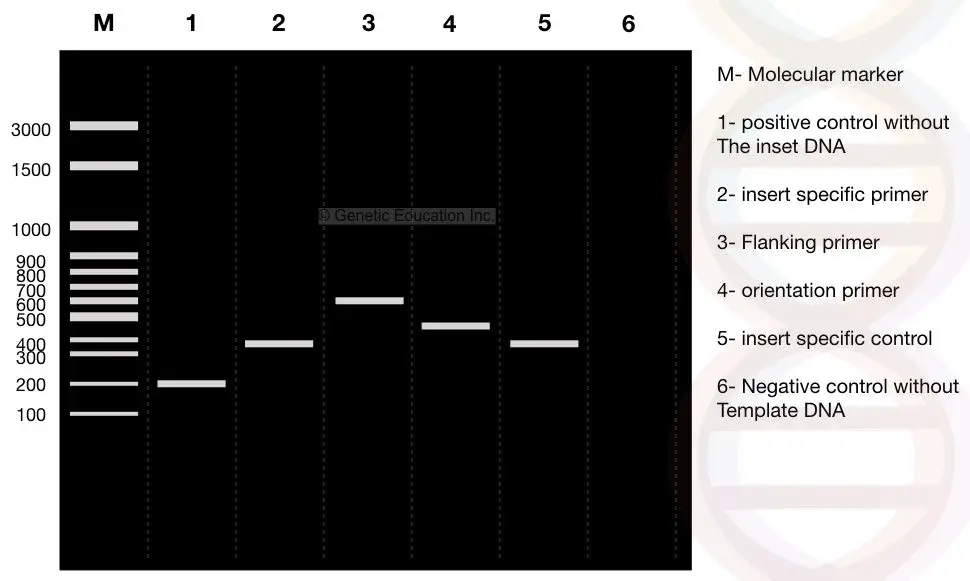
Jetzt sorgfältig beobachten die ergebnisse, die M ist die 3000bp molekulare DNA marker. Angenommen, die DNA unseres Interesses, “Insert”, ist ein 400bp-Fragment, das in das Plasmid eingefügt wird.
Siehe Spur 2: das 400 bp-Fragment unseres Inserts.
Wir haben flankierende Primer entworfen, die 100bp von beiden Seiten des Einsatzes entfernt sind. Wenn der flankierende Primer die DNA zusammen mit dem Insert amplifiziert, beträgt das Produkt 600 bp, siehe Spur 3 (400 bp der Insert-DNA + 200 bp flankierende Region).
Nun, siehe Spur 1, handelt es sich um eine Positivkontrolle ohne Insert oder um ein normales Plasmid ohne transformierte DNA. Daher amplifizieren die flankierenden Primer nur 200 bp DNA.
Siehe Spur 1, 200bp DNA-Fragment ohne Insert (Positivkontrolle).
Beachten Sie nun Spur 4. Spur 4 ist das Ergebnis der orientierungsspezifischen Primer. Der orientierungsspezifische Primer ist eine Kombination aus insertspezifischem Primer und flankierbereichsspezifischem Primer.
Zur orientierungsspezifischen Primeramplifikation werden ein Primer aus Insert-DNA und ein Primer aus dem flankierenden regionsspezifischen Primer ausgewählt.
Daher werden 100 bp Fragment aus dem flankierenden Regionsprimer und 400bp aus der Insert-DNA amplifiziert und 500 bp Fragment DNA in Spur 4 beobachtet.
Spur 5 ist die Insert-spezifische Kontrolle, die 400 bp DNA-Fragment ergibt.
Spur 6 ist die Negativkontrolle ohne Vorlage. Durch die Verwendung einer Negativkontrolle kann jede Kontamination identifiziert werden. Das Reaktionsröhrchen enthält alle Bestandteile außer der Vorlage. Im Idealfall sind also keine DNA-Banden in dieser Spur vorhanden.
Wenn eine DNA-Bande beobachtet wird, ist die Probe kontaminiert.
Vorteile der Kolonie-PCR:
- Die Technik ist schnell und kostengünstig.
- Ferner sind die Genauigkeit und Spezifität der Technik höher.
- Der Aufbau ist ebenso einfach wie die konventionelle PCR, DNA-Extraktion und Plasmid-Aufreinigung wie mühsame Schritte entfallen.
- Kein Restriktionsverdau zur Identifizierung der Insert-DNA erforderlich.
- Das gesamte Experiment kann innerhalb von 90 Minuten abgeschlossen werden.
Nachteile der Kolonie-PCR:
- Das Verfahren ist kostengünstig, schnell und zuverlässig, jedoch kann keine Mutation im Insert nachgewiesen werden.
- Darüber hinaus kann die Sequenzinformation nicht durch die Kolonie-PCR erhalten werden. wir müssen sequenzieren, um die DNA-Transformation zu bestätigen.
- Die Wahrscheinlichkeit falsch positiver Ergebnisse ist hoch.
Lesen Sie mehr;
- Was ist eine Multiplex-PCR?
Nach Abschluss des Experiments wird die Probe zur Sequenzierung geschickt, wo die DNA-Sequenz unseres Interesses bestimmt werden kann.
Wir können sogar Multiplex-PCR durchführen, indem wir sowohl insert-spezifische Primer als auch Plasmid-spezifische Primer kombinieren.
Schlussfolgerung:
Obwohl die Kolonie-PCR die beste Wahl für die Identifizierung des Gentransfers ist, reicht die einzige Kolonie-PCR-Technik nicht aus, um die Ergebnisse zu interpretieren. Es ist möglich, dass einige der Mutationen im Insert vorhanden sind, die von der PCR nicht nachgewiesen werden können.
Zur Bestätigung der Ergebnisse ist eine DNA-Sequenzierung erforderlich. Nach der Bestimmung der Sequenzreihenfolge können wir sagen, ob unser interessierendes Gen korrekt eingefügt ist oder nicht.