Zastosowanie testów uzupełniania białek-fragmentów w biologii komórki
- wprowadzenie
- zasada
- ograniczenia
- standardowe kontrole w badaniu PCA
- zastosowanie PCA w projektowaniu białek: Library vs. Library Screening for Optimally Interacting Proteins
- zastosowanie PCA do badań przesiewowych Biblioteki cDNA w komórkach ssaków
- wykorzystanie PCA jako linijki molekularnej: badania receptorów
- mapowanie sieci biochemicznych
- wniosek
- podziękowania
wprowadzenie
ścieżki biochemiczne są tak naprawdę systemami dynamicznego montażu i demontażu kompleksów białkowych, a zatem wiele współczesnych badań biologicznych zajmuje się tym, jak, kiedy i gdzie białka wchodzą w interakcje z innymi białkami uczestniczącymi w procesach biochemicznych. Zapotrzebowanie na proste podejścia do badania interakcji białko-białko, szczególnie na dużą skalę, wzrosło ostatnio wraz z postępem w projektach genomu, ponieważ kojarzenie nieznanych ze znanymi produktami genowymi zapewnia jeden kluczowy sposób ustalenia funkcji genu. Właśnie z myślą o tym wyzwaniu w naszym laboratorium opracowano testy dopełniania fragmentów białek (PCAs). W tej strategii dwa interesujące białka (białka a i B) łączą się z komplementarnymi fragmentami białka reporterowego (enzymu, białka fluorescencyjnego itp.). Jeśli białka A i B oddziałują ze sobą, fragmenty reportera są łączone, składane w natywną strukturę reportera i odtwarzają jego aktywność (Fig. 1). Białka reporterowe PCA zostały wybrane jako te wytwarzające wiele wykrywalnych aktywności, w tym sygnały fluorescencyjne, luminescencyjne i kolorymetryczne, a także proste testy selekcji przetrwania (1-14). Wykazaliśmy, że strategia PCA ma następujące możliwości: (i) umożliwia wykrywanie interakcji białko-białko in vivo i in vitro w dowolnym typie komórki; (ii) umożliwia wykrywanie interakcji białko-białko w odpowiednich przedziałach subkomórkowych lub organelach; (iii) umożliwia wykrywanie interakcji, które są szczególnie indukowane w odpowiedzi na sygnały rozwojowe, żywieniowe, środowiskowe lub hormonalne; (iv) umożliwia monitorowanie kinetycznych i równowagowych aspektów montażu białka w komórkach; oraz (v) umożliwia badanie przesiewowe pod kątem nowych interakcji białko-białko w dowolnym typie komórki (2,3,6,9) (15-19).
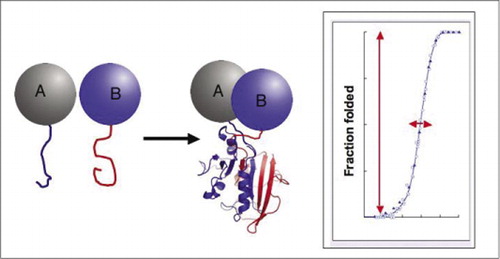
jeśli oba białka wchodzą w interakcję, fragmenty reporterowe są połączone, składają się w natywną strukturę białka reporterowego, a jego aktywność jest odtwarzana (po lewej). Te protein-fragment complementation assays (PCA) mają cechy fizyczne, które czynią je szczególnie przydatnymi jako reporterów dynamicznych kompleksów białkowych. Po prawej stronie znajduje się krzywa fałdowania białek, gdzie oś x jest pewnym parametrem zmiennym (np., stężenie jednego fragmentu względem drugiego). Wysoka kooperatywność tego procesu (niezwykle gwałtowny wzrost ułamków złożonych gatunków w bardzo wąskim zakresie) oznacza, że testy mają ogromny zakres dynamiczny, co sprawia, że wykrywanie kompleksu jest wirtualnym zjawiskiem “wszystko albo nic”. Kontrastuje to z metodami takimi jak fluorescencyjny rezonansowy transfer energii (FRET), który ma bardzo niski zakres dynamiczny i wymaga starannej optymalizacji wielu parametrów. Natomiast pomiar tworzenia kompleksu białkowego przez PCA nie jest trudniejszy niż pomiar aktywności nienaruszonego enzymu reporterowego.
zasada
zademonstrowaliśmy zasadę PCA, zaczynając od enzymu reduktazy dihydrofolianowej (DHFR) jako reportera (1). Było oczywiste, że jeśli składanie się enzymu z jego fragmentów (wykryte przez odtworzenie aktywności) było całkowicie zależne od wiązania ze sobą oddziałujących ze sobą białek, to opisany układ jest w rzeczywistości detektorem oddziaływań. My i inni od tego czasu wykazaliśmy, że zasada ta może być uogólniona na wiele enzymów, w tym Lucyferazy Gaussia i Renilla, tem β-laktamazy, a także zielone białko fluorescencyjne (GFP) i jego warianty (1-14). Kluczową cechą fragmentów PCA jest to, że są one zaprojektowane tak, aby nie składały się samoistnie, nie będąc zbliżonymi do siebie przez interakcję białek, z którymi są skondensowane (1,20). Jeśli wystąpi spontaniczne składanie, PCA po prostu nie zadziała. Spontaniczne składanie prowadziłoby do fałszywego sygnału dodatniego, sytuacji, która beznadziejnie zakłócałaby interpretację ekranów bibliotecznych in vivo (przewidywano, że będzie to ważne zastosowanie). W przeciwieństwie do PCA istnieją systemy oznaczania oparte na β-galaktozydazie i rozszczepionych inteinach, które przypominają PCA, ale różnią się koncepcyjnie i praktycznie (21,22). W obu przypadkach dobrze znane naturalnie występujące i samoistnie kojarzące się podjednostki enzymów są łączone z oddziaływującymi białkami. Głównym problemem jest to, że podjednostki, nawet jeśli są słabo powiązane, zawsze są w stanie to zrobić w pewnym stopniu, co oznacza, że istnieje stałe tło spontanicznego montażu.
ograniczenia
strategia PCA jest ogólna, w tym sensie, że nie ogranicza się do pojedynczego reportera enzymu i została opracowana w kilku różnych formach, z których każda najlepiej nadaje się do rozwiązania konkretnego pytania. Na przykład, proste PCAs z selekcją przetrwania, takie jak te oparte na DHFR, są najbardziej przydatne do wyboru biblioteki, podczas gdy PCAS z odczytem luminescencji lub fluorescencji są najlepsze do badań dynamiki przestrzennej i czasowej kompleksów białkowych. Ponieważ białka fuzyjne mogą być wyrażone w komórkach, które są istotnymi modelami do badania określonego szlaku biochemicznego, są one prawdopodobnie w ich rodzimym stanie biologicznym, w tym prawidłowych modyfikacji posttranslacyjnych (oczywiście same fragmenty PCA nie mogą zakłócać celowania lub modyfikacji białek i to musi być testowane).
wśród najprostszych i dlatego najbardziej popularnych PCAs są te oparte na białkach fluorescencyjnych (takich jak GFP i warianty), ponieważ sygnał jest dostarczany przez wewnętrzny fluorofor(7-9) (14,15,17,23). Jednak fluorescencyjne proteiny muszą wyrażać przy wysokim poziomie zapewniać że sygnał jest powyżej tła komórkowa fluorescencja, i fluorescencyjne proteiny PCAs demonstrowali być nieodwracalni, który może być użyteczny (pułapkowanie i wizualizujący rzadcy kompleksy)ale może także prowadzić mylna interpretacja obroty lub lokalizacja interakci proteiny (8,23,24). Z drugiej strony wykazano, że PCA oparte na DHFR i β-laktamazie jako reporterach, oparte na pośrednich dowodach, są odwracalne po zakłóceniu interakcji, podczas gdy PCA oparte na Lucyferazie Gaussia jest bezpośrednio odwracalne (2,3,6). Odwracalność PCA pozwala zatem na wykrycie kinetycznych i równowagowych aspektów montażu i demontażu kompleksu białkowego w żywych komórkach (fig.2).
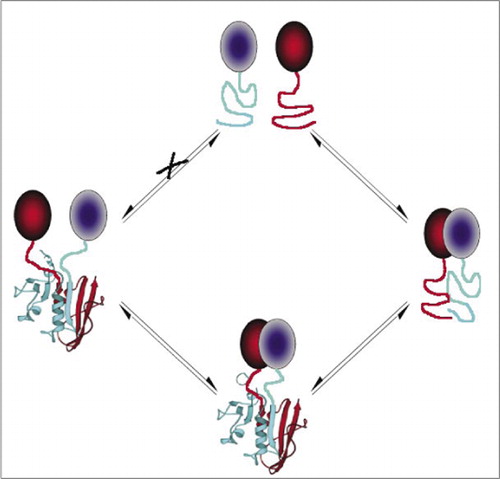
zapobiega to samoistnemu połączeniu się fragmentów (ścieżki X), które mogą prowadzić do fałszywego sygnału. Podobnie wybiera się fragmenty, dla których spontaniczne rozwijanie się fragmentów powinno nastąpić, gdy kompleks białkowy zostanie zakłócony (lewa strona).
standardowe kontrole w badaniu PCA
strategia PCA wymaga, aby fragmenty białka reporterowego gromadziły się i składały po tym, jak białka będące przedmiotem zainteresowania utworzyły kompleks. Montaż i prawidłowe składanie reportera zależy od odzyskania zarówno geometrii strukturalnej wewnętrznej białek reporterowych, jak i kompleksu utworzonego przez oddziałujące białka. Jest to jedna z głównych różnic w testach PCA w porównaniu z transferem energii rezonansu fluorescencyjnego (FRET) lub transferem energii rezonansu bioluminescencyjnego (BRET) lub testami dwóch hybryd drożdży, a ta cecha pozwoliła nam przeprowadzić badanie oparte na strukturze receptora erytropoetyny (19). Zazwyczaj wstawiamy 10-aminokwasowy elastyczny łącznik polipeptydowy składający się z (Gly.Gly.Gly.Gly.Ser) 2 pomiędzy interesującym białkiem a fragmentem reportera PCA (dla obu fuzji). Łącznik ten został wybrany, ponieważ jest najbardziej elastyczny z możliwych i empirycznie zaobserwowaliśmy, że łączniki o tej długości są wystarczająco długie, aby umożliwić odnajdywanie się fragmentów i składanie się, niezależnie od wielkości oddziałujących białek, z którymi fragmenty są skondensowane (16).
aby zapewnić, że niespecyficzne odpowiedzi nie wystąpią, należy przeprowadzić zestaw kontroli. Kontrole te mogą obejmować następujące, chociaż pierwsza jest najważniejsza: (i) niewspółpracujące białka. Odpowiedź PCA nie powinna być obserwowana, jeśli jako partnerzy PCA stosowane są białka nie oddziałujące ze sobą; nadekspresja samego nie oddziałującego białka nie powinna również konkurować o znaną interakcję. (ii) mutacje interfejsu białka partnerskiego. Mutacja punktu lub delecji partnera, o której wiadomo, że zakłóca interakcję, powinna również zapobiegać odpowiedzi PCA. iii) Konkurs. Odpowiedź PCA powinna być zmniejszona przez równoczesną nadekspresję jednego lub drugiego z oddziałujących białek, które nie są połączone z komplementarnym fragmentem PCA. (iv) Zamiana fragmentów. Obserwowana interakcja między dwoma białkami powinna wystąpić, nawet jeśli białka są zamieniane z odpowiednimi fragmentami reporterowymi.
zastosowanie PCA w projektowaniu białek: Library vs. Library Screening for Optimally Interacting Proteins
wśród pierwszych zastosowań PCA był problem projektowania białek. Test DHFR PCA został użyty w Escherichia coli do przesiewania dwóch bibliotek uzupełniająco zaprojektowanych sekwencji tworzących zamek błyskawiczny leucyny ze 1010 potencjalnymi oddziałującymi parami, z których mogliśmy praktycznie pokryć 106. Zademonstrowaliśmy, że ekran PCA został wybrany zarówno pod kątem optymalnej specyficzności wiązania, jak i rozpuszczalności i ekspresji interakcji zamków błyskawicznych (18,25). Najważniejszą cechą tego podejścia jest to, że możliwe było jednoczesne wyświetlanie dwóch bibliotek względem siebie, co nie jest łatwe do osiągnięcia przy porównywalnych dwu-hybrydowych ekranach drożdży. Prostota tego podejścia i specyfika uzyskanych informacji na temat strategii projektowania sugerują szeroką użyteczność DHFR PCA w projektowaniu białek i ukierunkowanych eksperymentach ewolucyjnych. Pokazuje również, że PCA uzupełnia strategie wyświetlania fagów, ponieważ cała selekcja, optymalizacja i testy surowości są wykonywane in vivo, dzięki czemu takie podejście jest łatwe do wykonania.
zastosowanie PCA do badań przesiewowych Biblioteki cDNA w komórkach ssaków
pierwszym krokiem w definiowaniu funkcji nowego produktu genowego jest określenie jego interakcji z innymi produktami genowymi. Jednak czysto proteinowe podejście przesiewowe oparte na interakcjach (takie jak drożdże dwu-hybrydowe) jest ograniczone, ponieważ mówi tylko, że dwa białka współdziałają, nie dostarczając żadnych innych informacji, które mogłyby powiązać białko z jego funkcją. Dlatego wykazaliśmy, że PCA może być stosowany w strategii przesiewania biblioteki cDNA, która łączy prosty ekran interakcji białek oparty na komórkach ze specyficznymi testami funkcjonalnymi, które zapewniają wstępną walidację biologicznego znaczenia interakcji (9). Pierwszy krok polega na Przesiewaniu fizycznych interakcji między przynętą a biblioteką białek ofiar kodowanych cDNA, poprzez monitorowanie odtwarzania reportera PCA w nienaruszonych żywych komórkach. Ważną cechą tego pierwszego etapu jest to, że interakcje mogą być wykrywane bezpośrednio i między pełnowymiarowymi białkami w komórkach, w których białko przynęty normalnie funkcjonuje, zapewniając w ten sposób, że mogą wystąpić niezbędne subkomórkowe ukierunkowanie, modyfikacje posttranslacyjne i interakcje z innymi białkami. Oczywiście, dla ważności eksperymentalnej, fragmenty PCA muszą być wykazane, aby nie kolidować z celowaniem lub modyfikacją białek. W drugim etapie interakcję z białkami można funkcjonalnie zwalidować w następujący sposób: po pierwsze, interakcja białkowa, wykrywana przez PCA, musi być zaburzona przez czynniki, takie jak hormony lub specyficzne inhibitory, które są znane z modulowania specyficznego szlaku biochemicznego, w którym uczestniczą białka. Wykazaliśmy to dla DHFR PCA i wykorzystaliśmy tę właściwość do mapowania szlaków sygnałowych w żywych komórkach ssaków (16). Po drugie, subkomórkowa lokalizacja interakcji białkowej, ponownie wykryta przez PCA, może być zmieniona przez środki, które modulują szlak. Tak więc strategia przesiewowa oparta na PCA łączy prosty etap przesiewowy z bezpośrednimi testami funkcjonalnymi. My i inni zastosowaliśmy tę strategię do identyfikacji nowych substratów lub regulatorów kinazy białkowej serynowo-treoninowej, PKB/Akt (9,15,26,27).
wykorzystanie PCA jako linijki molekularnej: badania receptorów
szczególną cechą strategii PCA jest to, że jeśli znamy trójwymiarową strukturę enzymu reporterowego, możliwe jest dokładne przewidzenie, jak blisko siebie muszą znajdować się fragmenty, aby zapewnić, że enzym będzie się prawidłowo zwijał i będzie miał mierzalną aktywność. Fakt ten wykorzystano do przetestowania strukturalnego modelu allosterycznego aktywacji dimerycznego receptora erytropoetyny (EpoR) przy użyciu DHFR PCA, a podejście to można rozszerzyć na badanie przejść allosterycznych w interfejsach dimerycznych lub multimerycznych białek (19). W przypadku EpoR wykazano, że domeny transmembrane dimerów receptora są oddzielone o 73 Å, jak zaobserwowano w strukturze krystalicznej niezaangażowanego EpoR. Uzasadniono, że jeśli ten nieaktywny stan istniał na błonie żywej komórki, wówczas fragmenty DHFR zespolone z końcem C domen transbłonowych składałyby się tylko wtedy, gdy ligand wywołał zmianę konformacji, która pozwala fragmentom zbliżyć się wystarczająco blisko siebie, aby zapewnić, że precyzyjna trójwymiarowa struktura DHFR może zostać utworzona (19,28). Wymagałoby to, aby końcówka N fragmentów była oddalona o 8 Å. Wstawienie elastycznych peptydów łącznikowych między domeną transbłonową a fragmentami DHFR pozwoliło nam zbadać odległość między punktami wstawiania dimeru domeny zewnątrzkomórkowej i potwierdzić, że łączniki wystarczająco długie, aby rozciągnąć się na 73 Å były potrzebne do złożenia DHFR z jego fragmentów.
mapowanie sieci biochemicznych
komórkowe maszyny biochemiczne do metabolizmu, kaskad sygnałowych i cyklu komórkowego są przykładami dynamicznego montażu i demontażu kompleksów Makromolekularnych. Są one definiowane przez grupowanie oddziałujących białek zgodnie z ich podobnymi reakcjami na zestaw perturbacji (hormony, metabolity, inhibitory enzymów itp.). Interakcje białko-białko mogą być użyte do połączenia białka o nieznanej funkcji z białkami, o których wiadomo, że biorą udział w znanym procesie biochemicznym. Wykazaliśmy, że profilowanie farmakologiczne (monitorowanie wpływu leków specyficznych dla szlaku i hormonów białkowych na interakcje białko-białko) i określanie komórkowej lokalizacji interakcji białko-białko można osiągnąć za pomocą PCAs (9)(15-17)(26). Analiza tych wyników pozwala na przedstawienie tego, jak sieci biochemiczne ewoluują w czasie i przestrzeni oraz w odpowiedzi na określone bodźce. Jako dowód Zasady, zgłosiliśmy zastosowanie tej strategii do mapowania szlaku transdukcji sygnału pośredniczonego przez receptorowe kinazy tyrozynowe (RTKs) (16). Profile farmakologiczne i komórkowa lokalizacja interakcji, które obserwowaliśmy, pozwoliły nam umieścić każdy produkt genowy w odpowiednim punkcie szlaków (ryc. 3). Z wyników naszej analizy wyłoniła się Mapa organizacji sieci RTK, która była spójna z istniejącymi modelami, ale obejmowała również kilka nowatorskich interakcji. Zdolność do monitorowania sieci interakcji białkowych w żywych komórkach zawierających wszystkie składniki badanego szlaku ujawniła ukryte połączenia, nie obserwowane wcześniej, pomimo intensywnej kontroli tej sieci. Przedstawione wyniki pokazują, że strategia PCA posiada cechy niezbędne do ogólnej walidacji funkcji genów i strategii mapowania szlaku. Niedawne zastosowanie większego zestawu PCAs umożliwiło opracowanie ogólnego podejścia w celu powiązania działań leków na określonych szlakach sygnałowych i wykrycia nieprzewidzianych działań leków (17).
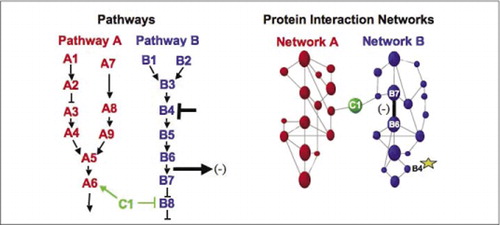
(po lewej) działanie hamującego środka drażniącego, który działa na białko B4 (T-bar), jest wykrywane w dół przez zmianę interakcji białek B6 i B7 ze sobą (arrow). W tym przypadku efektem perturbacji jest zmniejszenie liczby oddziałujących białek ( – ) wykrytych przez reportera tej interakcji (na przykład sygnał wyjściowy interakcji wykryty przez PCA sentinel). Jednak efekt może być równie pozytywny, w zależności od konsekwencji hamowania białka wyjściowego. (Po prawej) w ramach sieci interakcji białek dla szlaku B, perturbacja białka B4 (star) w jakiś sposób propaguje się przez sieć, aby w jakiś sposób wpłynąć na połączenie (szeroki pasek) między białkami B6 i B7. Nie oznacza to, że białko B4 fizycznie wchodzi w interakcję z B6 lub B7; propagacja oddziaływania poprzez sieć interakcji z białkami może być spowodowana bezpośrednimi powiązaniami fizycznymi lub procesami enzymatycznymi, które nie są oczywiste w sieci.
wniosek
rozwój i stosowanie PCA są nadal w toku. Na przykład, oprócz ograniczonych, choć pouczających, zestawów zastosowań opisanych tutaj, strategia jest stosowana do badań przesiewowych całych genomów na dużą skalę. Bardziej wyrafinowane problemy projektowania białek i fałdowania białek są badane, w tym badania czynników, które kontrolują wybór sekwencji dla optymalnych interakcji między białkami, białkami i kwasami nukleinowymi oraz białkami i małymi cząsteczkami organicznymi. PCA jest bardzo ogólnym i elastycznym podejściem eksperymentalnym, dlatego powinniśmy spodziewać się rosnącej liczby nowych zastosowań tego podstawowego narzędzia do biologii molekularnej i komórkowej w najbliższej przyszłości.
podziękowania
Stephen Michnick prowadzi Canada Research Chair in Integrative Genomics. Badania cytowane z naszego laboratorium zostały sfinansowane przez Canadian Institutes of Health Research.
- 1. Pelletier, J. N., F. X. Campbell-Valois, and S. W. Michnick. 1998. Domena oligomeryzacji-ukierunkowany ponowny montaż aktywnej reduktazy dihydrofolianowej z racjonalnie zaprojektowanych fragmentów. Proc. Natl. Acad. Sci. USA 95:12141-12146.Crossref, Medline, CAS, Google Scholar
- 2. Remy, I. I S. W. Michnick. 1999. Selekcja klonalna i ilościowe oznaczanie in vivo interakcji białek z testami uzupełniania białek-fragmentów. Proc. Natl. Acad. Sci. USA 96:5394-5399.Crossref, Medline, CAS, Google Scholar
- 3. Galarneau, A., M. Primeau, L. E. Trudeau, and S. W. Michnick. 2002. Testy uzupełniania fragmentów białka Beta-laktamazy jako czujniki interakcji białek in vivo i in vitro. Nat. Biotechnol. 20:619–622.Crossref, Medline, CAS, Google Scholar
- 4. Wehrman, T., B. Kleaveland, J. H. Her, R. F. Balint, and H. M. Blau. 2002. Interakcje białko-białko monitorowane w komórkach ssaków poprzez uzupełnianie fragmentów enzymu beta-laktamazy. Proc. Natl. Acad. Sci. USA 99:3469-3474.Crossref, Medline, CAS, Google Scholar
- 5. Spotts, J. M., R. E. Dolmetsch, and M. E. Greenberg. 2002. Obrazowanie poklatkowe dynamicznej zależnej od fosforylacji interakcji białko-białko w komórkach ssaków. Proc. Natl. Acad. Sci. USA 99:15142-15147.Crossref, Medline, CAS, Google Scholar
- 6. Remy, I. I S. W. Michnick. 2006. Bardzo wrażliwy test interakcji białko-białko oparty na Gaussia luciferase. Nat. Metody 3: 977-979.Crossref, Medline, CAS, Google Scholar
- 7. Ghosh, I., A. D. Hamilton, and L. Regan. 2000. Antiparallel leucine-directed protein reembly: zastosowanie do zielonego fluorescencyjnego białka. J. Am. Chem. Soc. 122:5658–5659.Crossref, CAS, Google Scholar
- 8. Hu, C. D., Y. Chinenov, and T. K. Kerppola. 2002. Wizualizacja interakcji pomiędzy białkami z rodziny bZIP i Rel w żywych komórkach za pomocą bimolekularnego uzupełniania fluorescencji. Mol. Cela 9: 789-798.Crossref, Medline, CAS, Google Scholar
- 9. Remy, I. I S. W. Michnick. 2004. Funkcjonalna strategia przesiewania biblioteki cDNA oparta na testach fluorescencyjnego uzupełniania białek w celu identyfikacji nowych składników szlaków sygnałowych. Metody 32:381-388.Crossref, Medline, CAS, Google Scholar
- 10. Remy, I., F. X. Campbell-Valois, G. Ghaddar, S. Aquin, and S. W. Michnick. 2005. Detection of protein interactions and library screening with protein-fragment complementation assays, P. 637-672. In Protein-Protein Interactions: A Molecular Cloning Manual, 2nd ed. Csh Laboratory Press, Cold Spring Harbor, NY.Google Scholar
- 11. Paulmurugan, R. and S. S. Gambhir. 2003. Monitorowanie interakcji białko-białko przy użyciu syntetycznej suplementacji renilla luciferase wspomaganej fragmentem białka. Anal. Chem. 75:1584–1589.Crossref, Medline, CAS, Google Scholar
- 12. Paulmurugan, R., Y. Umezawa, and S. S. Gambhir. 2002. Nieinwazyjne obrazowanie interakcji białko-białko u żywych osobników za pomocą reporterowych strategii uzupełniania i odtwarzania białek. Proc. Natl. Acad. Sci. USA 99: 15608-15613.Crossref, Medline, CAS, Google Scholar
- 13. Luker, K. E., M. C. Smith, G. D. Luker, S. T. Gammon, H. Piwnica-Worms, and D. Piwnica-Worms. 2004. Kinetyka regulowanych interakcji białkowo-białkowych ujawniła się za pomocą obrazowania uzupełniającego lucyferazę firefly w komórkach i żywych zwierzętach. Proc. Natl. Acad. Sci. USA 101: 12288-12293.Crossref, Medline, CAS, Google Scholar
- 14. Jach, G., M. Pesch, K. Richter, S. Frings, and J. F. Uhrig. 2006. Ulepszony mRFP1 dodaje czerwieni do bimolekularnego uzupełniania fluorescencji. Nat. Metody 3: 597-600.Crossref, Medline, CAS, Google Scholar
- 15. Remy, I., A. Montmarquette, and S. W. Michnick. 2004. PKB / Akt moduluje sygnalizację TGF-beta poprzez bezpośrednią interakcję z Smad3. Nat. Cell Biol. 6:358–365.Crossref, Medline, CAS, Google Scholar
- 16. Remy, I. I S. W. Michnick. 2001. Wizualizacja sieci biochemicznych w żywych komórkach. Proc. Natl. Acad. Sci. USA 98:7678-7683.Crossref, Medline, CAS, Google Scholar
- 17. Macdonald, M. L., J. Lamerdin, S. Owens, B. H. Keon, G. K. Bilter, Z. Shang, Z. Huang, H. Yu, et al.. 2006. Identyfikacja efektów ubocznych i ukrytych fenotypów leków w komórkach ludzkich. Nat. Chem. Biol. 2:329–337.Crossref, Medline, CAS, Google Scholar
- 18. Pelletier, J. N., K. M. Arndt, A. Pluckthun, and S. W. Michnick. 1999. Wybór in vivo biblioteka-versus-biblioteka zoptymalizowanych interakcji białko-białko. Nat. Biotechnol. 17:683–690.Crossref, Medline, CAS, Google Scholar
- 19. Remy, I., I. A. Wilson, and S. W. Michnick. 1999. Aktywacja receptora erytropoetyny poprzez zmianę konformacji indukowaną przez ligand. Nauka 283:990-993.Crossref, Medline, CAS, Google Scholar
- 20. Gegg, C. V., K. E. Bowers i C. R. Matthews. 1997. Sondowanie minimalnych niezależnych jednostek składanych w reduktazie dihydrofolianowej poprzez rozwarstwienie cząsteczkowe. Protein Sci. 6:1885–1892.Crossref, Medline, CAS, Google Scholar
- 21. Rossi, F., C. A. Charlton, and H. M. Blau. 1997. Monitorowanie interakcji białko-białko w nienaruszonych komórkach eukariotycznych poprzez uzupełnienie beta-galaktozydazy. Proc. Natl. Acad. Sci. USA 94:8405-8410.Crossref, Medline, CAS, Google Scholar
- 22. Ozawa, T., S. Nogami, M. Sato, Y. Ohya, and Y. Umezawa. 2000. Fluorescencyjny wskaźnik do wykrywania interakcji białko-białko in vivo w oparciu o łączenie białek. Anal. Chem. 72:5151–5157.Crossref, Medline, CAS, Google Scholar
- 23. Magliery, T. J., C. G. Wilson, W. Pan, D. Mishler, I. Ghosh, A. D. Hamilton, and L. Regan. 2005. Detecting protein-protein interactions with a green fluorescent protein fragment reembly trap: scope and mechanism. J. Am. Chem. Soc. 127:146–157.Crossref, Medline, CAS, Google Scholar
- 24. Nyfeler, B., S. W. Michnick, and H. P. Hauri. 2005. Wychwytywanie interakcji białkowych w drodze wydzielniczej żywych komórek. Proc. Natl. Acad. Sci. USA 102:6350-6355.Crossref, Medline, CAS, Google Scholar
- 25. Arndt, K. M., J. N. Pelletier, K. M. Muller, T. Alber, S. W. Michnick, and A. Pluckthun. 2000. Heterodimeryczna para peptydów zwojowo-zwojowych wybrana in vivo z zaprojektowanego zespołu bibliotek-kontra-bibliotek. J. Mol. Biol. 295:627–639.Crossref, Medline, CAS, Google Scholar
- 26. Remy, I. I S. W. Michnick. 2004. Regulacja apoptozy przez białko Ft1, nowy modulator kinazy białkowej B / Akt. Mol. Cell. Biol. 24:1493–1504.Crossref, Medline, CAS, Google Scholar
- 27. Ding, Z., J. Liang, Y. Lu, Q. Yu, Z. Songyang, S. Y. Lin, and G. B. Mills. 2006. Badanie przesiewowe dotyczące uzupełniania białek oparte na retrowirusie ujawnia funkcjonalnych partnerów wiążących AKT1. Proc. Natl. Acad. Sci. USA 103:15014–15019.Crossref, Medline, CAS, Google Scholar
- 28. Cody, V., J.R. Luft, E. Ciszak, T.I. Kalman, and J.H. Freisheim. 1992. Crystal structure determination at 2.3 A of recombinant human dihydrofolate reductase ternary complex with NADPH and methotrexate-gamma-tetrazole. Anticancer Drug Des. 7:483–491.Medline, CAS, Google Scholar